SPSS筛选因子之后如何采用python建模和反演整个研究区?(以反演生物量为例)
引言
在遥感数据分析中,因子筛选和建模是关键步骤。筛选出与目标变量(如生物量)显著相关的因子,不仅可以提高模型的预测精度,还能简化模型结构。
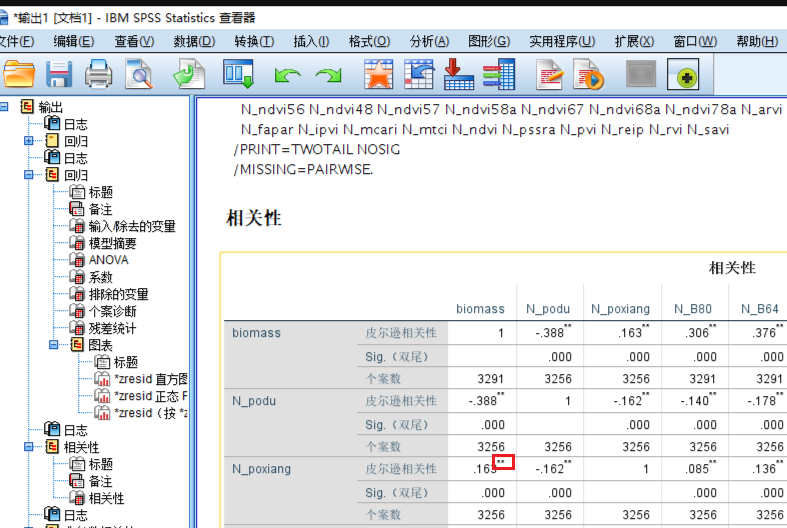
通常我们使用SPSS软件进行因子的初步筛选,通过相关性分析选出与目标变量相关性较高的因子。在SPSS中,我们可以使用皮尔逊相关系数来评估因子的显著性,通常选择带有两颗星号(表示相关性显著)的变量用于后续建模。
本教程将以反演森林生物量为例,详细介绍如何使用Python对筛选后的因子进行建模,并应用于整个研究区的反演任务。这一流程具有较强的通用性,适用于其他类似的遥感反演任务。
任务描述
本文以建模和反演森林生物量为例。我们将使用随机森林算法构建预测模型,并将其应用于整个研究区的生物量反演。
本文所用的示例数据和最终的反演结果已上传至百度网盘,整理不易,如有需要公众号(Python与遥感)后台回复 49 获取。
通过本文,读者可以完整地掌握从因子筛选到建模、再到研究区生物量反演的整个流程。

文件描述
- 随机森林反演.ipynb:Jupyter Notebook代码文件,用于实现建模和反演。
- jianmo.csv:用于建模的数据文件,包含反演变量(生物量)和5个最重要的影响因子。
- 5个.tif文件:建模和反演所用的栅格变量数据。
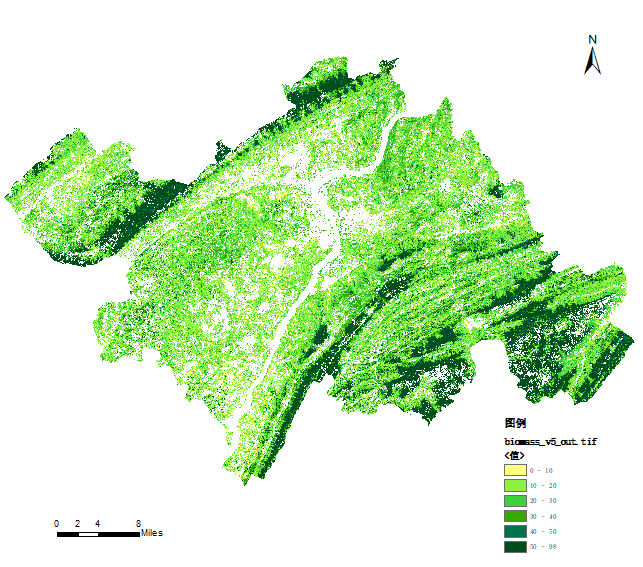
- 生物量.tif:反演得到的研究区生物量结果,并已导出为png方便查看。
完成反演后,我们将得到整个研究区的生物量分布图。下图展示了根据森林区域掩膜文件提取后的研究区生物量分布结果。

代码文件截图
如何打开代码运行,请看这篇文章



代码详解
1. 导入所需的库
首先,导入进行数据处理、建模和可视化所需的库:
import pandas as pd
import numpy as np
from matplotlib import pyplot as plt
from sklearn.ensemble import RandomForestRegressor
from sklearn.model_selection import train_test_split
from sklearn.metrics import mean_squared_error, r2_score
import scipy.stats as stats
from sklearn import metrics
from sklearn.model_selection import GridSearchCV, RandomizedSearchCV
from joblib import dump
pandas和numpy:数据处理和数值计算。matplotlib:绘图和可视化。sklearn:机器学习建模、模型评估和超参数调整。scipy:统计计算。
2. 读取数据并进行预处理
我们从CSV文件中读取数据,并将其分为自变量(影响因子)和因变量(生物量):
data = pd.read_csv(r'.\jianmo.csv', na_values=[' '])
y = data.iloc[:, 0].values # 生物量(因变量)
X = data.iloc[:, 1:].values # 影响因子(自变量)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=44)
pd.read_csv读取CSV文件,na_values=[' ']将空白值视为缺失值。y为因变量(生物量),X为自变量(影响因子)。train_test_split将数据分为训练集和测试集,测试集占比30%。
3. 构建随机森林模型并进行训练
使用随机森林算法进行建模:
regressor = RandomForestRegressor(n_estimators=500, max_features=5)
regressor.fit(X_train, y_train)
RandomForestRegressor构建随机森林回归模型,设置n_estimators=500构建500棵树,max_features=5表示每次分裂时最多考虑5个特征。fit函数训练模型。
4. 误差分析
接下来,对模型的预测误差进行分析,并计算Pearson相关系数、R²和均方根误差(RMSE):
random_forest_predict = regressor.predict(X_test)
random_forest_error = random_forest_predict - y_test
plt.figure(1)
plt.clf()
plt.hist(random_forest_error)
plt.xlabel('Prediction Error')
plt.ylabel('Count')
plt.grid(False)
plt.show()
random_forest_pearson_r = stats.pearsonr(y_test, random_forest_predict)
random_forest_R2 = metrics.r2_score(y_test, random_forest_predict)
random_forest_RMSE = metrics.mean_squared_error(y_test, random_forest_predict) ** 0.5
print('Pearson correlation coefficient = {0}、R2 = {1} and RMSE = {2}.'.format(random_forest_pearson_r[0], random_forest_R2, random_forest_RMSE))
regressor.predict对测试集进行预测,并计算误差。- 绘制误差分布图,帮助理解模型的误差情况。
- 计算Pearson相关系数、R²和RMSE,评估模型的预测性能。
5. 栅格数据的读取与处理
读取研究区的栅格变量数据,并整合为一个矩阵:
import rasterio
from tqdm import tqdm
with rasterio.open(r'.\podu.tif') as src:
data1 = src.read(1)
meta = src.meta
with rasterio.open(r'.\dem.tif') as src:
data2 = src.read(1)
with rasterio.open(r'.\lai.tif') as src:
data3 = src.read(1)
with rasterio.open(r'.\ireci.tif') as src:
data4 = src.read(1)
with rasterio.open(r'.\mcari.tif') as src:
data5 = src.read(1)
X = np.stack((data1, data2, data3, data4, data5), axis=-1)
- 使用
rasterio库读取栅格文件,将每个变量的数据存储在data1至data5中。 meta保存栅格文件的元数据,用于保存预测结果。np.stack将所有栅格数据整合为一个三维矩阵。
6. 数据清洗
对栅格数据进行清洗,去除缺失值(NaN):
X_2d = X.reshape(-1, X.shape[-1])
print(np.isnan(X_2d).any()) # 检查是否存在 NaN 值
X_2d[np.isnan(X_2d)] = 0 # 将 NaN 值替换为0
- 将三维数据转换为二维数据,便于处理。
- 检查并处理缺失值,确保数据完整性。
7. 使用模型进行预测
利用训练好的随机森林模型对栅格数据进行生物量预测:
y_pred = []
for i in tqdm(range(0, X_2d.shape[0], 10000)):
y_pred_chunk = regressor.predict(X_2d[i:i+10000])
y_pred.append(y_pred_chunk)
y_pred = np.concatenate(y_pred)
- 将栅格数据分块输入模型预测,减少内存占用。
- 使用
tqdm显示进度条,实时监控预测进度。
8. 保存预测结果
将预测得到的生物量数据保存为新的栅格文件:
with rasterio.open(r'.\生物量.tif', 'w', **meta) as dst:
dst.write(y_pred.reshape(X.shape[:-1]), 1)
print("预测结束")
- 创建新的栅格文件,并将预测结果写入其中。
- 确保预测结果与原始栅格的空间信息一致。
9. 结果展示
最后,我们得到了整个研究区的生物量反演图。这一过程不仅适用于生物量的反演,也适用于其他多光谱遥感数据的反演任务。
总结
通过这篇文章,我们展示了如何使用Python对筛选后的影响因子进行随机森林建模,并应用于遥感数据反演森林生物量的任务。该流程不仅可以提高预测精度,还能有效处理大规模的遥感数据,为生态监测和森林管理提供重要支持。
如有更多问题或需要进一步指导,欢迎公众号(Python与遥感)后台联系作者获取更多信息。