小罗碎碎念
这篇论文题为《Vision Transformers for Computational Histopathology》,综述了视觉变换器(Vision Transformers, ViTs)在计算病理学中的应用,包括图像分类、分割和生存风险回归等任务,并探讨了面临的挑战和未来发展方向。
今天这篇文章于2023年7月发表于《IEEE Reviews in Biomedical Engineerin》,目前IF=17.2,比去年降了0.4分。这期推文属于文献的作者约稿,很感谢老师能给我这个机会,也欢迎其他的老师积极推荐自己的优秀研究成果给小罗!!有问题可以在交流群内讨论!!

| 作者 | 单位 | 单位(中文) |
|---|---|---|
| Hongming Xu | School of Biomedical Engineering, Faculty of Medicine, and Key Laboratory of Integrated Circuit and Biomedical Electronic System, Liaoning Province, Dalian University of Technology, Dalian, 116024 China | 辽宁工程技术大学医学与生物工程学院集成电路与生物医学电子系统重点实验室,大连,116024中国 |
| Qi Xu | School of Artificial Intelligence, Faculty of Electronic Information and Electrical Engineering, Dalian University of Technology, Dalian, 116024 China | 大连理工大学电子信息与电气工程学院人工智能学院,大连,116024中国 |
| Jeonghyun Kang | Department of Surgery, Gangnam Severance Hospital, Yonsei University College of Medicine, Seoul, 06273 Republic of Korea | 首尔延世大学医学院外科学系,首尔,06273韩国 |
| Anant Madabhushi | Wallace H. Coulter Department of Biomedical Engineering, Georgia Institute of Technology and Emory University, Atlanta, GA, 30322 USA | 乔治亚理工学院和埃默里大学华莱士·休伊特生物医学工程系,亚特兰大,GA,30322美国 |
| Chu Han | 1. Department of Radiology, Guangdong Provincial People’s Hospital (Guangdong Academy of Medical Sciences), Southern Medical University, Guangzhou 510080, China; 2. Medical Research Institute, Guangdong Provincial People’s Hospital (Guangdong Academy of Medical Sciences), Southern Medical University, Guangzhou 510080, China; 3. Guangdong Provincial Key Laboratory of Artificial Intelligence in Medical Image Analysis and Application, Guangzhou 510080, China | 广东省人民医院(广东医学科学院)放射科,广州,510080中国;广东省人民医院(广东医学科学院)医学研究所,广州,510080中国;广东省人工智能在医学影像分析与应用重点实验室,广州,510080中国 |
| Zaiyi Liu | corresponding author | 通讯作者 |
| Anant Madabhushi | corresponding author | 通讯作者 |
| Cheng Lu | corresponding author | 通讯作者 |
-
研究背景:
- 问题:计算病理学旨在自动分析包含丰富表型信息的全幅切片图像(WSIs),以提供更准确的癌症诊断、预后和治疗建议。
- 难点:由于癌症组织异质性和标注数据稀疏性,传统的深度学习方法在处理WSIs时面临巨大挑战。
- 相关工作:现有的计算机辅助病理学图像分析方法主要依赖于传统机器学习和卷积神经网络(CNN),但这些方法在处理多源数据集和长距离依赖关系方面存在局限性。
-
研究方法:
- Vision Transformer(ViT)作为最新的深度学习技术,通过自注意力机制学习特征表示和全局依赖关系。ViT将输入图像分割成一系列图像块(tiles),并计算这些图像块之间的关系。
- 具体来说,自注意力(SA)模块通过计算查询矩阵(Q)、键矩阵(K)和值矩阵(V)之间的相似度来生成注意力权重,从而强调图像中的关键区域。多头自注意力(MSA)模块并行执行多个SA模块,以提高模型的表达能力。
- ViT模型包括一个图像块嵌入块、一个Transformer编码器和一个任务特定的解码器(如基于MLP的分类头)。图像块通过预训练的深度学习模型(如Resnet-50)进行嵌入,并考虑相对位置信息。
-
实验设计:
- 论文回顾了近年来在计算病理学中应用的ViT模型,涵盖了分类、分割和生存风险回归等任务。
- 在肿瘤病理分类方面,研究了完全监督的ViT、弱监督的ViT、自监督的ViT和半监督的ViT。例如,Ikromjanov等人使用ViT对前列腺癌WSIs进行Gleason评分分级,取得了有希望的结果。
- 在病理成分分割方面,探讨了将Transformer块插入到编码器和解码器块中,以及同时使用编码器和解码器块的方法。例如,Ji等人提出了一个多复合Transformer用于生物医学图像分割,显著提高了Dice相似系数(DSC)分数。
- 在生存风险回归方面,研究了利用ViT进行生存预测的方法。例如,Shen等人使用ViT结合卷积操作预测肺癌患者的生存风险,取得了良好的效果。
-
结果与分析:
- 实验结果表明,ViT模型在计算病理学任务中表现出色。例如,在前列腺癌Gleason评分分级任务中,ViT模型的性能与最先进的CNN模型相当,甚至在某些情况下更优。
- 在肿瘤病理分类任务中,ViT模型在各种癌症类型上均表现出良好的泛化能力。例如,在乳腺癌分类任务中,ViT模型在公共BreaKHis数据集上的性能优于其他基线CNN模型。
- 在病理成分分割任务中,ViT模型显著提高了分割精度。例如,在肺腺癌分割任务中,ViT模型在Swin Transformer-based MIL方法中表现出色,提供了高分辨率的可视化结果。
- 在生存风险回归任务中,ViT模型在多模态数据融合方面表现出色。例如,在结肠癌生存预测任务中,TransSurv模型有效整合了来自WSIs的多模态特征和基因组数据,显著提高了C-index值。
-
总体结论:
- ViT模型在计算病理学领域具有广阔的应用前景,能够提供比CNN模型更优越的性能。
- 尽管ViT模型在数据需求和硬件资源方面存在挑战,但通过改进模型结构、采用自监督学习和半监督学习等方法,可以克服这些问题。
- 未来研究方向包括开发更轻量级的ViT模型、结合图神经网络和Transformer、以及开发多模态数据融合模型,以满足临床需求。
一、引言
组织病理学图像分析是病理学家在癌症诊断和预后中的关键环节。除了提供诊断信息外,组织切片中丰富的表型信息被认为对患者的临床预后和遗传改变具有指导意义[1]。
由于在常规临床环境中存在显著的观察者变异性和手动分析的工作负担,随着数字病理切片扫描仪的普及,计算机辅助的组织病理学图像分析不断发展。早期研究主要使用经典图像处理算法检测或分割组织图像中的感兴趣区域(ROI),如细胞核和腺体[2]、[3]。
根据手动判断的诊断指南,组织图像表征通常通过纹理和颜色特征以及分割ROI的形态和空间特征来刻画。利用组织图像特征和传统机器学习模型(如支持向量机、随机森林等)进行计算机辅助的癌症诊断或分级。尽管经典特征提取和分类在可解释性方面具有优势,但它们在跨多源数据集的泛化能力上往往较弱。
随着大数据分析和数字化病理切片的产生,深度学习已成为自动组织学图像分析的主流方法,它在许多计算组织病理学任务中革新了诊断性能[1]、[5]、[6]、[7]。例如,Bejnordi等人[8]报告称,深度卷积神经网络(CNN)在识别淋巴结中乳腺癌转移的H&E染色组织切片方面可以超越专家病理学家。Tellez等人[9]表明,CNN可以有效检测H&E染色的乳腺癌全切片图像(WSIs)中的有丝分裂。
除了在诊断性能上取得更好表现外,深度学习模型在预测分子或遗传改变方面显示出卓越的能力。
特别是,Kather等人[10]报告称,深度残差学习可以直接从胃肠道癌症的H&E组织学预测微卫星不稳定(MSI)。
作者之前的一项工作[11]表明,可以使用基于ImageNet数据集的预训练Xception模型提取的WSIs的组织学特征来预测膀胱癌患者的肿瘤突变负担。尽管深度学习架构在各种组织学图像分析任务中提供了令人印象深刻的性能,但深度学习引导的WSI分析在诊断、分级和临床预后预测方面也因癌症组织的异质性和弱标注的存在而面临一些特定挑战。
WSI通常具有巨大的尺寸(通常达到100K×100K像素甚至更多),因此需要将其分解成成千上万的小图块以输入深度学习模型。数百个WSIs可以轻易产生数百万个图像图块,其中许多小图块与患者级标签(如临床结果)无关,这是由于组织学组织的异质性[12]、[13]。
由于数据量巨大,实际上对于病理学家来说,手动标注所有图像图块以训练深度学习模型是一项挑战。
为了解决这些问题,弱监督学习,尤其是多实例学习(MIL),在计算组织病理学中得到了广泛探索。
在MIL的背景下,每个WSI被视为一个“包”,每个包包含多个实例(即图像图块)。这些实例是通过裁剪高分辨率WSI生成的小尺寸图块。
然后,通过使用通常与临床信息一起提供的患者级标签来实现MIL。MIL方法帮助减轻了病理学家在组织切片上进行广泛手动标注的需求。
遵循经典的MIL假设,Campanella等人[14]训练了一个基于MIL的深度学习系统,该系统在前列腺癌和正常区分等弱标注WSI分类任务上提供了有希望的性能。Ilse等人[15]提出了一种基于注意力的深度MIL方法,该方法通过可训练的神经网络确定实例权重,并通过使用实例级特征的加权平均值来聚合包级表示。该方法在乳腺癌和结肠癌检测的组织学图像中进行了测试,提供了优越的性能。Lu等人[16]将基于注意力的MIL方法扩展到多类预测,并引入了实例级聚类以辅助学习类特定特征。在肾细胞癌亚型分类、肺癌亚型分类和淋巴结转移检测上的实验显示了有希望的性能和可视化结果。
尽管在WSI级分类上展示了令人印象深刻的结果,但这些基于MIL的方法尚未探索不同实例之间的全局、长程交互。换句话说,它们没有考虑不同图像图块之间的空间相关性和交互注意力,这可能会限制它们在WSI级分类任务上的性能。
Transformer最初用于处理自然语言处理(NLP)中的序列推理任务,它通过堆叠自注意力层在捕捉序列数据的长期依赖性方面显示了显著的能力[17]。
受Transformer在NLP领域的显著成功启发,视觉Transformer(ViT)和混合卷积-Transformer模型已引入到计算机视觉领域,旨在克服CNN中局部卷积的有限感受野[18]、[19]。
ViT通过将输入图像分解为一系列图块并基于注意力机制计算这些图块之间的关系来构建图像级表征[20]。由于在图像图块上执行的注意力操作,ViT可以更好地探究上下文信息和长程依赖性,与CNN相比。一些近期研究表明,ViT在训练所需的计算资源大幅减少的情况下,与现有的SOTA CNN相比取得了优秀的结果[21]、[22]。
鉴于ViT模型在计算机视觉中的优势和成功,近期的一些研究已将ViT应用于解决计算组织病理学中的广泛问题,如癌症亚型分类[23]、[24]、[25]、细胞核分割[26]、[27]和生存风险预测[28]、[29]。这些研究表明,ViT在计算组织病理学领域的应用前景广阔,尽管在将ViT模型应用于WSI分析时仍存在许多挑战,如较差的可解释性和高计算复杂度需要解决。
在计算组织病理学领域,已有关于深度神经网络模型和多实例学习的综述工作[1]、[30]、[31],以及关于ViT在医学图像分析中广泛应用的综述[20]、[17]。
与现有综述[1]、[30]、[31]不同,本文主要突出了ViT在计算组织病理学上的最新进展和应用,旨在专门鼓励和促进基于Transformer的组织学图像分析方法的开发。与现有ViT综述[20]、[17]相比,本文深入全面地回顾了ViT在计算组织病理学上的最新发展,并讨论了在组织病理学图像分析中应用Transformer的具体挑战和前景。
二、肿瘤病理分类
使用ViT进行癌症诊断或预后预测的方法可以表述为分类任务,其中ViT作为从WSIs到患者级诊断或预后标签的映射函数。
如图4所示,根据是否存在图块级标注和自监督学习,作者将这些分类研究分为四个主要类别:全监督ViT、弱监督ViT、自监督ViT和半监督ViT。
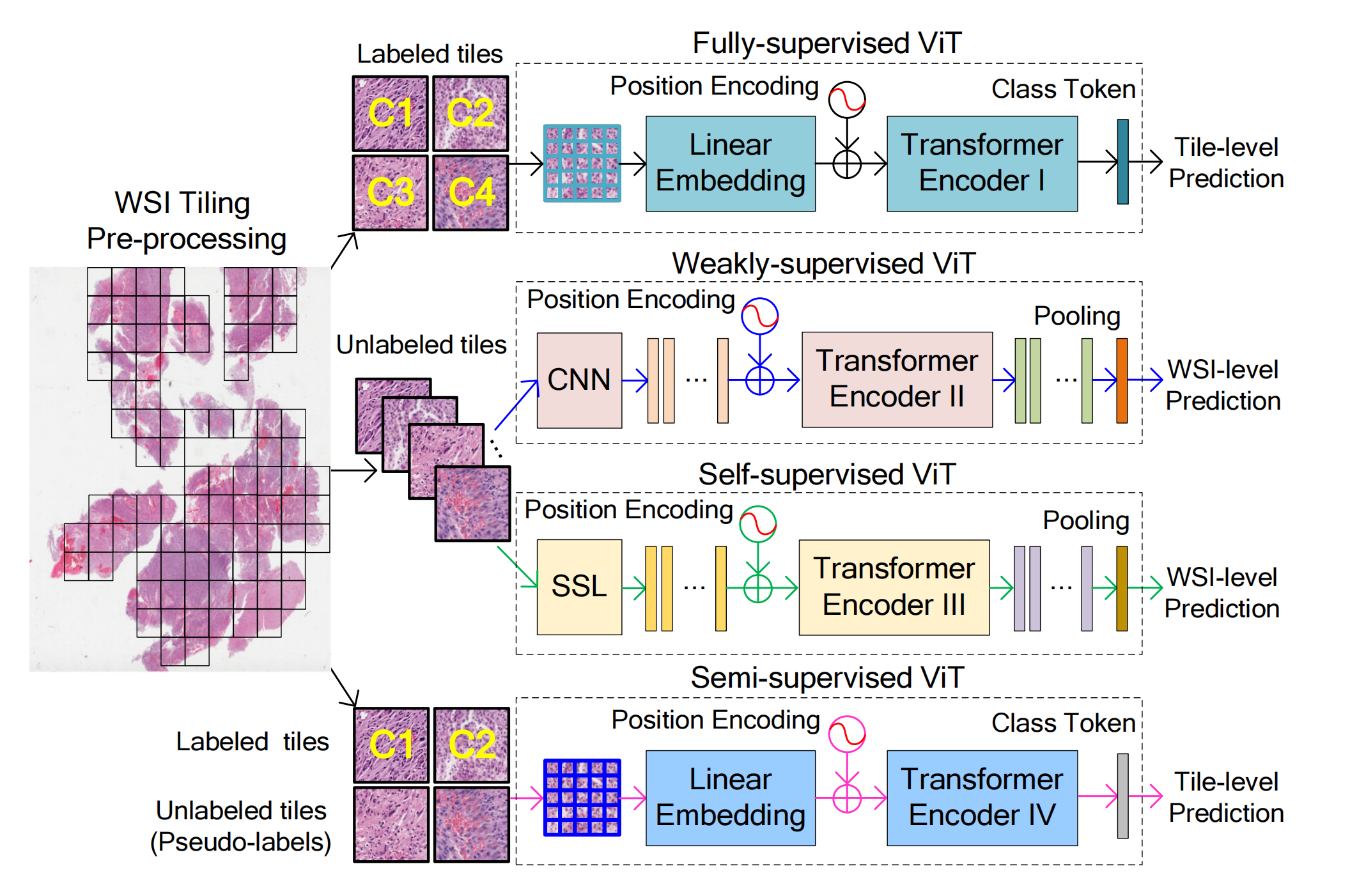
2-1:全监督ViT
本部分总结了使用全监督ViT的计算组织病理学应用,其中WSIs的组织学图块(或补丁)由病理学家手动分配真实标签。
类似于原始文献[21]中提出的ViT被视为经典Transformer,其中特征嵌入是通过扁平化图块的线性投影获得的。使用经典ViT模型,Ikromjanov等人[46]在Kaggle PANDA挑战赛[47]提供的前列腺癌WSIs的组织学图像图块(256×256像素)上探索了Gleason评分分级,并展示了有希望的结果。
Alotaibi等人[48]利用预训练的ViT和DeiT模型[40]的集成网络进行乳腺癌组织病理学图像分类,研究表明通过平均两个模型的预测可以提高分类性能。Laleh等人[13]对六种弱监督深度学习流程进行了基准测试,用于WSI分类,包括经典ViT模型、Resnet、Efficientnet和三种其他MIL模型。
在他们的实现中,假设图像图块具有相同的WSI级标签以进行全监督学习。
在预测结直肠、胃和膀胱癌的生物标志物和遗传突变等不同分类任务上的评估表明,ViT的表现与CNN相当,但从未显著优于CNN。Su等人[49]将经典ViT模块集成到图神经网络(GNN)中,用于分级结直肠癌组织学图像,其中图节点对应于基于CNN的细胞核特征,而Transformer模块用于捕获全局图节点特征。
在两个公共数据集[50]、[51]上的评估显示,相较于其他竞争性深度学习模型略有改进。Gao等人[23]提出了一种基于实例的ViT(i-ViT),用于识别乳头状肾细胞癌的亚型。i-ViT首先根据细胞核分割选择top-k肿瘤图块实例,然后基于ViT聚合肿瘤核特征和位置进行预测。
通过对TCGA-KIRP项目中的171张诊断WSIs手动选择的1162个ROI(每个ROI 2000×2000像素)进行验证,他们展示了优于基线CNN的性能。
为了同时利用CNN和ViT模型的优势,一些研究探索了双分支网络或集成模型。
Chen等人[52]提出了一种多尺度的ViT方法,用于在开源数据集(HE-GHI-DS[53])中的组织学图像上识别胃癌。他们的模型基于BoTNet-50[39]和Inception-V3[54]的双分支组成,通过融合全局和局部特征表征进行诊断预测。Tummala等人[55]通过使用Swin Transformer[41]的集成,对乳腺癌组织学图像进行多类分类,并在公共BreaKHis数据集[56]上报告了良好的性能。
Fu等人[57]开发了一个名为StoHisNet的双分支网络,基于Swin Transformer和CNN模块,该网络在分类胃癌亚型方面表现更好。Zhou等人[58]提出了一个基于Resnet和ViT的并行双分支网络(DBNet),该网络采用自适应稀疏交互块(ASIB)来捕获组织学图像中的局部和全局依赖。
他们报告了在预测脑肿瘤分级方面相较于四个SOTA模型的性能提升。Huang等人[59]提出了一种端到端的ViT-AMC网络,具有自适应模型融合和多目标优化,有效地融合了ViT和集成了注意力机制的卷积(AMC)块。ViT-AMC在喉癌分级中展示了良好的性能。
2-2:弱监督ViT在计算组织病理学中的应用
弱监督ViT方法在组织病理学图像分析中的应用,涉及仅提供患者级标签,而不涉及病理学家分配的图块级标注。这些方法的特点是在训练ViT模型时不应用自监督学习。
由于多尺度组织学特征在癌症诊断中通常被认为是有用的,因此开发了多种弱监督ViT方法来探索多尺度特征在肿瘤病理分类中的应用。
例如,张等人[60]提出了多路径跨尺度ViT(MCViT),用于从WSI中分类胸腺瘤亚型。该方法使用两个子网络分别预测图块级病理信息和WSI级胸腺瘤亚型。第一个子网络,称为跨注意力的尺度感知Transformer,由三个分支的Swin Transformer[41]、Pyramid ViT[61]和经典ViT[21]组成,用于学习多尺度图像特征。第二个子网络使用经典ViT块和多尺度图像特征来预测WSI级胸腺瘤亚型。
其他弱监督ViT方法探索了使用Transformer块进行实例级特征提取和聚合,以便在弱监督学习设置下进行WSI分类。例如,邵等人[24]提出了基于Transformer的多实例学习(TransMIL)方法,用于WSI分类。该方法使用Transformer层来聚合形态信息,并使用金字塔位置编码生成器来编码空间信息。他们评估了TransMIL在各种WSI分类任务上的表现,如乳腺癌转移检测(CAMELYON16[8])和肺癌亚型分类(TCGA-NSCLC)。从TransMIL计算出的注意力分数被可视化以展示诊断解释性。
2-3:自监督ViT在计算组织病理学中的应用
自监督学习在训练ViT模型中的应用,以应对在组织学图像上收集全面标注的挑战。
自监督ViT方法涉及在WSI上仅提供患者级标签,并且在训练ViT模型时涉及自监督学习。例如,卢等人[25]提出了稀疏注意力基于多实例对比学习(SMILE)方法,用于从WSI中进行胶质瘤亚型分类。该方法首先使用SimCLR提取图像图块的有意义表示。然后,将top-N实例嵌入馈入Transformer模块,形成包级特征表示,并使用MLP分类头将它们映射到不同的胶质瘤亚型。
其他自监督ViT方法包括使用DINO(无标签的自蒸馏)方法进行自监督学习。例如,古尔等人[73]提出了用于检测乳腺癌淋巴结转移的自监督ViT模型,该模型在性能上优于其他方法。该模型使用DINO方法学习图像图块的特征表示。然后,冻结ViT模型,并在ViT的潜在特征上训练双流MIL聚合器以进行包级预测。
2-4:半监督ViT在计算组织病理学中的应用
半监督学习方法在计算组织病理学中的应用,涉及在训练过程中使用少量标注数据和大量未标注数据。
这种方法可以有效减少标注工作的努力。例如,米罗内科等人[78]建立了一个金字塔Transformer MIL网络,用于预测前列腺癌Gleason评分。该方法使用预训练的Resnet50提取多尺度图块级特征,并使用Transformer块聚合不同级别的特征。他们基于半监督生成了实例级伪标签,以辅助优化过程。在Kaggle PANDA挑战数据集[47]上的评估显示,该方法的表现与比赛前三名获奖队伍相当。
其他半监督ViT方法包括使用自适应令牌采样技术,以增强模型的鲁棒性。例如,王等人[77]提出了一个半监督ViT模型,该模型在乳腺癌检测上优于基线CNN。
三、病理成分分割
病理成分分割的目标是从组织学图像中自动描绘感兴趣区域(ROI)或解剖结构,这是计算机辅助诊断应用程序中的一个关键模块。
本部分重点介绍了各种尝试将Transformer块集成到组织病理学图像分割模型中的尝试。考虑到U-Net[80]仍然是基于Transformer的分割模型的主要骨干架构,作者将涉及Transformer的分割研究分为三类:
- 在编码器和解码器块之间的Transformer
- 在编码器块中的Transformer
- 在编码器和解码器块中的Transformer
需要注意的是,尽管许多回顾性研究进行了各种分割任务,但本研究只报告了与组织学组织分割相关的结果。图5展示了典型的基于Transformer的组织学图像分割框架,其中Transformer块插入到分割模型的不同模块中。
Fig. 5 展示了三种基于变换器(Transformer)的组织学图像分割框架,每种框架都采用了不同的策略来整合变换器到图像分割模型中。
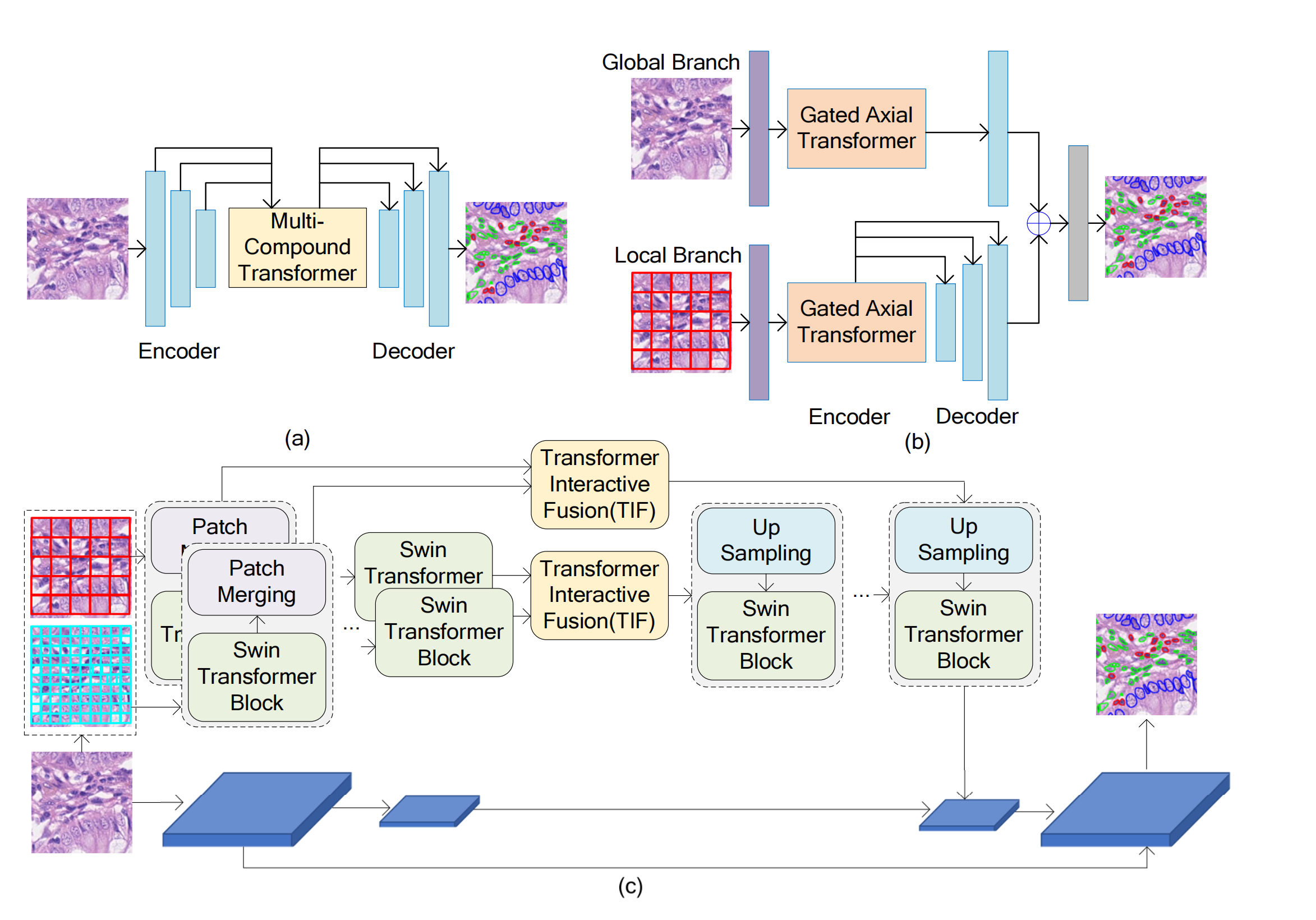
(a) 编码器和解码器块之间的变换器:这种设计尝试将丰富的特征学习和语义结构挖掘整合到一个统一的框架中。在这种框架下,变换器被放置在U-Net模型的编码器和解码器之间,目的是为了捕捉不同特征和不同标签之间的上下文信息和语义关系。这种结构有助于模型更好地理解图像的全局和局部结构,从而提高分割的准确性。
(b) 编码器块中的变换器:在这个框架中,变换器被集成到U-Net的编码器中,使用门控注意力模型来同时提取全局和局部特征。门控轴向变换器模块允许模型在不同层次上学习图像的高级信息和细节信息,这有助于改善分割性能,特别是在处理具有复杂结构的组织学图像时。
© 编码器和解码器块中的变换器:这种框架在U-Net的编码器和解码器中都使用了变换器,特别是采用了基于Swin Transformer的双尺度编码器子网络,用于提取不同语义尺度的粗粒度和细粒度特征表示。这种设计允许模型在不同尺度上捕捉图像特征,增强了对图像细节和整体结构的理解,从而可能提高分割任务的性能。
这些框架的共同目标是利用变换器的能力来处理组织学图像的复杂性和变异性,以提高分割任务的准确性和效率。通过在不同的模型部分集成变换器,研究者可以探索不同特征的整合和上下文信息的利用,进而推动组织学图像分割技术的发展。
3-1:在编码器和解码器块之间的Transformer:
Ji等人[26]提出了一种多化合物Transformer用于生物医学图像分割(参见图5(a)),包括公共Pannuke数据集中的细胞核分割。
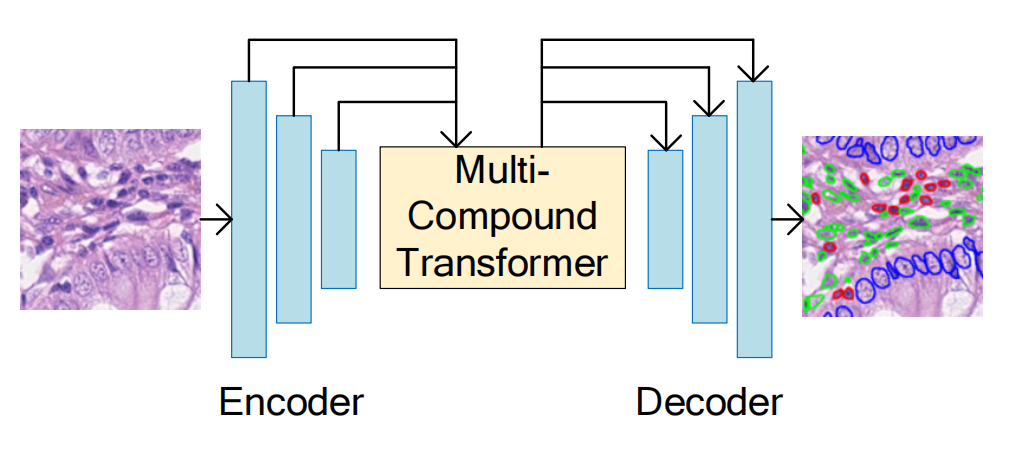
他们使用了Transformer自注意力模块和Transformer交叉注意力模块在U-Net编码器和解码器结构之间,旨在学习多个特征之间的上下文信息和不同标签之间的语义关系。实验结果表明,该方法在Dice相似系数(DSC)得分上优于U-Net模型及其变体。
Chen等人[82]提出了一种名为TransAttUnet的基于Transformer的注意力引导网络用于医学图像分割。TransAttUnet结合了Transformer自注意力模块和全局空间注意力模块,嵌入在编码器和解码器子网络之间,以捕获更广泛和更丰富的上下文表示。评估结果显示,TransAttUnet在公共数据集中的核和腺体分割任务上优于最先进的基线分割模型。
Wang等人[83]引入了一种通道Transformer模块来替换U-Net编码器和解码器之间的原始skip连接,以及一个通道间交叉注意力模块来融合解码器中的多尺度特征。他们的模型在腺体和细胞核分割任务上进行了测试,并显示了优于其他基于Transformer的方法,包括TransUNet[84]、Medical-Transformer[27]和Swin-UNet[85]。
Liao等人[86]提出了一种名为Swin-PANet的Swin Transformer辅助的先验注意力网络用于医学图像分割,包括组织学图像中的腺体和细胞核分割。Swin-PANet将Swin Transformer块和注意力引导解码器集成到先验注意力网络[87]中,通过使用从粗到细的学习策略来提高分割性能。
Li等人[88]提出了一种增强的Transformer混合分割网络ATTransUNet,其中Transformer层插入到U形编码器和解码器之间,用于组织学图像中的腺体和细胞核分割。ATTransUNet包括一个自适应令牌提取模块用于挖掘重要的视觉令牌,以及一个选择性特征增强模块用于鼓励网络关注主要语义特征。
Bao等人[89]在U-Net skip连接中应用了标准Transformer和池化Transformer,这有助于捕获更丰富的空间和语义信息以进行医学图像分割。在MoNuSeg数据集[90]上的实验结果表明,与其它方法相比,该方法在核分割性能上表现更优。
3-2:在编码器块中的Transformer:
Valanarasu等人[27]提出了一种名为Gated Axial Transformer的模块,用于U-Net编码器块中的医学图像分割。他们采用了一种全局-局部训练策略,同时学习高级信息和细节,以提高分割性能。在GLAS数据集[91]和MoNuSeg数据集[90]上的实验结果显示,该方法在基线分割模型上取得了更好的性能。
Zhang等人[33]提出了一种名为Pyramid Medical Transformer的方法,该方法包含三个Transformer分支和三个带有门控轴向注意力块的分支,以及一个CNN分支。三个Transformer分支分别捕捉图像像素之间的短程、中程和长程关系,而CNN分支提取多尺度图像特征。融合后的特征图用于生成分割结果。在GLAS数据集[91]和MoNuSeg数据集[90]上的实验结果显示,该方法比Valanarasu等人[27]的方法取得了更好的结果。
Yun等人[92]将Transformer块集成到U-Net编码器中,并在超光谱病理学图像中进行了胆管癌分割。他们采用了深度卷积[93]和光谱规范化来处理不同光谱之间的差异,并应用了具有稀疏约束的Transformer来学习分割任务中不同波段之间的有意义上下文。与专门为超光谱图像分割设计的比较方法相比,他们的方法取得了更好的结果。
Zhang等人[97]提出了一种CNN-Transformer混合网络,用于医学图像分割。该网络采用了CNN-Transformer串联编码器和双路径并行解码器,以有效学习上下文信息和处理多尺度特征图。Zheng等人[98]提出了一种双分支分割网络,其中CNN和Transformer作为特征编码分支,并采用交叉注意力和交叉尺度模块来融合多尺度特征。这两项研究[97]和[98]在GLAS数据集[91]上验证了他们的模型,并报告了略优于参考文献[97]的结果。
Dhamija等人[99]设计了一种双分支网络,其中一个分支包括Transformer编码器和CNN解码器,另一个分支是U-Net模型。他们在一个公开的核分割数据集[100]上报告了良好的性能。
通过使用基于Transformer的多实例学习框架,Qian等人[101]设计了一种弱监督的病理学图像分割模型,该模型在结肠癌分割上取得了优越的性能。他们采用了Swin Transformer[41]的前三个阶段作为多尺度特征编码器。三个解码器和多尺度特征融合分支用于进行像素级和图像级预测。深度监督用于增强对分层图像信息的利用,并促进模型的训练。
3-3:在编码器和解码器块中的Transformer:
Lin等人[79]提出了一种名为DS-TransUNet的双Swin Transformer U-Net,用于医学图像分割。该方法在U形分割框架的编码器和解码器中使用了分层Swin Transformer块。他们还引入了双分支编码器来提取多尺度特征表示,并使用了一种名为Transformer Interactive Fusion (TIF)的新模块,通过自注意力机制建立多尺度特征之间的长程依赖关系。
Pan等人[102]提出了名为EG-TransUNet的方法,该方法在U-Net编码器和解码器块中使用了多头自注意力,以学习全局上下文信息和融合多尺度特征。这两项研究[79]和[102]在GLAS数据集[91]和2018年数据科学碗[100]上进行了验证,并报告了比其他方法更好的性能。
3-4:Transformer基分割方法的比较:
为了系统地评估Transformer基网络在组织学图像分割中的性能,Nguyen等人[103]使用PAIP肝脏组织病理学数据集[104]对六种流行的Transformer基模型和六种CNN基模型进行了定量评估。评估的六种Transformer基模型包括:
- TransUNet[84]
- Swin-UNet[85]
- Swin Transformer[41]
- Segmenter[105]
- Medical-Transformer[27]
- BEiT[106]
他们的结果显示,Transformer基模型可以优于CNN基模型,并且Segmenter[105]在六种Transformer基模型中提供了最有前景的肝脏肿瘤分割性能。
这些研究结果表明,Transformer基的分割模型在组织病理学图像分析中表现出了显著的潜力,特别是在处理复杂的上下文信息和多尺度特征时。这些模型的性能提升得益于Transformer模块在捕捉全局上下文信息和特征间的依赖关系方面的优势。
然而,这些模型也面临着一些挑战,例如计算复杂性、模型解释性和在实际临床应用中的可扩展性。未来的研究需要继续探索这些挑战,并寻找更有效的方法来利用Transformer在病理学图像分析中的应用。
四、生存风险回归
与分类和分割任务处理分类目标变量不同,回归任务处理连续目标变量。
生存结果预测是一个具有挑战性的序数回归任务,旨在预测癌症死亡、疾病复发等相对风险。许多计算病理学研究已经探索了癌症患者的生存结果预测,例如生存CNNs[107]、注意力引导的深度多实例学习网络[108]、基于‘Noisy-And’池化的MobileNetV2模型[109]、弱监督深度序数Cox模型[110]等。
本研究不会回顾所有这些关于生存结果预测的工作。然而,由于分类和生存回归任务之间存在巨大差异,本节首先回顾了生存风险预测的基础知识,然后列出了相关的ViT-based研究。
4-1:组织学生存风险回归:
患者的生存数据通常是弱标记的,每个患者只有一个结果标签。同时,WSI通常非常大,可以被分割成成千上万的小图块。然而,其中许多图块可能对预测任务不具有信息性(即噪声图块)。这使得弱监督学习成为训练生存回归模型的合适方法。
ViT模型在考虑全局依赖性的同时,具有将多个图像图块的特征融合的优势,它们在生存结果预测方面显示出巨大的潜在应用。Shen等人[28]展示了使用ViT作为骨干,并结合卷积操作,是使用肺癌WSI预测患者生存风险的有效方法。由于WSI中的图块数量过多,ViT模型无法全局学习,因此他们应用卷积层将相邻的小图块聚合成更大的图块,以减少进入基于Nystrom的Transformer块的连续图块数量。
Chen等人[29]引入了一种新的ViT架构,称为分层图像金字塔Transformer (HIPT),它应用了基于生存交叉熵损失[113]的分层自监督学习ViT模型进行生存风险预测。这种HIPT架构对视觉令牌进行了嵌套聚合,这些令牌从区域级(即4096×4096像素)递归地分解为更小的令牌,直至到图块级(即256×256像素)和细胞级(即16×16像素)。通过DINO方法[36]进行分层自监督学习,用于预训练图块级和区域级的ViT块,以有效进行模型训练。尽管HIPT架构已经在多种癌症类型的生存风险预测中进行了评估,但他们的结果并不令人鼓舞,报告的C指数低于0.67。
Huang等人[114]使用SimCLR方法训练Resnet18作为特征提取器,从随机选择的图块中计算图像特征,然后应用Transformer来聚合组织学特征以预测患者生存风险。在TCGA鳞状细胞癌、乳腺癌浸润性癌和卵巢浆液性囊腺癌上的实验结果显示,其性能略优于其他比较方法(例如,C指数约为0.7)。
Jiang等人[115]开发了一种多头注意力方法用于生存预测,使用预训练的Resnet18提取图块级特征,然后使用多头注意力机制进行聚合。他们评估了多种TCGA数据集上的生存预测,并报告了改进的性能。
4-2:组织学和基因组生存风险回归:
除了WSI之外,基因组信息可以作为另一种模态进行联合学习,以产生更准确的生存预测。
Chen等人[111]提出了一种基因组指导的联合注意力Transformer,用于学习WSI特征嵌入,并使用基于MIL的Transformer集成功将组织学和基因组特征整合用于生存预测。他们的方法的流程图如图6所示,包括两个主要阶段。
Fig. 6 描述了一个基于基因引导的共同注意变换器(Gene-guided Co-attention Transformer),用于从整个幻灯片图像(Whole Slide Images, WSIs)中预测生存风险。
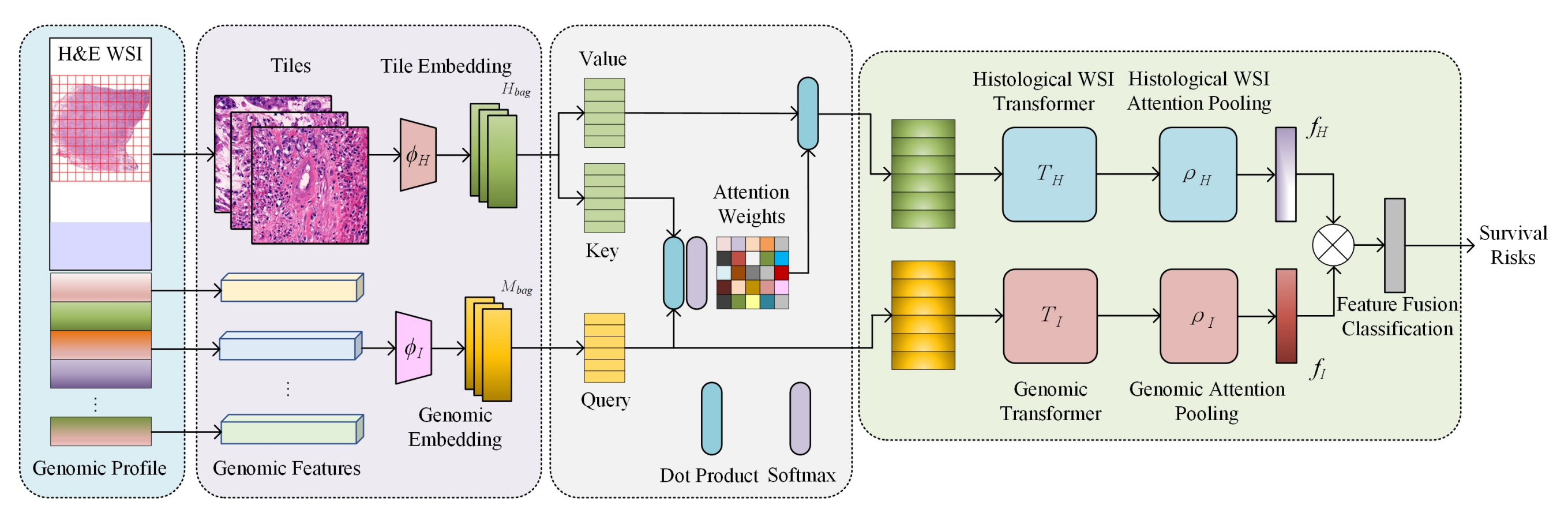
-
WSI的分割与特征编码:首先,将WSI分割成多个小块(tiles),这些小块被编码成特征表示。这一步骤通常涉及到将图像数据转换为能够被深度学习模型处理的数值形式。
-
基因组数据的特征编码:与WSI的图像特征同时,基因组数据(genomic profiles)也被编码成特征表示。这可能包括从基因表达数据中提取的特征,这些特征与癌症的发展和患者的生存结果有关。
-
特征映射:
- 图像特征被映射到值(value)矩阵和键(key)矩阵。
- 基因组特征被映射到查询(query)矩阵。
-
基因引导的注意力加权:利用基因组数据,通过注意力机制对组织学特征进行加权,生成图像表示。这一步骤意味着模型会根据基因组信息的重要性来调整图像特征的权重,从而突出对生存预测最重要的组织学特征。
-
变换器处理:加权后的图像特征和基因组特征分别输入到视觉变换器(ViT)和注意力池化(attentional pooling)模块中。这些模块能够进一步提取和精炼特征,为生存风险预测提供更丰富的信息。
-
特征整合:从ViT和注意力池化模块学习到的成像和基因组特征被连接(concatenated)在一起。这一整合步骤为模型提供了一个综合的、多模态的视角来评估患者的生存风险。
-
生存风险预测:最后,整合的特征被用于预测患者的存活风险。这通常涉及到一个下游的任务特定模型,如多层感知器(MLP)或其他回归模型,来预测患者的生存时间或风险。
总的来说,Fig. 6 展示的是一个多模态学习框架,它结合了组织学图像分析和基因组数据分析,通过共同注意机制来提高癌症患者生存预测的准确性。通过这种方式,模型能够同时考虑来自不同数据源的信息,以获得更全面的疾病表征。
受基因组指导的联合注意力Transformer[111]的启发,Li等人[116]进一步提出了一种模型,用于聚合分层组织学图像特征和基因组概况进行生存预测。他们的模型在参考文献[111]使用的相同数据集上进行了验证,并显示了整体提高的C指数值。
Wang等人[117]提出了一种可以同时学习组织学嵌入特征和基因表示的多模态Transformer编码器。将连接的多模态特征输入到MLP中,基于负Cox对数偏似然损失函数预测生存风险。在TCGA鳞状细胞癌(LUSC)和卵巢浆液性囊腺癌(OV)上的评估显示了出色的性能,C指数约为0.75。
Lv等人[118]提出了一种名为TransSurv的基于Transformer的生存分析模型,用于结直肠癌,该模型通过Transformer模块整合了多尺度成像特征和基因组数据。实验证明,TransSurv能够有效地整合组织学图像和基因组概况的多模态特征进行生存风险预测。
这些研究展示了ViT在组织学图像分析中的应用,特别是在处理复杂的多模态数据和连续目标变量时。ViT模型的优势在于其能够捕捉全局上下文信息和特征间的依赖关系,这有助于提高生存预测的准确性。然而,这些模型也面临着计算复杂性、模型解释性和在实际临床应用中的可扩展性的挑战。
未来的研究需要继续探索这些挑战,并寻找更有效的方法来利用ViT在病理学图像分析中的应用。
五、可解释性
深度学习因其缺乏可解释性而受到批评,被视为一个黑箱方法[124]。
ViT基于模仿人类视觉识别某些功能的注意力机制开发而成;然而,其决策仍然缺乏足够的可解释性。
与计算机视觉的其他任务不同,在医生可以接受并将其部署到真实临床环境中之前,提供视觉解释和清晰的推理逻辑来解释诊断AI模型是至关重要的。同时,由于注释组织学图像数据的困难,通过语义层次上的人机通信进行学习有助于提高深度学习性能。
因此,从组织学AI模型生成视觉解释,并将原因与效果相关联是亟待解决的关键挑战。
为了解释深度学习模型,已经进行了许多尝试,旨在可视化卷积层学习到的模式或特定类的热图。
例如,广泛使用的去卷积[125]和引导后向传播[126]方法被设计用于将特征激活映射到原始输入像素空间,以解释中间层中的特征活动。
Fig. 9 展示了使用引导后向传播方法[126]可视化Resnet18模型[127]中学习到的肿瘤浸润淋巴细胞(TILs)检测器图像特征的示例。
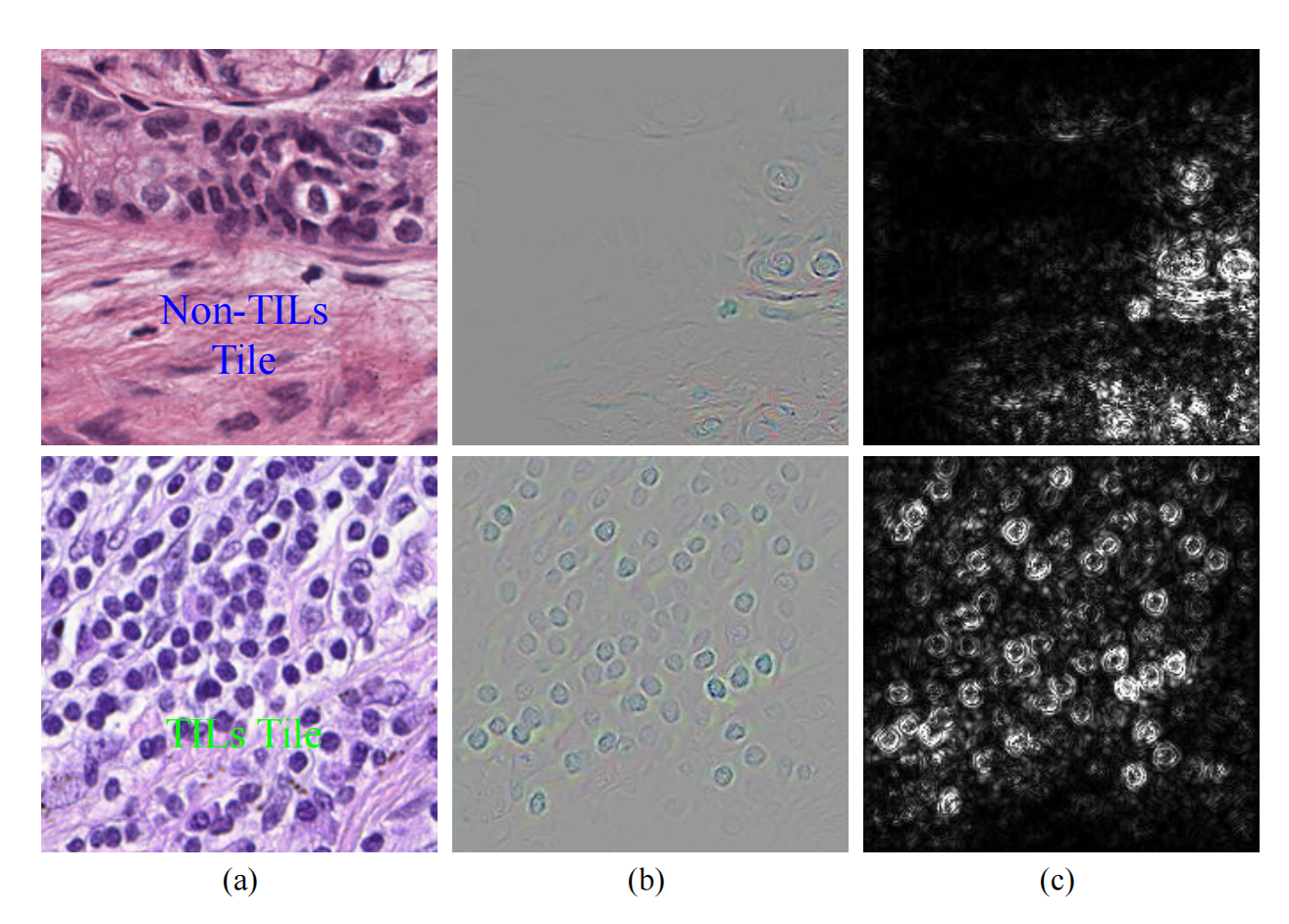
以下是对图的详细分析:
(a) TILs和非TILs图像:展示了两行图像,第一行是非TILs(Non-TILs)图像,第二行是TILs图像。这些图像是模型分类的基础。
(b) 彩色图像重建:通过GBP方法重建的彩色图像,显示了模型在做出分类决策时所依赖的区域。这些区域在图像中以亮色(高激活区域)的形式出现。
© 灰度图像重建:与彩色图像相同,但以灰度显示,更清晰地展示了高激活区域。
-
非TILs图像的可视化:在第一行图像中,由于病理学家将其归类为非TILs,因此高激活区域(在灰度图像中显示为更亮的区域)对应于背景和其他非TILs区域。这些区域支持模型做出非TILs的分类决策。
-
TILs图像的可视化:相比之下,第二行图像中的高激活区域主要对应于淋巴细胞区域和边界,这些区域支持模型做出TILs的分类决策。
GBP方法通过将模型的预测反向传播到输入图像,来识别对模型决策最为重要的特征区域。在Fig. 9中,GBP用于展示Restnet18模型是如何通过识别淋巴细胞的特征来区分TILs和非TILs图像的。这种可视化有助于理解模型的决策过程,并为病理学家提供模型预测的透明度和解释性。通过这种方式,可以更好地评估模型的可靠性,并在必要时进行调整或验证。
类激活映射(CAM)[128]和梯度CAM(grad-CAM)[129]方法也被开发出来,以定位与CNN模型分类决策相关的特定类图像区域。这些可视化策略将为使ViT模型更具可解释性提供宝贵的指导。
特别是,grad-CAM[129]已被采用来突出显示Swin Transformer-based MIL方法[70]中结直肠腺瘤病变分类中高度相关的区域。GraphCAM方法[69]被设计用于从其图表示构建WSI的类激活映射。
此外,由于ViTs中使用的注意力机制,通常可以制作注意力热图来解释不同特征令牌(或图像区域)对预测的贡献。典型的研究已经生成了注意力热图,以可视化肾细胞癌亚型[24]、组织学组织形态表型的[35]和乳腺癌转移检测[73]。
尽管这些研究促进了深度学习模型可视解释的发展,但仍然是一个挑战,即发现深度学习模型的直观解释,包括ViTs。
六、临床关注
在临床应用中开发和部署ViTs与风险和责任相关。
一个关注点是模型的稳健性,这在医疗保健应用中非常重要,因为它直接影响患者的结果和安全性。AI模型在诊断过程中经常出现的错误和不一致通常源于组织学数据的异质性。打算在临床实践中部署的组织学ViT模型应该能够跨实验室泛化,并且对染色变化具有稳健性。否则,它们将提供不可靠的诊断和建议,这可能导致严重后果,包括错过患者治疗的最佳时机。
另一个关注点是模型的可解释性,这是了解不透明的决策制定的关键。为了揭示ViTs使用的组织学特征,将临床医生与机器之间的互动纳入其中至关重要,因为这有助于利用临床知识并为模型预测提供更清晰的解释[1]。最后,ViTs的发展通常需要大量数据,这些数据可能来自不同临床机构的收集[13]。这可能会引发获取患者数据的所有权的监管和隐私问题。这些临床关注点为ViTs在计算组织病理学中的应用带来了额外的挑战。
为了解决这些挑战,有必要开发可解释的ViTs,并使用来自不同诊所的更多样化的数据集进行验证。为了避免直接数据共享并保护隐私,使用ViTs的联邦学习[130]可能是多中心验证的有前景的解决方案。
七、不同Transformer类别
7-1:CNN与Transformer
在ViTs出现之前,CNN是计算组织病理学的主流方法。
CNN使用像素数组,而ViTs将输入图像分割成视觉令牌。CNN在低层阶段提取的特征与高层阶段提取的特征有很大不同,因为网络越深,感受野逐渐扩大[20]。CNN在捕捉局部结构方面具有优势,这是由于其局部性和权重共享机制;然而,它们在捕捉图像区域之间的全局关系方面存在局限。
相比之下,自注意力操作使ViTs能够从全局视角聚合特征,从而为理解远距离关系提供良好的理解[17]。然而,ViTs通常显示出较弱的归纳偏差,因此在训练小型数据集时,对模型正则化或数据增强的依赖性增加。尽管ViTs已显示出是前景广阔的AI解决方案,但现有研究从未证明ViTs在所有情况下都能优于CNN,特别是在少样本和低分辨率医学图像分析中[17]、[13]、[131]、[132]。
因此,作者预期会有更多CNN和ViT混合模型被开发出来以解决组织学图像分析任务。在计算病理学领域,WSIs和注释的可用性通常有限。CNN和ViT混合模型,由于其消耗相对较少的GPU内存且易于训练,可以缓解这一问题。
此外,WSI具有包含多尺度特征的独特特性。为了提取这些特征,将使用在多分辨率图像上工作的卷积操作,如在[33]、[27]中提到的那样。
7-2:图Transformer
在组织病理学图像中,组成组织学实体的表型和拓扑分布对癌症诊断至关重要。
因此,为了处理这些关系感知的表示,一些现有研究使用图算法量化了不同类型细胞核之间的空间交互,例如[133]、[134]、[135]。
为了进一步利用深度学习的优势,图神经网络(GNNs)已被探索并引入,用于编码组织表示,并捕获实体级别的内部和外部交互。GNNs在各种任务上取得了显著的成功,正如最近关于图深度学习框架及其应用的调查所强调的,包括乳腺癌诊断、结直肠癌分级等[31]、[136]、[137]。
此外,图表示可以增强最终表示的解释性,通过建模感兴趣区域(例如细胞核和组织成分)之间的关系,从而生成可解释的诊断结果映射。
由于Transformer强大的学习远距离交互的能力以及WSI中细胞、组织和其他成分之间的固有空间关系,越来越多的尝试将Transformer融入GNN架构中[138]。
在一个开创性工作中,Zheng等人[69]构建了一个图网络,并引入了一个Transformer层,该层选择图中最显著的节点并通过注意力机制聚合信息,从而实现了WSI级别的分类,例如肺癌亚型的分类。这项研究展示了优于其他广泛使用的深度学习模型的分类性能,并提供了类别特定的可视化。
总体而言,作者主张越来越多的组织学图像分析研究将ViTs与图结合起来,以更准确和可解释地评估WSI。
7-3:多任务和多模态Transformer
在医学图像分析中,AI模型能够同时解决多个任务(例如癌症诊断和亚型分类)是非常有价值的,因为医生通常对估计患者的不同临床终点感兴趣。
据报道,构建具有多个任务的模型有助于提高泛化能力[17],这对训练更有效的深度学习模型是有益的。因此,多任务学习在计算病理学中吸引了相当多的关注。
例如,核分割和分类已合并为一个模型,在SOTA研究中,如Hover-Net[139]和PointNu-Net[140]。为WSI中的癌症检测和亚型分类设计了一个多任务学习框架,并在多个TCGA数据集上展示了卓越的性能,在准确性和泛化能力方面。还有一些最近的ViT-based模型能够同时执行分割和分类任务[142]、[143],尽管这些研究关注的是皮肤镜和放射学图像。
然而,作者预期会有越来越多的ViT-based多任务学习模型被开发出来以解决组织学图像分析任务。多模态数据可以为癌症诊断、分级和预后提供互补证据,尽管由于不同模态之间的异质统计特性和噪声水平,有效学习联合表示一直具有挑战性。存在不同的融合特征表示的策略,例如向量拼接、元素间求和、元素间乘法、双线性池化和注意力池化[111]。使用注意力机制设计的Transformer为跨模态学习联合表示提供了另一种有前景的策略。
特别是,WSI的组织学特征和基因表达数据已通过Transformer编码器块联合学习并融合在一起,以预测癌症患者的生存风险[111]、[117]。在一个先前的研究中[111],将基因组特征打包成一个查询矩阵,以指导组织学图像特征的注意力加权(参见图6)。
然而,Wang等人[117]认为基因数据是噪声表示,因此每个基因数据的隐藏表示都从组织学图像特征诱导出来,并在注意力的指导下更新自己(参见图10)。
Fig. 10 描述了一个用于处理多模态数据的变换器编码器的示意图,特别是针对组织学图像(Whole Slide Images, WSIs)和基因组数据。
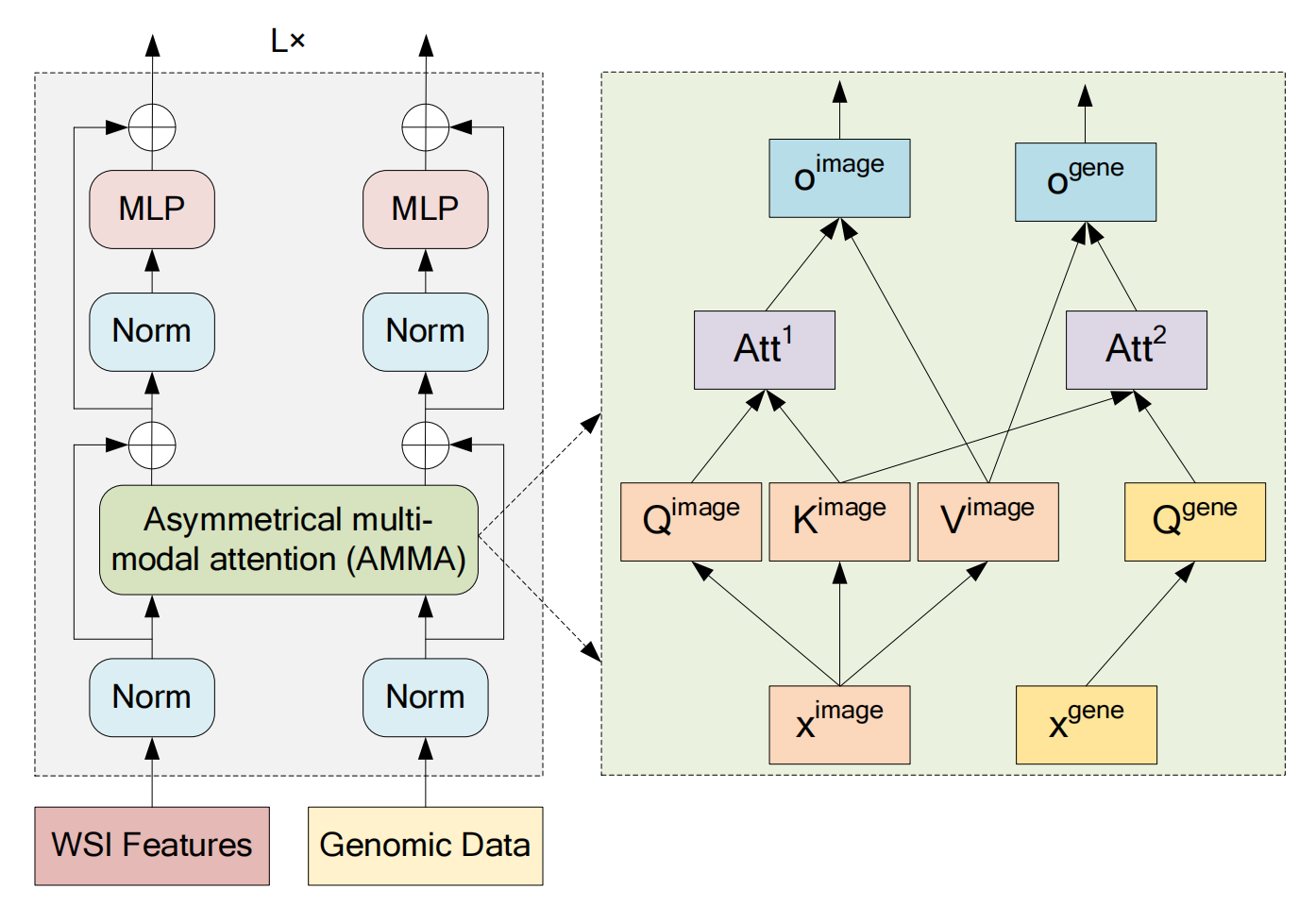
以下是对图的分析:
左侧部分:多模态数据的预处理和编码
- 归一化处理:WSI特征和基因组数据首先被归一化处理,以确保数据的一致性和稳定性。
- 编码:归一化后的数据通过非对称多模态注意力(Asymmetrical Multi-Modal Attention, AMMA)模块进行编码。
右侧部分:AMMA模块的工作原理
- 自注意力操作:WSI特征(表示为 ( x_{\text{image}} ))通过自注意力机制进行处理,这允许模型在不考虑序列位置的情况下捕捉特征之间的相关性。
- 基因数据的隐藏表示:基因数据的隐藏表示(表示为 ( x_{\text{gene}} ))是由组织学图像特征引导的,并在注意力的引导下更新。这意味着模型在处理基因数据时,会参考图像特征来调整基因数据的表示,因为基因数据相对于组织学图像来说可能更嘈杂且重要性较低。
多模态特征转换
- 归一化和多层感知器(MLP)层:经过AMMA模块编码后,每种模态的数据(即WSI特征和基因组数据)分别通过归一化和MLP层进一步转换,以利用每种模态的内在信息。
多模态特征融合
- 特征融合:经过上述处理后,WSI的成像特征和基因组特征被结合起来,以便于进行生存风险预测。这种融合策略允许模型同时考虑来自不同数据源的信息,从而可能提高预测的准确性。
应用场景
- 生存风险预测:这种多模态变换器编码器的设计用于预测癌症患者的生存风险,通过结合组织学图像分析和基因组数据分析,以期提供更全面和准确的预测结果。
总体而言,Fig. 10 展示的多模态变换器编码器通过AMMA模块有效地整合了来自不同数据源的信息,并通过自注意力机制和多层感知器层进一步提取和利用了每种模态的内在特征,以提高生存风险预测的性能。
受到这些新颖探索的启发,可以开发更多涉及Transformer的多模态数据研究,以更好地满足临床需求。
八、结论
由于现有CNN模型在构建长程依赖关系和捕获全局上下文信息方面的局限性,Transformer在医学图像分析领域,包括计算组织病理学中,正在迅速增长。
尽管已经有关于ViT在医学图像分析中应用的广泛综述,但目前缺乏专门关注ViT在计算组织病理学中使用的综述。由于ViT模型已成为深度学习中的最先进技术,在本调查中,作者全面回顾了在计算组织病理学图像分析背景下开发的ViT模型。
通过主要在过去两年内发表的文献的综述,作者发现ViT-based模型已被探索用于分类、分割和生存风险回归等多种组织学任务。实验倾向于证明,与CNN-based模型相比,ViT-based模型可以提供相当或甚至更优越的性能。
本调查中呈现的方法论为探索文献中可用的ViT模型提供了参考指南。作者还讨论了该领域的一些开放问题和未来趋势,以帮助读者探索ViT模型在计算组织病理学领域的应用。














![[数据集][目标检测]集装箱缺陷检测数据集VOC+YOLO格式4127张3类别](https://i-blog.csdnimg.cn/direct/88ef9ef0679a4468914c2a2ad44956d6.png)



