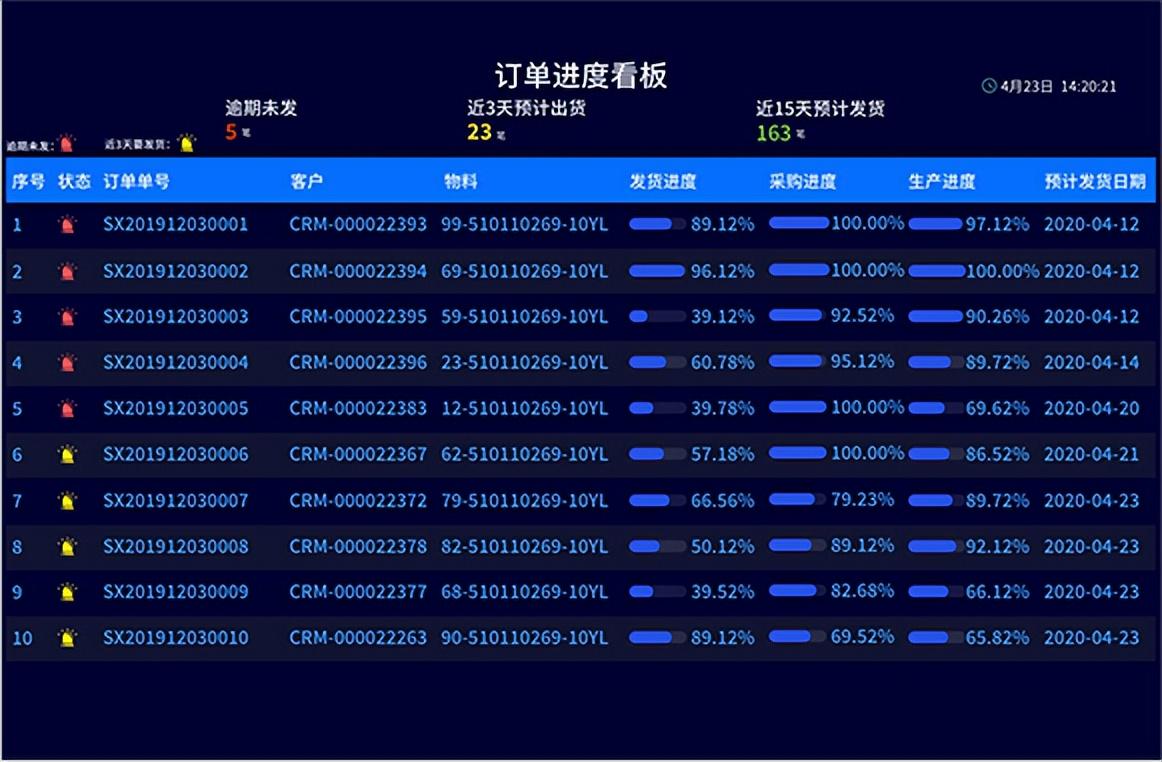
大家好,这是专注表观组学十余年,领跑多组学科研服务的易基因。今天跟大家介绍一下易基因的新产品:细菌全基因组甲基化纳米孔测序(ONT)。
表观修饰不需要改变DNA序列便能实现对性状的改变,表观修饰的改变与基因功能乃至细胞状态、发育、衰老、疾病等存在重要的关联。在众多的表观遗传修饰中,最为重要且研究最为广泛的修饰之一是DNA甲基化。
由于PCR的扩增过程会消除碱基修饰信息,通过传统测序技术对其进行检测需要使用特殊的文库制备步骤,从而造成核酸损坏,最终导致测序读长序列非常短。
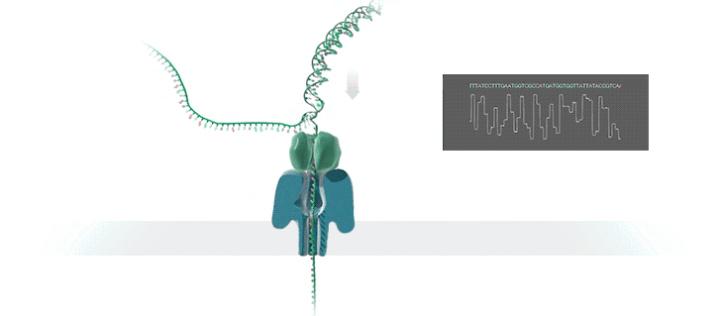
Nanopore测序是Oxford Nanopore Technologies(ONT)公司开发的基于纳米孔电信号的单分子实时测序技术,DNA双链在马达蛋白的作用下与锚定在生物膜上的八聚体纳米孔蛋白结合并解螺旋。由于生物膜两测存在电势差,解旋后的单链DNA以一定速率通过纳米孔。由于化学性质不同,不同碱基通过纳米孔时,会引起不同电信号的变化。通过对这些电信号变化进行检测和解析,完成序列的实时测定。Nanopore的碱基判读是依据其电流信号而产生,因此其判定过程比较复杂。目前Nanopore根据电流的大小及电流大小的变化情况,通过“递归神经网络(Recurrent Neural Network)”的复杂算法对碱基进行判读。
ONT全基因组甲基化测序原理示意图如下:
技术优势:
长读长:平均读长>10Kbp,最长可达2M左右,直接跨越重复序列、高度多态性区域等特殊区域;
直接测序:无需PCR扩增,可直接保留并检测DNA甲基化修饰;
全基因组覆盖:可同时检测全基因组范围单碱基的5mC与6mA修饰位点,并给出单碱基的甲基化水平;
性价比高:ONT测序成本相对于二代NGS测序技术大幅降低。
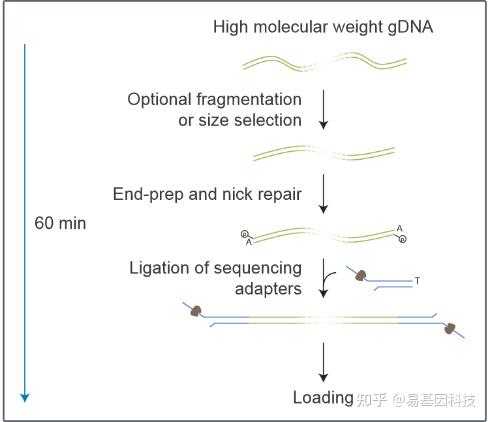
实验策略:
分析内容:
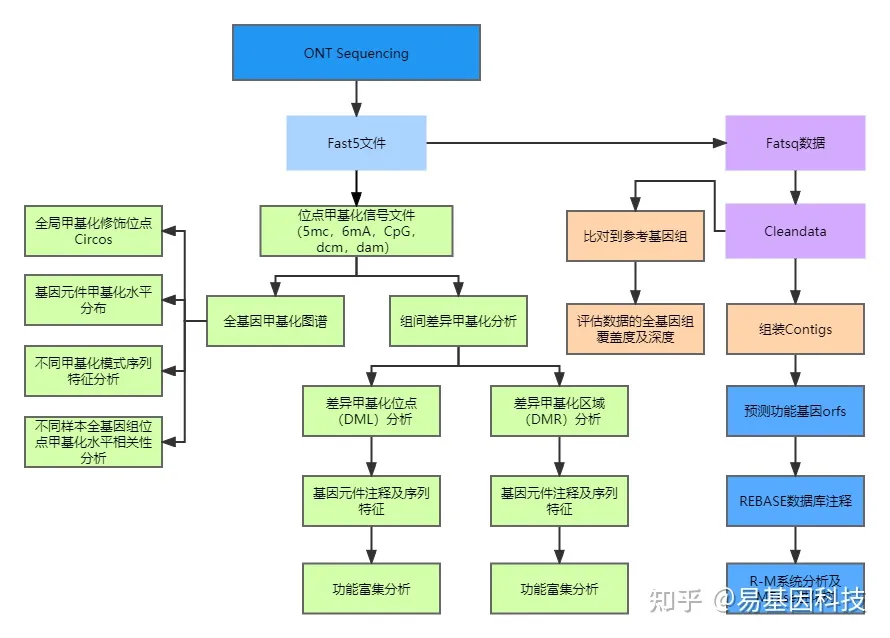
注:个性化分析、多组学关联分析请联系对接销售或技术支持
送样要求:
送样要求 | 项目周期 |
样本类型:细菌、菌泥样品DNA需求量:不同样本需求不同,细菌样本≥2μg;样品DNA纯度:OD260/280=1.8~2.0;样品DNA完整性:DNA无明显降解,需提供凝胶电泳检测胶图;其它注意事项:①最终DNA量以我们的检测结果为准;②若DNA总量超过样品要求,可要求保存样品; | 35-40个工作日。 |
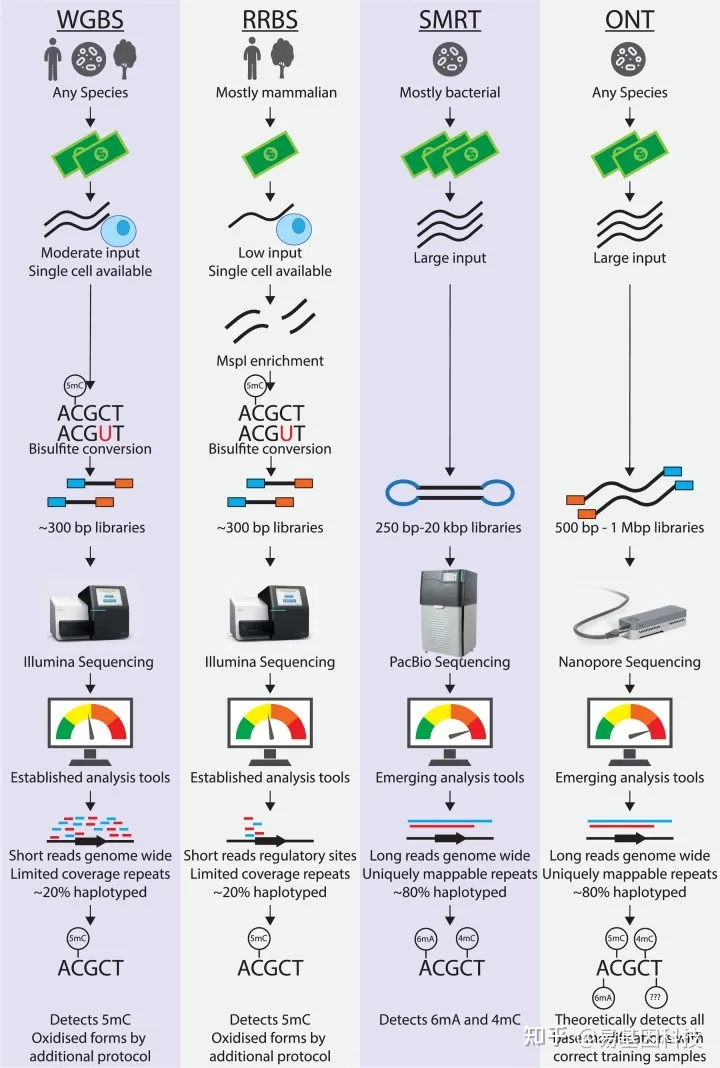
不同DNA甲基化测序技术比较:
经典案例
Engineering selectivity of Cutibacterium acnes phages by epigenetic imprinting
通过表观遗传印迹研究痤疮杆菌(C.acnes)噬菌体的选择
背景
痤疮杆菌(C.acnes)是一种革兰氏阳性细菌,是人类皮肤微生物组成员。尽管是最丰富的皮肤共生体,但某些成员与常见的炎症性疾病(如痤疮)有关。各种C.acnes分支的完整基因组序列可以鉴定推定的甲基转移酶,其中一些可能属于保护入侵DNA宿主的限制性修饰(R-M)系统。然而,关于这些系统是否在不同的C.acnes菌株中起作用,目前知之甚少。
方法
通过Oxford Nanopore Technologies(ONT)测序分析了六种代表性痤疮C.acnes菌株的甲基化水平。
结论
在菌株KPA171202中确定的DNA共有序列上检测到6mA修饰,且该R-M系统的重组表达证实了其甲基化活性,而R-M敲除突变体验证了该菌株甲基化特性的缺失。此外还研究了C. acnes噬菌体(PAD20)杀死各种C. acnes菌株的潜力,并将其特异性的增加与甲基化敏感株获得的噬菌体DNA甲基化联系起来。研究证明R-M缺失菌株中繁殖的噬菌体选择性地杀死R-M缺失痤疮敏感分支,而益生菌保持对噬菌体感染的抗性。
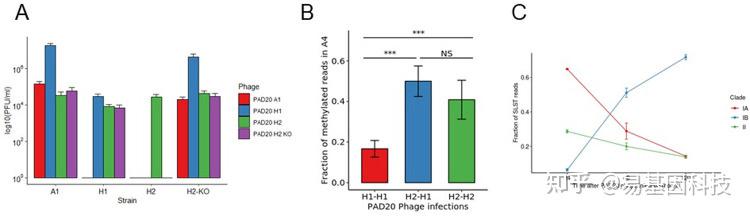
ONT测序揭示C. acnes KPA171202的R-M系统IIIB影响PAD20噬菌体感染特性并保护细菌免于裂解。
参考文献:
Knödlseder N, et al. Engineering selectivity of Cutibacterium acnes phages by epigenetic imprinting. PLoS Pathog. 2022 Mar;18(3):e1010420.
Gouil Q, Keniry A. Latest techniques to study DNA methylation. Essays Biochem. 2019 Dec 20;63(6):639-648.
相关阅读:
技术推介|RNA m7G甲基化测序(m7G-MeRIP-seq)
技术推介|高通量单细胞甲基化测序技术介绍(sc-RBS)
技术推介|RNA m⁶A甲基化测序(MeRIP-seq)技术介绍
技术推介 | 染色质免疫共沉淀测序 (ChIP-seq)
技术推介|细菌全基因组甲基化纳米孔测序(ONT)