内容如下:
1.外泌体和肝癌TCGA数据下载
2.数据格式整理
3.差异表达基因筛选
4.预后相关外泌体基因确定
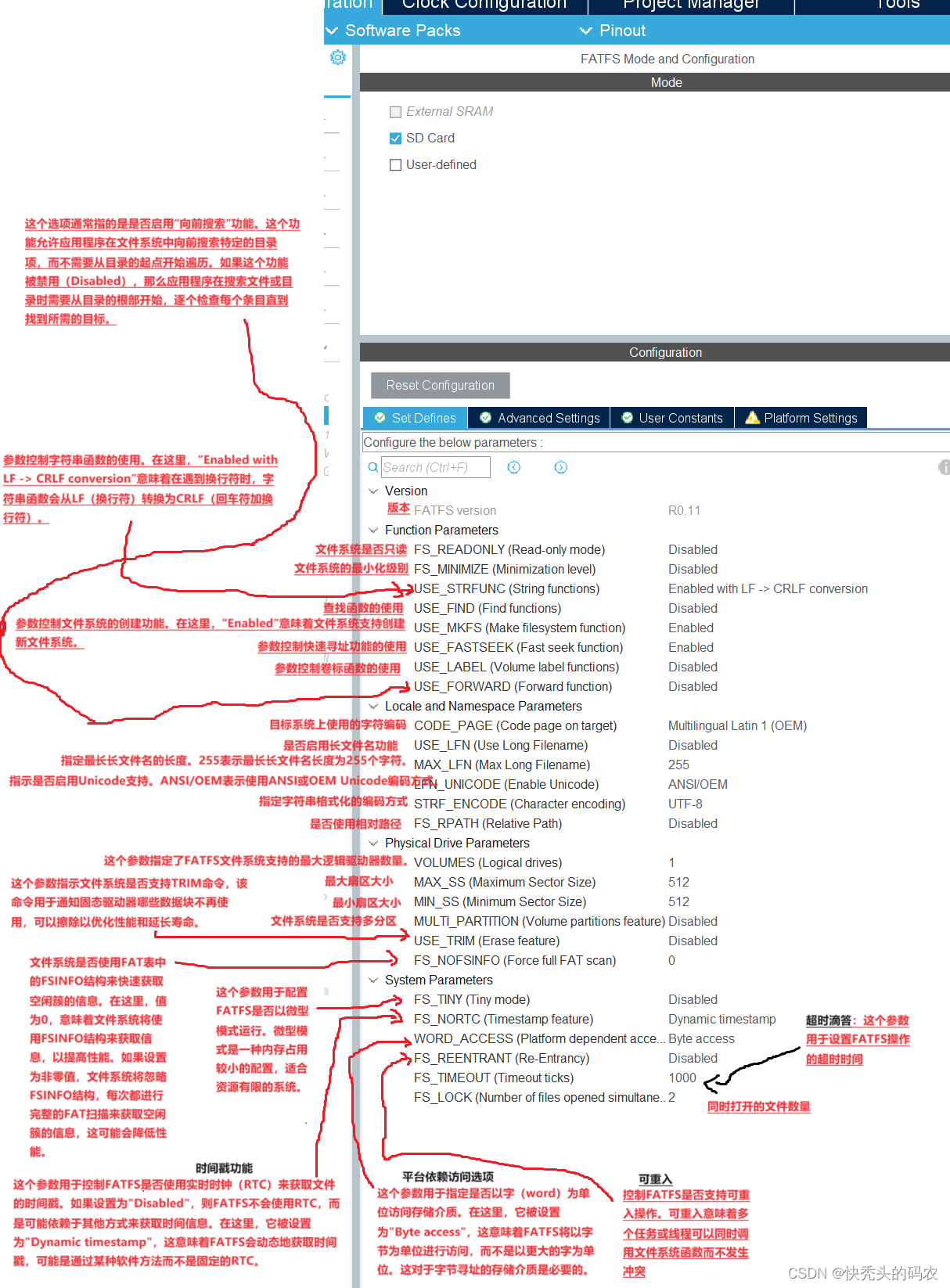
5.拷贝数变异及突变图谱
6.外泌体基因功能注释
7.LASSO回归筛选外泌体预后模型
8.预后模型验证
9.预后模型鲁棒性分析
10.独立预后因素分析及与临床的相关性分析
11.列线图,ROC曲线,校准曲线,DCA曲线
12.外部数据集验证
13.外泌体模型与免疫的关系
14.外泌体模型与单细胞测序
########################### 4.预后相关外泌体基因确定 ############################
下面进行批量单因素cox回归分析。
使用上一节的数据,代码如下:
setwd("C:\\Users\\ASUS\\Desktop\\自噬")
## install.package("pheatmap")
dir()
data <- read.csv("LIHC_clinical_mRNA_expression_data.csv",header = T,sep = ",")
data[1:5,1:5]
rownames(data) <- data$sampleID
data <- data[,-1]
data[1:5,1:5]
pFilter=1
names(data)[1:2] <- c("futime","fustat")
head(data)
dim(data)
data[1:5,1:5]
rt <- data
anyNA(data)
data <- na.omit(data)
outTab=data.frame()
sigGenes=c("futime","fustat")
dim(data)
head(data)
library(survival)
for(i in colnames(rt[,3:ncol(rt)])){
cox <- coxph(Surv(futime, fustat) ~ rt[,i], data = rt)
coxSummary = summary(cox)
coxP=coxSummary$coefficients[,"Pr(>|z|)"]
if(coxP<pFilter){
sigGenes=c(sigGenes,i)
outTab=rbind(outTab,
cbind(id=i,
HR=coxSummary$conf.int[,"exp(coef)"],
HR.95L=coxSummary$conf.int[,"lower .95"],
HR.95H=coxSummary$conf.int[,"upper .95"],
pvalue=coxSummary$coefficients[,"Pr(>|z|)"])
)
}
}
outTab
write.table(outTab,file="uniCox_LTPM.txt",sep="\t",row.names=F,quote=F)
uniSigExp=rt[,sigGenes]
uniSigExp=cbind(id=row.names(uniSigExp),uniSigExp)
write.table(uniSigExp,file="uniSigExp_LTPM.txt",sep="\t",row.names=F,quote=F)
从数据中可以看到,和预后有关的基因有35个。
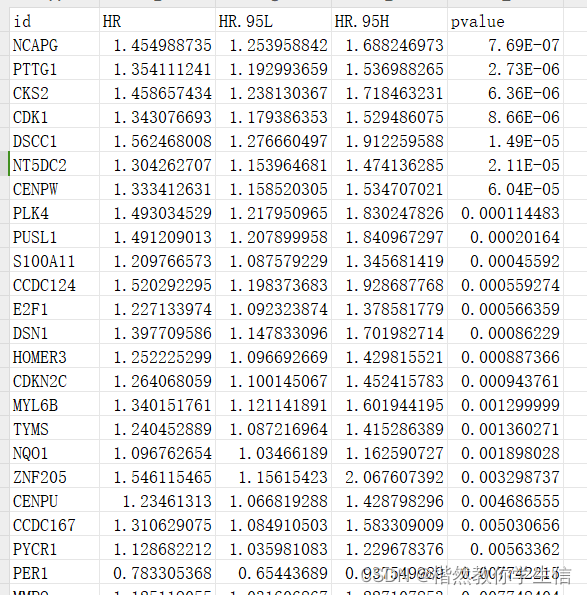
下一节绘制森林图。