在蛋白质结构预测和功能预测领域,基于机器学习的方法最近取得了显著的进展。特别是深度学习技术在这个领域中展现出了强大的能力,代表性的技术有 DeepMind 的 AlphaFold 和 RoseTTAFold。这些技术利用了大量的生物数据和先进的神经网络架构,极大地推动了蛋白质研究的边界。
1. AlphaFold
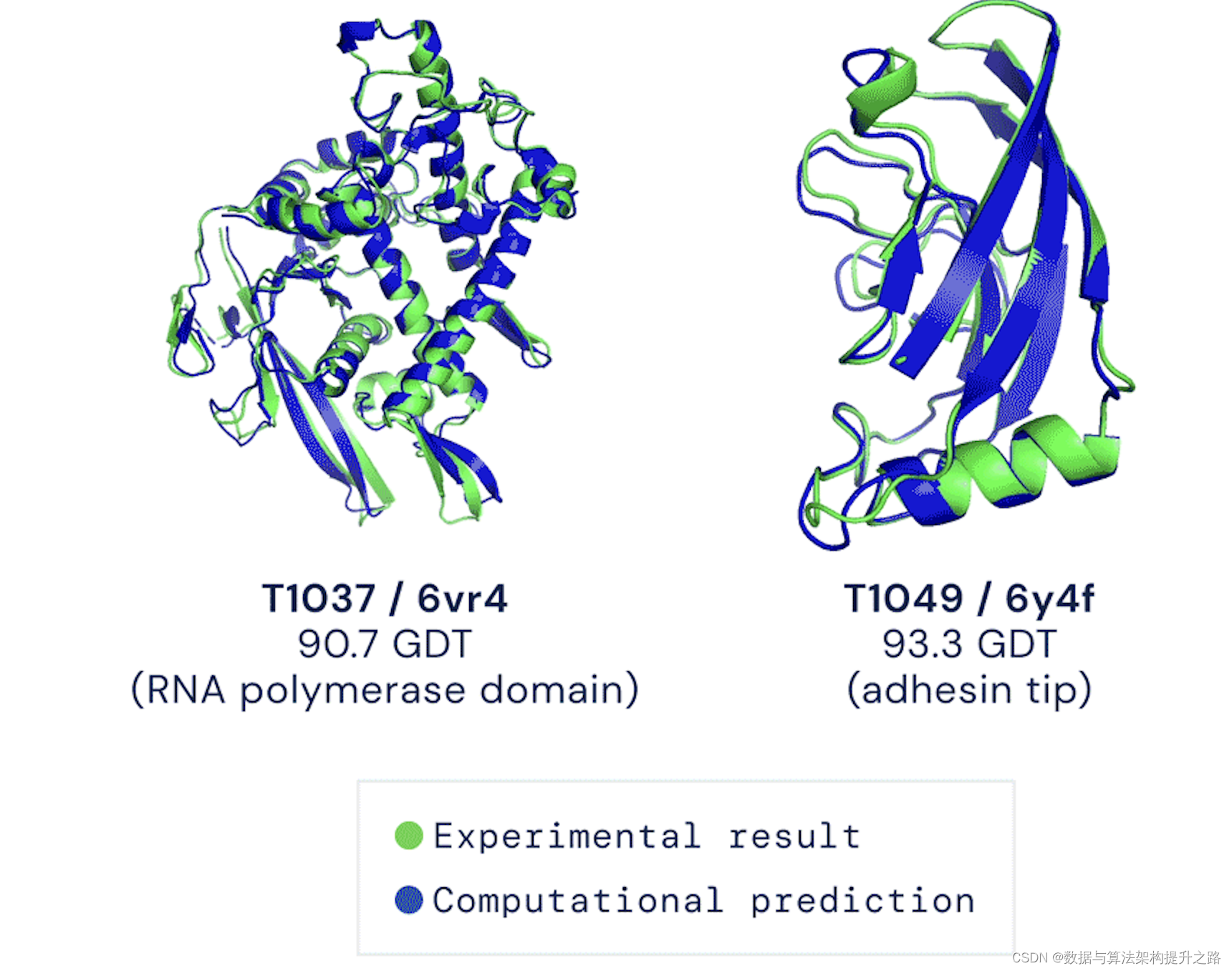
DeepMind 的 AlphaFold 是近年来蛋白质结构预测领域的突破性成就。AlphaFold 通过使用深度学习算法预测蛋白质的三维结构,其准确度在2020年CASP14比赛中被证实远超其他方法。AlphaFold 的核心是一个基于深度学习的模型,它能够预测蛋白质的氨基酸残基之间的距离和角度分布。
技术要点
- 结构预测:AlphaFold 使用了一个深度学习网络,该网络预测残基对之间的距离和角度。
- 多序列比对:利用多序列比对输入来增强预测的准确性。
- 注意力机制:使用了注意力模型来处理蛋白质的序列信息,使模型能够集中于序列中最相关的部分。
- 端到端训练:整个模型是端到端训练的,直接预测蛋白质的三维坐标。
开源地址
AlphaFold GitHub
运行条件
运行 AlphaFold 最简单的方法是使用提供的 Docker 脚本。我们在 Google Cloud 上测试了该脚本,测试机器有 12 个 vCPU、85 GB RAM、100 GB 启动盘、数据库位于额外 3 TB 磁盘上以及一个 A100 GPU。首次运行时,请按照安装和运行第一个预测nvidia-gpu-cloud-image部分 的说明进行操作 。
2. RoseTTAFold
由华盛顿大学开发的 RoseTTAFold 也是一种基于深度学习的蛋白质结构预测工具,它类似于 AlphaFold,但结构稍有不同,且计算效率更高。RoseTTAFold 通过一个三轨神经网络架构进行蛋白质的结构预测,这种架构可以同时处理一维序列和二维相互作用图。
技术要点
- 三轨网络架构:包括处理一维序列特征、二维配对特征和三维结构特征的网络。
- 快速预测:与 AlphaFold 相比,RoseTTAFold 在保持相近的预测精度的同时,提供了更快的预测速度。
- 广泛的应用:除了蛋白质结构预测,还可以用于预测蛋白质与RNA等其他生物分子的相互作用。
开源地址
RoseTTAFold GitHub
3. ESMFold
ESMFold 是一个新兴的蛋白质结构预测工具,由 Meta AI(以前是 Facebook AI)开发。它采用了类似于 AlphaFold2 的深度学习方法,但显著地提高了预测的速度和效率,同时在准确性上与 AlphaFold2 相当。ESMFold 的核心是使用进化缩放模型(Evolutionary Scale Modeling,ESM)进行蛋白质结构预测。
核心技术
ESMFold 的关键技术基础是 Meta AI 之前开发的 ESM 系列语言模型,这些模型专门用于解析和理解蛋白质序列。ESMFold 利用了这些语言模型的功能来预测蛋白质的空间结构,这种方法展现了在蛋白质结构预测任务上的强大能力。
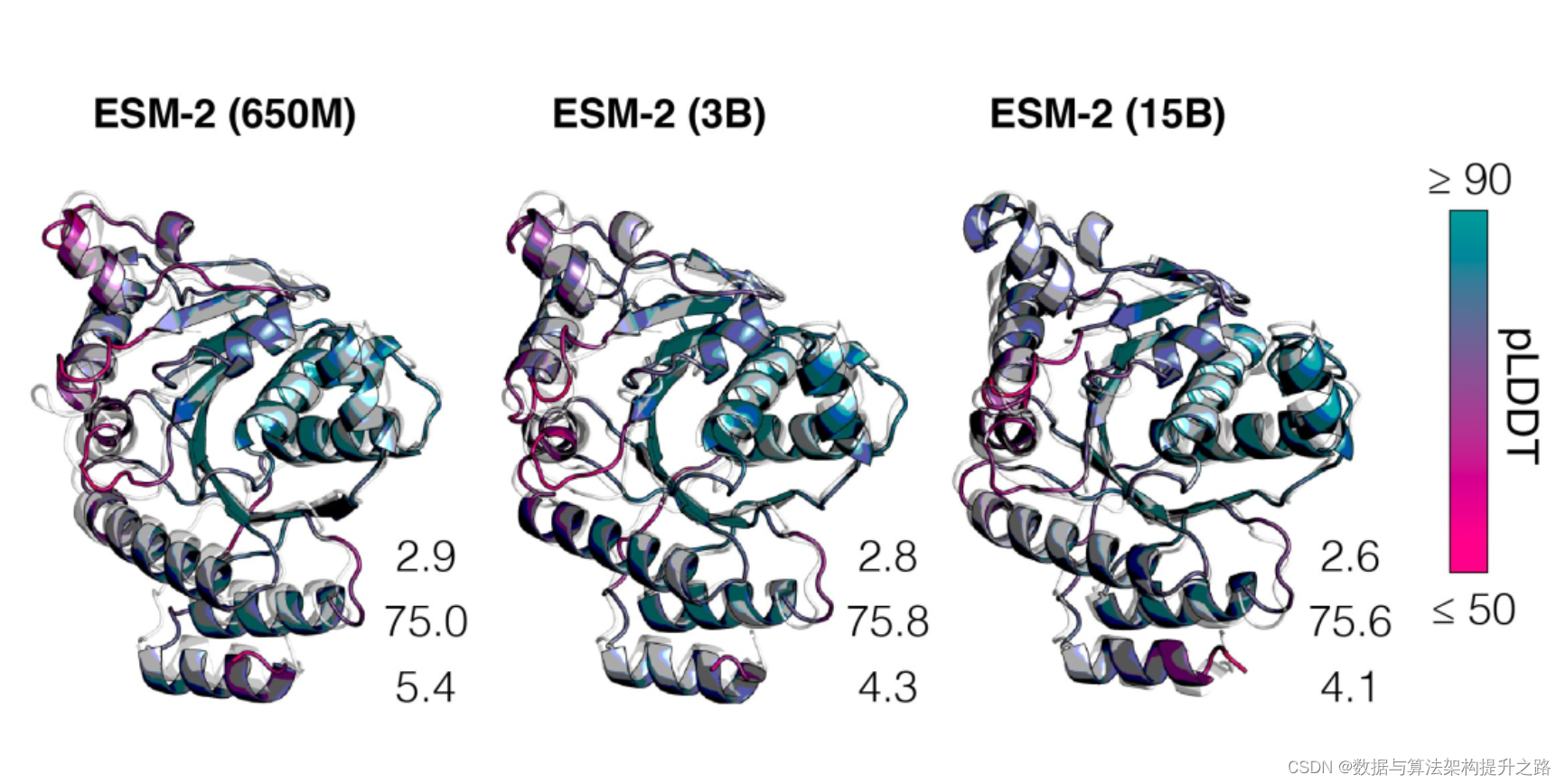
结构预测的准确性与语言模型的复杂度息息相关,也就是说,当语言模型能更好地理解序列时,便可以更好地理解结构
开源地址
ESMFold on GitHub
相关文章
ESMFold: AlphaFold2之后蛋白质结构预测的新突破_esm-2-CSDN博客