初看题目好像我在教你怎么导入数据,不不不,我是在教你正确的导入数据,不是说数据导入R就叫正确导入数据了。本章为新手教程,老手可以跳过。
这个内容早就想写了,今天有点空和大家聊一下。为什么R语言对于新手而言不太友好,有些人觉得R很难学。很多原因都有,我今天来说下没有正确导入数据的报错,比如下面报错
Error in xtfrm.data.frame(x) : cannot xtfrm data frames
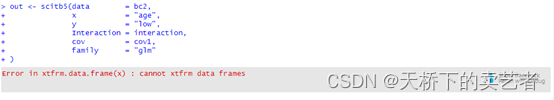
这对新手来说绝对是个大打击,你根本没法找到原因,会影响及的积极性和学R的信心,因为我就是这么走过来的。
因为R语言是个注重格式的语言,目前导入数据的R包种类繁多,有些R包存在格式转换问题,所以导入数据后分析容易出现各种各样的问题。我以我一个粉丝发给我的数据和代码为例子,就是以我写得scitb5函数来做个亚组分析得交互表,分析的内容详见这篇文章《scitb5函数2.1版本(交互效应函数P for interaction)发布----用于一键生成交互效应表、森林图》
先导入R包和我写得函数
library(tidyverse)
setwd("E:/公众号文章2024年/新手第一课,从学导入数据开始/scitb5")
source("./helper/21scitb5.R")
用我文章示例得代码导入数据
d1 <- read.csv("zaochan.csv", sep = ",", header = TRUE)
设置协变量和交互变量,
cov1 <- c("lwt", "smoke", "ptl", "ui", "ftv", "race")
interaction <- c("race", "smoke", "ui")
转换数据
bc1 <- d1 %>%
mutate(
race = case_match(race,
"black" ~ 1,
"white" ~ 2,
"other" ~ 3
)
) %>%
mutate(smoke = if_else(smoke == "nonsmoker", 0, 1)) %>%
mutate(across(c(race, low, ht, ui), factor))
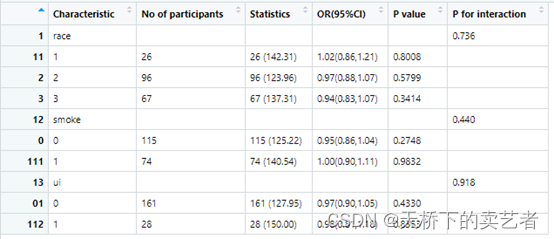
最后使用我的sicitb5函数生成表,似乎一切很顺利,得出结果。
out <- scitb5(data = bc1,
x = "age",
y = "low",
Interaction = interaction,
cov = cov1,
family = "glm"
)
下面咱们用readr包导入数据再跑一次
library(readr)
d2 <- readr::read_csv("zaochan.csv")
好像几乎一样,没什么问题,但是还是不一样的
bc2 <- d2 %>%
mutate(
race = case_match(race,
"black" ~ 1,
"white" ~ 2,
"other" ~ 3
)
) %>%
mutate(smoke = if_else(smoke == "nonsmoker", 0, 1)) %>%
mutate(across(c(race, low, ht, ui), factor))
生成结果
out <- scitb5(data = bc2,
x = "age",
y = "low",
Interaction = interaction,
cov = cov1,
family = "glm"
)
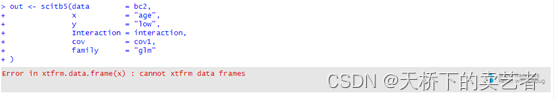
同样的代码,换个导入数据方法就跑不出来了,而且这个报错你根本看不出来是什么回事,没办法修改。
有人可能会说这是我scitb5函数的bug,但其实不是是这句很普通代码的报错,一句涉及数据转换的代码,这句代码应该很常用,涉及很多场景,很多R包都会用
length(levels(factor(bc2[,"low"]))) == 2

还有这句很普通的代码也会经常报错
d2<-rbind(d2,d1)
也会有人会说这只是个小概率事件,但是这样的例子很多,两三页都翻不完
这只是一个小例子,还有其他很多千奇百怪的报错。
这样类似容易出错的R包还有readxl包,haven包等因为这些包可以直接导入excel文件,方便是方便了,但是有时会容易出现莫名其妙的错误。不是说这些包没有用,还是有很大用处的(比如在一些特殊的场景),但是对新手来说,稳定不容易出错才是他们目前最需要的。
因此正确的常规导入数据方法是很重要的
我推荐两种方法导入数据,这些都是我长期实践觉得比较稳定,不容易出错的,
第一:就是如果你是excel数据:
我们点:文件----另存为----csv格式
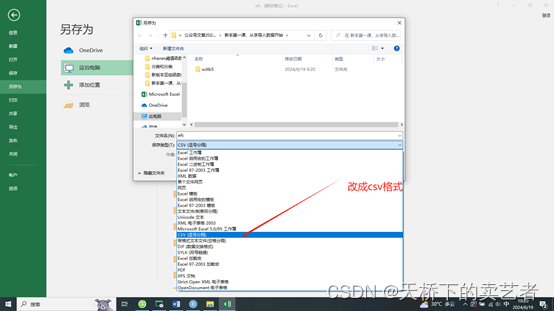
然后用R自带的格式导入,示例如下
bc<-read.csv("E:/r/test/nahnesme.csv",sep=',',header=TRUE)
第二:如果是SPSS数据,咱们可以使用foreign包,如我乳腺癌的例子,to.data.frame=T这个一定要有,因为这句等于把数据转成数据框。
library(foreign)
bc <- read.spss("E:/r/Breast cancer survival agec.sav",
use.value.labels=F, to.data.frame=T)
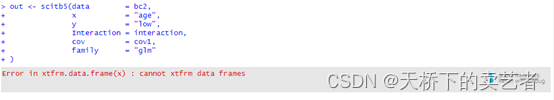
上面这个报错在部分情况使用下面代码把数据强制转成数据框格式有一定几率也可以解决问题
d2<-as.data.frame(d2)
但是对于新手来说,良好的数据导入习惯是成长很好的帮助。
其他数据以此参考,本期结束。
下面是个视频介绍
新手学R第一课,从学正确导入数据开始
最后和大家汇报一下,目前nhanes阈值效应函数已经基本写好,目前正在测试中,估计过段时间就可以和大家见面了。