多目标跟踪 距离的可视化(有动图)
flyfish
马氏距离的计算涉及到协方差矩阵的逆,而协方差矩阵的特征值和特征向量决定了数据分布的形状。椭圆的中心是数据的均值向量,椭圆的形状和方向由协方差矩阵的特征向量和特征值决定。椭圆的长轴和短轴长度与马氏距离的值相关联,较大的马氏距离对应于较大的椭圆。

马氏距离
马氏距离是一种度量,表示一个点到数据集中心(平均值)的距离,同时考虑了数据集的形状和分布。这种距离考虑了不同方向上的方差,因此它比欧几里得距离更适合高维数据的分析。
为什么使用马氏距离?
在多维空间中,数据点分布的形状可能不是圆形,可能是椭圆形。马氏距离可以考虑数据的这种形状差异,从而提供更准确的距离度量。
椭圆的意义
在二维空间中,马氏距离等于某个常数的点形成一个椭圆。这个椭圆展示了所有与中心点(平均值)具有相同马氏距离的点。因此:
-
椭圆中心 :表示数据的平均值。
-
椭圆形状和方向 :反映数据在各个方向上的方差和协方差。例如,数据在某个方向上的方差越大,椭圆在那个方向上就越长。
结合起来看
当我们在动画中移动一个点时,每个位置上这个点与数据集其他点的马氏距离会变化。这个变化用颜色来表示。与此同时,图中显示的椭圆展示了与数据集中心点(平均值)具有相同马氏距离的点的集合。
动画展示了:
- 给定点的马氏距离变化:通过点的颜色反映。
- 数据分布形状 :通过椭圆反映。
- 红点的位置以及它的马氏距离。
- 数据点的散布情况。
- 椭圆表示的数据的分布形状和方向。
通过展示椭圆,我们可以更直观地理解马氏距离是如何根据数据分布的形状和方向变化的。
分析为什么是椭圆的原因
在多维空间中,数据点的分布形状可能不是圆形而是椭圆形,这主要是因为数据在不同维度上的方差不同。为了更好地理解这一点,让我们逐步解释其中的原因。
方差与协方差
-
方差 :表示数据在某一维度上的离散程度。方差越大,数据在该维度上分布得越广。
-
协方差 :表示两个维度之间的关系。如果协方差为正,表示两个维度在相同方向上变动;如果为负,表示两个维度在相反方向上变动。
数据分布形状
当我们有多维数据时,每个维度的方差可能不同,这意味着数据在某些维度上分布得更广,在某些维度上分布得更窄。此外,维度之间的协方差决定了数据在多维空间中的方向性分布。
马氏距离的计算
马氏距离计算时会考虑数据的协方差矩阵(包含方差和协方差信息)。协方差矩阵的逆矩阵用于衡量数据点与数据中心之间的距离。这种距离度量方式会自动考虑各维度的方差和维度之间的相关性。
椭圆形状
假设我们有二维数据,分别在 x x x 和 y y y 方向上:
-
如果数据在 x x x 和 y y y 方向上的方差相同,并且两个方向上没有相关性(协方差为零),则数据分布呈现圆形。
-
如果数据在 x x x 和 y y y 方向上的方差不同,则数据分布在方差较大的方向上更广,呈现椭圆形。
-
如果 x x x 和 y y y 之间有相关性(协方差不为零),则椭圆会倾斜,表示两个维度之间的关系。
上面动图的代码
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from scipy.spatial.distance import mahalanobis
from scipy.stats import chi2
import imageio
# 生成示例数据
np.random.seed(42)
mean = [0, 0]
cov = [[1, 0.5], [0.5, 1]]
data = np.random.multivariate_normal(mean, cov, 500)
# 计算马氏距离的函数
def calculate_mahalanobis_distance(data, point):
cov_matrix = np.cov(data, rowvar=False)
inv_cov_matrix = np.linalg.inv(cov_matrix)
distances = [mahalanobis(d, point, inv_cov_matrix) for d in data]
return distances
# 绘制马氏距离等高线
def plot_mahalanobis_ellipse(data, ax, color='blue'):
cov_matrix = np.cov(data, rowvar=False)
mean_data = np.mean(data, axis=0)
# 特征值和特征向量
eigvals, eigvecs = np.linalg.eigh(cov_matrix)
# 长轴和短轴
order = eigvals.argsort()[::-1]
eigvals = eigvals[order]
eigvecs = eigvecs[:, order]
# 计算角度
angle = np.degrees(np.arctan2(*eigvecs[:,0][::-1]))
# 计算椭圆半径
chi2_val = chi2.ppf(0.95, 2)
width, height = 2 * np.sqrt(eigvals * chi2_val)
# 绘制椭圆
from matplotlib.patches import Ellipse
ellipse = Ellipse(mean_data, width=width, height=height, angle=angle, edgecolor=color, fc='None', lw=2)
ax.add_patch(ellipse)
# 绘制散点图并添加马氏距离和椭圆
def plot_mahalanobis(data, point, frame_number):
fig, ax = plt.subplots(figsize=(10, 8))
# 计算马氏距离并设置颜色映射
distances = calculate_mahalanobis_distance(data, point)
scatter = sns.scatterplot(x=data[:, 0], y=data[:, 1], hue=distances, palette='coolwarm', ax=ax, s=50, edgecolor=None)
# 调整颜色条(colorbar)的范围和标签
norm = plt.Normalize(vmin=min(distances), vmax=max(distances))
sm = plt.cm.ScalarMappable(cmap="coolwarm", norm=norm)
sm.set_array([])
fig.colorbar(sm, ax=ax, label='Mahalanobis Distance')
# 绘制给定点和椭圆
plt.scatter(point[0], point[1], color='red', s=100)
plot_mahalanobis_ellipse(data, ax)
plt.title(f'Mahalanobis Distance at Frame {frame_number}')
plt.xlabel('X')
plt.ylabel('Y')
plt.savefig(f'frame_{frame_number}.png')
plt.close()
# 定义动画点
points = np.linspace(-3, 3, 30)
frames = []
# 生成每一帧图像
for i, p in enumerate(points):
plot_mahalanobis(data, [p, p], i)
frames.append(imageio.imread(f'frame_{i}.png'))
# 保存为GIF
imageio.mimsave('mahalanobis_distance.gif', frames, fps=2, loop=0)
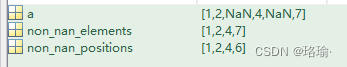
下面三种情况
方差相同且无相关性的数据,这种情况下,数据分布呈现圆形。
方差不同且无相关性的数据,这种情况下,数据分布在方差较大的方向上更广,呈现椭圆形。
方差相同且有相关性的数据,这种情况下,椭圆会倾斜,表示两个维度之间的关系。

import numpy as np
import matplotlib.pyplot as plt
from matplotlib.patches import Ellipse
import seaborn as sns
from scipy.stats import chi2
def plot_ellipse(mean, cov, ax, color='blue'):
# 特征值和特征向量
eigvals, eigvecs = np.linalg.eigh(cov)
# 长轴和短轴
order = eigvals.argsort()[::-1]
eigvals = eigvals[order]
eigvecs = eigvecs[:, order]
# 计算角度
angle = np.degrees(np.arctan2(*eigvecs[:,0][::-1]))
# 计算椭圆半径
chi2_val = chi2.ppf(0.95, 2)
width, height = 2 * np.sqrt(eigvals * chi2_val)
# 绘制椭圆
ellipse = Ellipse(mean, width=width, height=height, angle=angle, edgecolor=color, fc='None', lw=2)
ax.add_patch(ellipse)
# 生成数据
np.random.seed(42)
# 情况 1:方差相同,无相关性
mean1 = [0, 0]
cov1 = [[1, 0], [0, 1]]
data1 = np.random.multivariate_normal(mean1, cov1, 500)
# 情况 2:方差不同,无相关性
mean2 = [0, 0]
cov2 = [[3, 0], [0, 1]]
data2 = np.random.multivariate_normal(mean2, cov2, 500)
# 情况 3:方差相同,有相关性
mean3 = [0, 0]
cov3 = [[1, 0.8], [0.8, 1]]
data3 = np.random.multivariate_normal(mean3, cov3, 500)
# 绘制图形
fig, axs = plt.subplots(1, 3, figsize=(18, 6))
# 情况 1
axs[0].scatter(data1[:, 0], data1[:, 1], alpha=0.6)
plot_ellipse(mean1, cov1, axs[0])
axs[0].set_title('Variance same, No Correlation')
axs[0].set_xlim(-5, 5)
axs[0].set_ylim(-5, 5)
# 情况 2
axs[1].scatter(data2[:, 0], data2[:, 1], alpha=0.6)
plot_ellipse(mean2, cov2, axs[1])
axs[1].set_title('Variance different, No Correlation')
axs[1].set_xlim(-10, 10)
axs[1].set_ylim(-5, 5)
# 情况 3
axs[2].scatter(data3[:, 0], data3[:, 1], alpha=0.6)
plot_ellipse(mean3, cov3, axs[2])
axs[2].set_title('Variance same, Correlation')
axs[2].set_xlim(-5, 5)
axs[2].set_ylim(-5, 5)
plt.tight_layout()
plt.show()