Introduction
分享一些R进阶使用的技巧,相当于是之前写的R语言学习的实践和总结了。
-
Online slide: https://asa-blog.netlify.app/R_tips_for_advanced_use_byAsa/R_tips.html
-
下载slide和相关的各种test文件: https://asa-blog.netlify.app/R_tips_for_advanced_use_byAsa/R_tips_for_advanced_use_byAsa.zip
-
视频教程: https://www.bilibili.com/video/BV1eg4y1w7AL/
-
*才疏学浅,仅代表本人学习经验,R是一个宝库,此处只是冰山一角
Use R
R语言简介
R语言是S语言的一个变种。S语言由Rick Becker等人在贝尔实验室开发(著名的C语言、Unix系统开发实验室)。
R是一个自由源代码软件,GPL授权,于1997年发布,实现了与S语言基本相同的功能和统计功能。 现在由R核心团队开发,但全世界的用户都可以贡献软件包。
R的网站: http://www.r-project.org/
特点:
- 自由软件,免费、开放源代码,支持各个主要计算机系统;
- 具有完善的数据类型,代码简洁、可读;
- 强调交互式数据分析,支持复杂算法描述,图形功能强;
- 统计科研工作者广泛使用R进行计算和发表算法。R有上万扩展包(截止2023年7月5日在R包网站CRAN上有19784个)。
S语言作者,R语言专家J. M. Chambers指出R的.red[本质特征]:
- R中所有的存在都是对象(object);
- R中发生的动作都是函数调用(function call)。
详细地说R有如下技术特点:
- 函数编程(functional programming)。
- 支持对象类和类方法,面向对象的程序设计。
- 动态类型语言,解释执行,速度较慢。
- 数据框(data.frame)是基本数据类型,类似于数据库的表。
- 可以用作C和C++、FORTRAN语言编写的算法库的接口。
- 内部数值算法采用已广泛测试和采纳的算法实现,如排序、线性代数等。
R & Rstudio
- R是一种统计学编程语言
- Rstudio是R语言最好的IDE(Integrated development environment)
2022年RStudio公司正式改名Posit,拥抱大数据科学生态:R、Python、Julia…
RStudio使用方法概要PDF下载:rstudio-ide.pdf
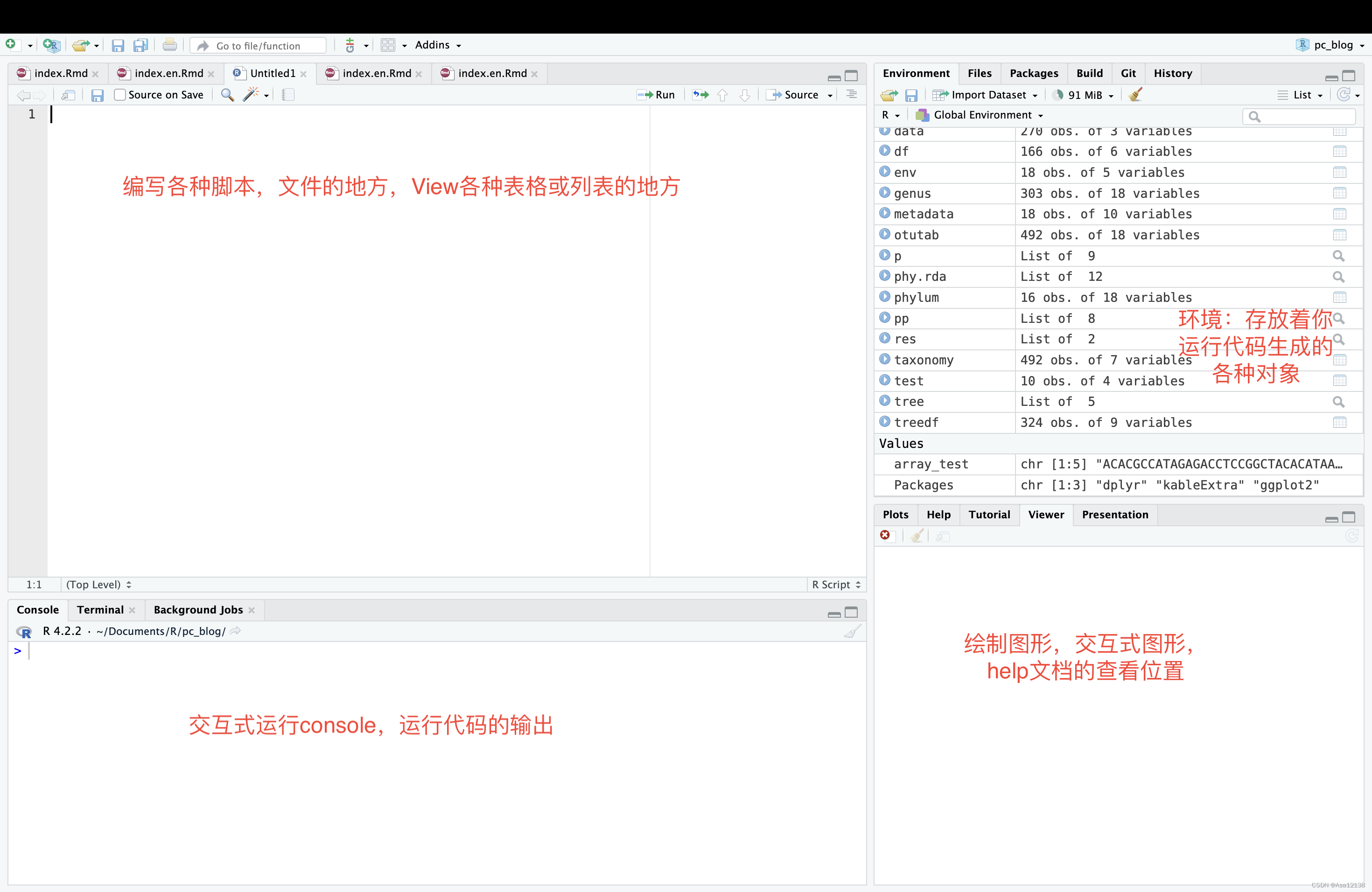
Rstuido的魔法:按住Ctrl(Command)单击某个变量名或函数名,如果是data.frame就可以进入预览窗格,
如果是函数就可以进入函数内部看具体的代码。
其实是调用了View()函数,对于不是以上类型的对象我们可以手动调用。
学习资料
-
全面教程
小白上手首推北大李老师的课程讲义《R语言教程》,中文讲义,简单易读,甚至整本书就是用Rbookdown编写的,电子书也比较好复制代码进行学习。《R语言实战》,也有中文版,介绍地更加全面且有不少进阶知识,值得一看!
然后是著名的英文书和参考手册:
- 《An Introduction to R》
- 《R Cookbook》
-
数据处理
tidyverse全家桶,其中包含ggplot2,tibble,dplyr,readr,stringr等常用的全面的数据处理包,能够很好的满足我们下游数据处理需求:
- 读取数据
- 清洗数据
- 转换数据
- 合并数据
- 筛选数据
- 可视化
官方教程:《R for Data Science》,非常建议熟读并使用
-
可视化
ggplot官方教程:《ggplot2: Elegant Graphics for Data Analysis》
为可视化而建的网站:From data to Viz | Find the graphic you need
基本使用
很多人使用 R,只为用其中的某个包的某个功能,只需学会以下几步即可:
1. 装好R & Rstudio ✅
2. 认识基本数据类型,结构
- 数值型常量
1,1e2,5L,Inf,-Inf; - 字符型常量
"str",'str'; - 逻辑型常量
TRUE,FALSE,T,F; - 缺失值
NaN,NA, 空值NULL; - 复数常量
2.2 + 3.5i; - 变量,区分大小写的字母、数字、下划线和句点组成变量名,支持但不建议中文;
- 向量(vector):用
c()函数把多个元素或向量组合成一个向量。如果元素基本类型不同,将统一转换成最复杂的一个,复杂程度从简单到复杂依次为: logical<integer<double<character。此外还可以有Date和factor类型的元素 - 列表(list):包装保存不同类型的数据。单个列表元素必须用两重方括号格式访问,单重方括号结果还是列表而不是列表元素。普通有names属性的列表可以用
$访问元素,S4对象可以用@访问元素。 - 矩阵,数组(matrix, array):矩阵用matrix函数定义,实际存储成一个向量,根据保存的行数和列数对应到矩阵的元素, 存储次序为按列存储。
- 数据框(data.frame):数据框类似于一个矩阵,有多个横行、多个纵列,各列允许有不同类型:数值型、因子、字符、日期时间,但同一列的数据类型相同。取子集应注意
drop。 - 改进数据框(tibble):tibble类型是一种改进的数据框,tidyverse全家桶推荐使用,确实有不少好处:
不会随意改变我们的数据名称,类型。打印优美。
3. 装好需要的包
包是R函数、数据、预编译代码以一种定义完善的格式组成的集合。
- 找到包,主要平台:
- CRAN 官方存储库,多个镜像
- BioConductor 用于生物信息学的开源软件专题库
- Github 开发版本,无限惊喜,但也可能缺少维护
- Bitbucket、SVN…
- 装在哪,本机位置:
计算机上存储包的目录称为库(library)。
函数.libPaths()能够显示库所在的位置,函数library()可以显示库中有哪些包。
.libPaths()
这个地址很重要,有时候会有多个(如Windows分区),我们要搞清楚装在哪里。
- 怎么装,例子:大部分情况下CRAN用
install.packages("pkg"),BioConductor用BiocManager::install("pkg"),Github用devtools::install_github("repos")即可。
但有时候会有依赖包相关的报错,我们可以手动下载特定版本包文件后安装。
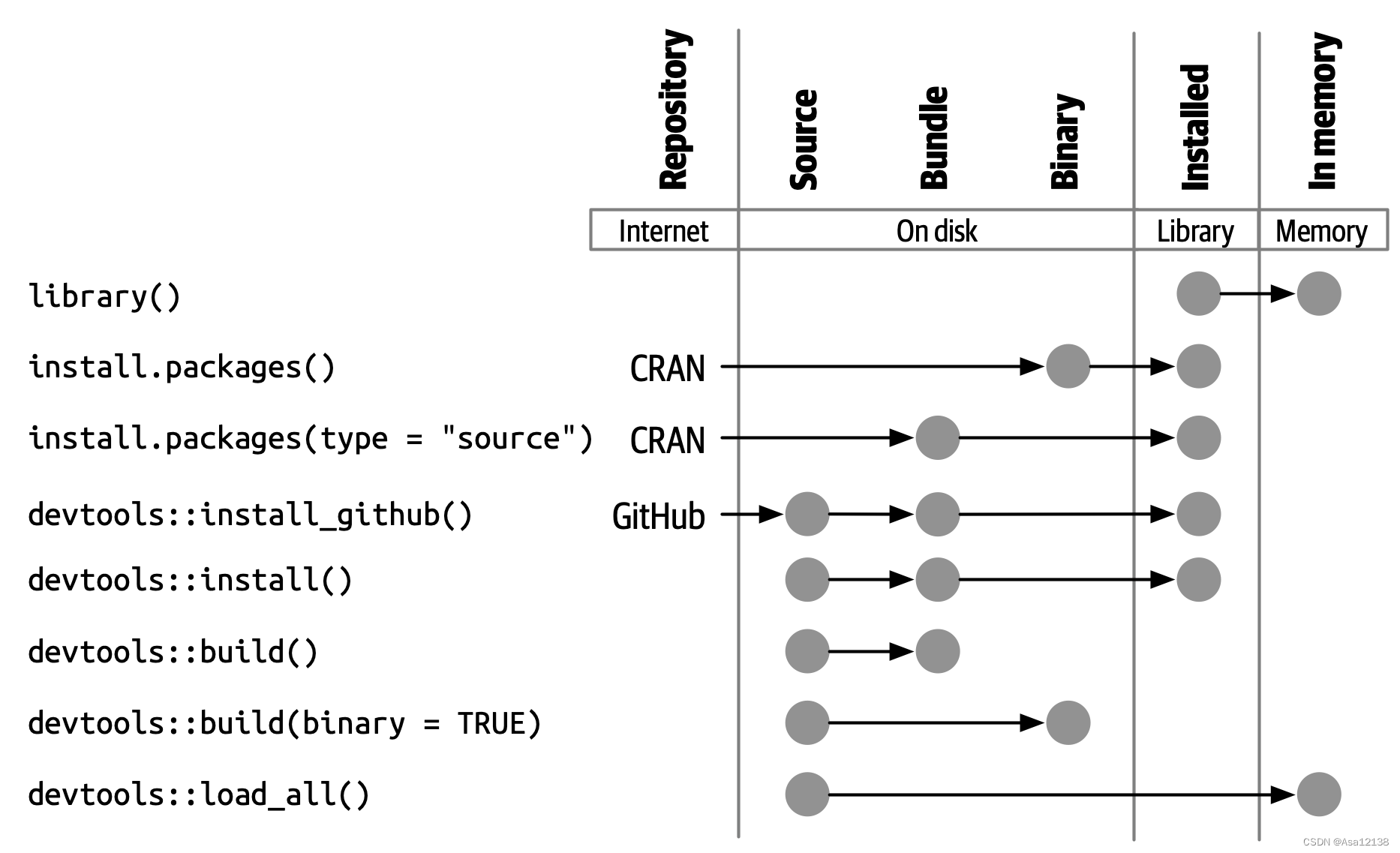
- source, 源码,一堆R脚本和文本文件,我们直接在github上看的就是这个。
- bundle, 捆绑态,其实就是打包过的source,一般.tar.gz结尾。
- binary, 二进制版本,架构不一样,.zip(Windows)或.tgz(macOS)结尾。
所谓安装包就是把source变成binary在放在我们的.libPaths()文件夹下。
还有问题,谷歌大法,或github问包作者。
带C++的R包出问题会麻烦一些,需要考察自己本机的C语言编译器。
- 怎么用包
首先可以用library(pkg)或require(pkg)将包导入内存,这样便可以调用其中的函数。
search()函数可以查看我们已经library的包。
library(pcutils)
search()
if(!require("pcutils")) install.packages("pcutils")
使用pkg::加上tab键可以列出某个包里所有导出的函数,多用help(pkg::func)或?pkg::func查看某个函数的用法。
如果不想用某个包了,可以用detach从当前环境移除(不是删除包)
detach("package:pcutils")
4. 正确数据/图像读入和写出
各种表格读取:read.table(), read.csv(), read.delim()
read.table("test.txt") #无法读取
当我们的表格含有 ", ', # 或者列名中含有任何非变量名字符时都要小心
read.table("test.txt",header = T,sep = "\t",comment.char = "",quote = "",check.names = F)
建议在readr包的支持下用read_csv(), read_table2(), read_delim()等函数读入。
保存为tibble类型,快得多,也不自动将字符型列转换成因子,不自动修改变量名为合法变量名。
readr::read_delim("test.txt",delim = "\t")
excel用readxl包,剪切板可用datapasta包或clipr::read_clip()。表格写出用write_*(),与读入一致。
任意文件访问,跟python很像,使用file()打开文件,close()关闭。
- r—文本型只读;
w—文本型只写;
a—文本型末尾添加; - rb—二进制只读;
wb—二进制只写;
ab—二进制末尾添加; - r+或r+b—读写;
w+或w+b—读写,清空文件;
a+或a+b—读和末尾添加。
或者可以用cat()直接输出任意文本到文件。
clipr::write_clip()可以把R数据框或向量写入剪切板,可以直接粘贴他处。
图片保存:
pdf("new.pdf") #png(),jpg(),tiff(),bmp()
#画一些图
dev.off() #要记得关闭设备
ggplot2::ggsave("new.pdf",plot = p,device = "pdf")
#p是一个ggplot对象,device可选各种文件格式
R变量储存:
R的save()命令可以将一个或者多个R变量保存到文件(.rda或.RData)中,保存结果是经过压缩的,在不同的R运行环境中兼容。
使用load()可以恢复保存的变量。
强烈建议传递数据时用这种文件,避免了数据写入和读取时引起的差异。
使用save.image()可以把整个环境的所有变量保存,方便结果复现。
保存单变量时推荐使用saveRDS(),用readRDS()载入并返回变量,这样可以将某个变量赋值给某个拟定的变量名,避免某个变量值变了我们还不知道, 如
saveRDS(old,file = "tmp.RDS")
new=readRDS("tmp.RDS")
5. 关闭R走人 ✅
R programming
程序控制结构
分支结构
x=1
if(x>0) {
print("x > 0")
} else {
print("x <= 0")
}
#或者用switch函数
switch (x>0,
T = {print("x > 0")},
F = {print("x <= 0")}
)
#或者用ifelse函数,向量式
x <- c(-2, 0, 1)
ifelse(x >=0, 1, 0)
循环结构
for (i in 1:3) {
print(i)
}
#或者用while函数
i=1
while (i<4) {
print(i)
i=i+1
}
#或者用repeat函数
i=1
repeat{
print(i)
i=i+1
if(i>3)break
}
注意用到的判断条件必须是逻辑标量值,不能为NA或零长度。
这是R编程时比较容易出错的地方。
循环结构进阶,apply()家族
上述显式循环是R运行速度较慢的部分,有循环的程序也比较冗长,建议使用apply, sapply, lapply, vapply, tapply等函数替代。
func=function(i)print(i)
a=lapply(1:3, func)
另外,建议循环过程不要制作副本:
类似于x <- c(x, y)这样的累积结果每次运行都会制作一个x的副本, 在x存储量较大或者重复修改次数很多时会减慢程序。
而是应该在循环前设置好用来保存结果的数据结构:
x=numeric(100)
x[[i]]={i循环100次产生的结果}
管道
原生管道,R从4.1.0版本开始提供了一个|>运算符实现管道,如计算
e
2
e^{\sqrt{2}}
e2:
2 |> sqrt() |> exp()
但是我还是更喜欢用magrittr提供的%>%,支持更复杂的应用,比如用.代表产生的中间变量放到合适的参数位置:
library(magrittr)
"hhh"%>%paste0("prefix_",.)
还有%<>%等方便的变体:
a="hhh"; a%<>%paste0("prefix_",.)
a
函数
使用自定义函数, 优点是代码复用、模块化设计。
函数名 <- function(形式参数表) 函数体
特殊形参:...
my_mean=function(...)sum(...)/length(c(...))
my_mean(1,2,3,4)
无名函数: 不定义函数名也可以调用
a=lapply(1:3, \(i)print(i))
变量作用域:全局变量 vs 局部变量
x=100
func=function(x){
print(x+1)
}
func(0)
- R中所有的存在都是对象(object)
对象其实就是一种封装的概念,它把事物封装成一个类(class),然后提供类的方法(method),而具体的过程人们看不到。
class(mean)
- R中发生的动作都是函数调用(function call)???
函数调用有四种方式:
1 前缀形式。
这也是一般的格式
mean(1:2)
2 中缀形式。二元运算符实际上都是函数
1+1
`+`(1,1)
`%love%`=function(a,b)paste0(a," loves ",b)
"Romeo"%love%"Juliet"
3 替换形式。
x <- 1:2
names(x) <- c("a", "b")
x
print(`names<-`)
4 特殊形式。x[1], x[[1]]这些取子集或元素等
x <- 1:5
x[1]
`[`(x, 1)
确实,R中发生的动作都是函数调用(function call),知晓函数调用的形式,可以让我们在用某些包的函数时明确问题来源,查找源码等。
Debug
某个函数突然失灵了
- 工作环境变化
环境是R语言比较困难的概念,一般不需要深入了解也能用。
但我们要知道,大多数时候我们处于Global Environment,我们敲出来的每一个变量名(函数名)都是优先在全局环境找,找不到的话就search()里面按顺序去每个环境找。
所以要注意环境优先级以及变量名冲突,极端情况如下:
1+1 #正常当然等于2
#定义了`+`,或者新导入的包包含一个`+`的函数名,覆盖了base的加法
`+`=`-`
1+1 #结果会变成0
常使用pkg::func的形式调用函数是个好习惯,可以防止不同包的同名函数冲突。
- 断点,
traceback(),debug(),browser()
反向追踪(traceback)将错误定位到某一函数调用。
可以用debug(func)命令对函数func开启跟踪运行。
用undebug(func)取消对func的这种操作。
- 我要debug的函数在哪?
一般来说我们可以找到报错的代码行以及具体的函数,便可以使用debug()。
但有时候会发现一些函数无法被正确定位,具体代码也只能看到UseMethod():其实是我们碰到了泛型函数。下面同样一个summary函数,为啥对women和fit做出了完全不同的处理?
summary(women)
fit <- lm(weight ~ height, data=women)
summary(fit)
summary(women)
fit <- lm(weight ~ height, data=women)
summary(fit)
class(women); class(fit)
原来他们是不同的类(class),泛型函数会更具对象的类选择运行什么样的代码(即见人说人话,见鬼说鬼话):
调用func.class()函数,上述就是分别调用了summary.data.frame(),summary.lm()。print和plot也是非常常见的泛型函数。
或者我们可能发现某些报错函数not found(但我们又确定有这样一个函数):
其实是我们碰到了非导出函数(R包开发者不想用户看到所有的函数,只把有用的导出)。
library(reshape2)
class(mtcars)
#melt是泛型函数,那应该调用melt.data.frame
melt.data.frame(mtcars,id.vars = "cyl")
## Error in melt.data.frame(mtcars, id.vars = "cyl") :
## could not find function "melt.data.frame"
找到这些函数的方法也很简单(毕竟R包需要都开源):pkg:::func即可
reshape2:::melt.data.frame(mtcars,id.vars = "cyl")
有时候我们通过debug可以发现一些有用的内部函数(比如从那种一步/很久完成XX分析的函数中找到我们需要的功能),
这样我们便可以使用pkg:::func做想做的部分。
代码整理
我们在做数据分析时会产生大量代码,做好整理非常重要。
-
最开始,每一个独立的R脚本进行分析 (代码冗余度太高,后期很难快速看懂)
-
学会编写函数,复制函数到不同的R脚本使用(n个地方的函数需要修改n次)
-
将常用的函数放在一个R脚本下,要用的时候
source()整个脚本(全局环境中出现大量函数名,可能还会引起冲突) -
将常用函数整理成R包,写好注释和帮助文档,方便调用和分享 (嘎嘎香)
接下来的3,4部分非必需,但我觉得学习后便会有惊喜:
R markdown (to everything)
R Markdown是一种文本格式,用于创建具有代码、文本、图形和输出结果的可重复性报告。
是“文学式编程”(literate programming, (Knuth 1984))思想的实践。
它是在R语言环境中开发的,也可以与其他编程语言(如Python,shell)结合使用。
优点:
- 可重复性:代码和结果都是可重复的,有助于确保研究结果的可靠性和透明度。
- 效率:使用R Markdown在单个文件中组织和记录所有内容,节省时间和精力。
- 灵活性:R Markdown支持多种格式和输出选项,根据需要自定义样式布局。
总之,比word好用,比Latex好学😂
Yihui Xie大佬的官方文档:R Markdown: The Definitive Guide (bookdown.org)
以及参考书:R Markdown Cookbook (bookdown.org)
To Documents
Rmd文档头部是YAML元数据,可指定各种输出参数
-
HTML
-
PDF
-
Word
优点:
-
公式,化学分子式非常好写
-
文献插入也不错(配合Zotero)
-
图表自动编号和引用、链接(我觉得比word好用点)
-
支持交互式图形(HTML)
To Presentation
R Markdown支持生成各种幻灯片文件(此幻灯片也是由Rmd生成的😂)
-
PowerPoint
常用,易分享
-
ioslide/Slidy
网页格式的幻灯片,支持交互 (拜托,PPT可交互真的很酷)
-
Beamer
PDF幻灯片,模版众多,大学课堂热衷使用
To Research Papers
rticle包,包含一套自定义 R Markdown 格式和模板,用于创作期刊文章和会议提交。
例子:
-
Frontiers
-
Bioinformatics
To Website
Rmarkdown可以帮助我们搭建简易网站:
-
index.Rmd是主页内容, 可以在此处人工加入其它页面链接。
-
_site.yml是一个YAML文件, 其中包含站点的设定和输出设定。
例子:
File -> New Project -> New directory -> Simple R Markdown Website ->
Create Project (click Open in new session) -> Build -> Build Website
To Blog
R扩展包blogdown可以与Hugo软件配合制作简单的静态博客网站。
网站的所有文件都在一个目录中,只要上传到任意的网站服务器就可以发布,没有任何限制。
我的博客:Asa’s blog
参考书:blogdown: Creating Websites with R Markdown (bookdown.org)
To Book
R的bookdown扩展包
在bookdown的管理下:一本书的内容可以分解成多个Rmd文件, 其中可以有可执行的R代码, R代码生成的文字结果、表格、图形可以自动插入到生成的内容中, 表格和图形可以是浮动排版的。 输出格式主要支持gitbook格式的网页图书。
我也稍微尝试写了一下:MetaNet Tutorial
参考书:bookdown: Authoring Books and Technical Documents with R Markdown
R package/shiny
写R包
包是可重现 R 代码的基本单元,包括可重用的 R 函数、描述如何使用它们的文档和示例数据。
一个包将代码、数据、文档和测试捆绑在一起,并且很容易与他人共享。
简单例子(需要下载roxygen2包):
File -> New Project -> New directory -> R package ->
Create Project (click Open in new session)-> Package name -> Build -> Install
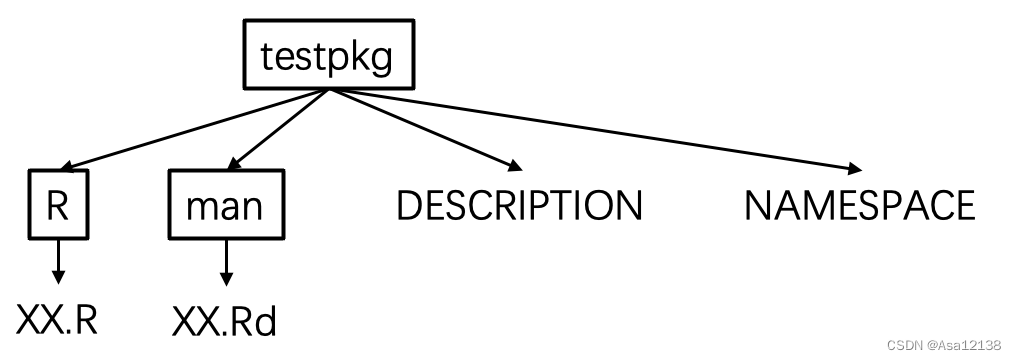
最简单的包只需要这些目录和文件即可,
- R目录下放在我们编写的R函数脚本
- man目录放着所有函数的帮助文档(
roxygenise()函数生成,不要自己写) - DECRIPTION定义了包的各种元数据,包名/版本/依赖包等等,非常重要
- NAMESPACE控制函数的可视性(
roxygenise()函数生成,不要修改!)
参考书:R Packages (2e) (r-pkgs.org)
如果真的有意开发R包,建议走devtools的流程,要比前面的简单例子好用。
library(devtools)
#创建R包
create_package("testpkg")
#配置git用于版本控制
use_git()
#创建一个R脚本,开始写函数
use_r("first")
#生成man文件和NAMESPACE
document("./")
#使所有函数可用(没有install但R包已在内存)
load_all("./")
#检查R包语法,建议每次重要修改后运行一次,解决完所有的Error,Warning,Note再进一步修改,不要积累较多错误
check("./")
#打包为bundle包,.tar.gz
build("./")
#install我们写好的R包
install("./")
#定义一些测试例子
use_testthat()
循环后面几步:function -> document -> load_all -> check ->install,直到功能完善,check无误。
- 函数应该功能明确,不能使用
library,而应该用pkg::func调用,并且这里的pkg一定要在DESCRIPTION中声明。 - 函数不应该产生不良副作用,比如修改用户环境变量,谨慎使用
option,par,setwd等函数。 - 函数的头文件很重要,包含了帮助文档以及决定函数性质的参数:
- 使用Shift+option+command+R可以帮助生成头文件
发布包:
-
CRAN submission 请仔细阅读CRAN Repository Policy,然后必须让代码通过
R CMD check再提交,不然会直接拒收。我们提交的包是bundle包,.tar.gz格式。过了机检后就会有“审包人”联系(一般是志愿者,我们应当态度好些),
再积极沟通修改代码就好,最后通过审核一天就能在CRAN上看到了。 -
BioConductor submission 用于生物信息学的开源软件专题库,上传前仔细阅读Guidelines,
明确包的主题要跟生物有相关性( Software, Experiment Data, Annotation and Workflow.),发布包的流程是Github issues,
审核过程全公开透明,你现在就能点进去看有哪些包正在提交。 -
Github 这里就最自由了,你可以随意上传在自己的仓库,也可以邀请别人直接从github安装。
制作Shiny APP
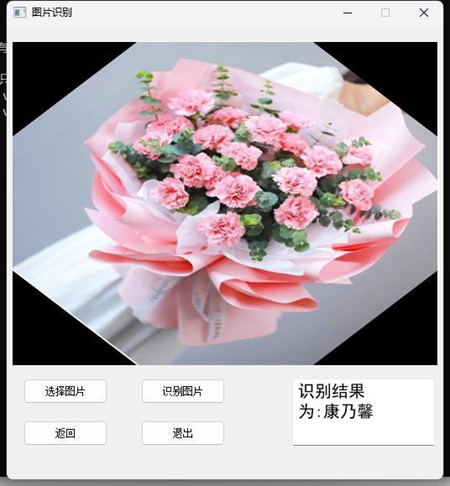
Shiny 是一个 R 包,可轻松地直接从 R 构建交互式 Web 应用程序。
可以在网页上托管独立应用程序或将它们嵌入 R Markdown 文档。还可以使用 CSS 主题、html 小部件和 JavaScript 操作扩展Shiny 应用程序。
例子:
File -> New Project -> New directory -> Shiny Application ->
Create Project (click Open in new session) -> Run App
shiny::runApp("test_shiny/app.R")
官方教程:Shiny Learning Resources
参考书:Mastering Shiny

关注公众号 ‘bio llbug’,获取最新推送。