一份环境样品中,可包含的DNA片段能达到上千万个,而每一个物种能携带的DNA又是独一无二的。将从样品中获得的遗传标记序列与DNA数据库中的序列相比对,从而确认分析的DNA来自什么物种,这就是环境DNA宏条形码技术(eDNA Metabarcoding)。该技术可基于物种在环境中释放的遗传物质(例如水体、土壤、粪便)进行检测,已成功地用于环境中不同类型生物物种检测研究分析。

利用Metabarcoding技术对环境样本中的环境DNA进行检测,可以获取环境样本中DNA所属物种的分类学信息和丰度信息。目前,eDNA研究方向主要包括:鱼类多样性分析、动物食性分析、濒危珍稀物种监测、入侵物种监测、生物多样性分析等,今天小编带大家把eDNA热门应用一网打尽!
案例1
鱼类多样性检测:eDNA揭示北京地区不同水境鱼类多样性分布特征[1]

图 北京采样点地图
本研究以北京为研究区域,对109个静水和动水水体开展eDNA的鱼类多样性调查,共鉴定到75种鱼类(52个本地原生鱼种和23个外来鱼种)。同时对水体进行水质理化指标测定,分析环境指标对鱼类多样性分布的影响。发现北京地区具有较高的鱼类多样性,但非本地鱼种占全部鱼类的30%。鱼类群落同质化较为明显,但不同水体类型间鱼类群落组成也存在一定差异。
-
采样方法
对流经北京的5个水系的49个静水点和60个流动点进行采样。静水点:等距离分布在岸边的3个位置各取1L水样; 流动点:相隔20米的3个位置各取1L水样,对同一流域进行多次采样,两次取样之间纵向距离超过3公里。
-
引物信息

案例2
濒危珍稀物种监测:时隔87年,被认为已灭绝的德温顿金鼹鼠重现南非[2]

环境 DNA (eDNA) 作为一种表征和监测生物多样性的新兴方法引起了全世界的关注,并且对于稀有、难以捉摸或濒临灭绝的物种变得尤为重要。本研究对南非西海岸进行广泛的 eDNA 采样,利用三对引物(mtDNA cyt b 、12S 和核 GHR)进行扩增,最终检测到四种金鼹鼠,包括三种已知物种和一种无法识别的物种。证明了eDNA技术存在巨大的机遇,可用于寻找其他消失或濒临灭绝的物种。
-
取样方法
每个采样点取15mL土壤
-
引物信息

案例3
入侵物种监测:eDNA揭示了北美五大湖入侵物种的遗传多样性和种群结构[3]
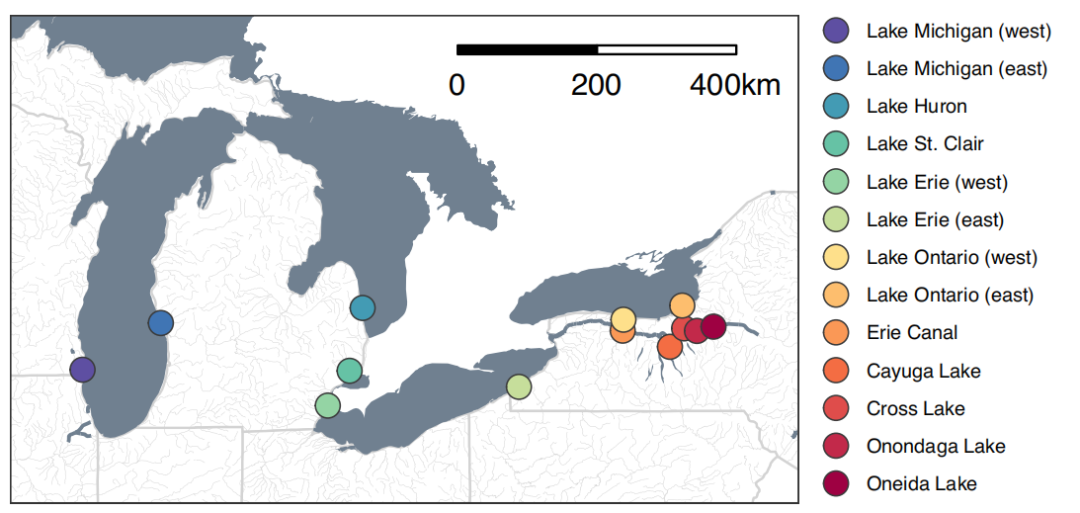
图 eDNA取样位置
eDNA通过分析生物体释放到环境中的DNA,用于收集各种生态系统中物种的信息。作为一种无创、高效的水生环境遗传物质取样方法。除了用于物种检测,eDNA还可以提供关于种群水平遗传变异的信息。一项发表在PNAS的研究对北美五大湖地区进行eDNA采样,分析了eDNA取样在鱼类物种的遗传多样性和结构方面的潜力,揭示了种群特征的潜力,提供了物种监测和管理计划信息,这些都是传统监测方法难以完成的。eDNA的研究进展为保护濒危和脆弱物种以及管理破坏性入侵物种开辟了新的可能性。
-
取样方法
在圆虾虎鱼(被监测的入侵对象)入侵范围内的14个地点分别采集2L地表水样品,装在无菌收集袋中,使用硝酸纤维素膜过滤(直径47 mm;1µm孔径)。
-
引物信息

案例4
食性分析:鲸类的监测与食性分析[4]
由于鲸类本身数量稀少、且其行为难以捉摸,加之传统研究方法的限制,其监测工作面临多重挑战,这极大阻碍了保护行动的开展。本研究利用eDNA技术对深圳大鹏湾的一头须鲸(昵称“小布”)进行种类鉴定。通过获取的4个线粒体基因序列片段,证实其属于布氏鲸近岸亚种(Balaenoptera edeni edeni)。利用eDNA和12S扩增子高通量测序,发现小布潜在的食物主要是沙丁鱼类和鳀鱼类。通过与邻近海域鱼类群落结构的对比,结合小布氏鲸的发现海域和时间,推断其很有可能是追踪这些洄游的鱼类进入大鹏湾。

-
取样方法
在小步出现的6个热点区域采集水样1L,采样点深度< 10 m,过醋酸纤维素微纤维膜(直径47 mm)后,保存在液氮中,在- 80℃下储存,直至提取eDNA。
-
引物信息

案例5
生物监测:空气eDNA捕获自然界陆地脊椎动物多样性[5]
现今的生物多样性和气候危机迫使我们开发更有效的陆地生态系统监测工具,本研究利用空气eDNA检测自然界中陆生脊椎动物群落。在丹麦的一个混交林,3天内收集143个空气eDNA样本,分析获得64种鸟类、哺乳动物、鱼类和两栖类群,其中57个“野生”类群代表了整个地区陆生脊椎动物的四分之一以上。这项研究证明了空气eDNA可用于陆地系统中脊椎动物的高分辨率生物监测。

-
取样方法
空气eDNA样本采集需要一个专门的被动采样设备,每个采样设备有2种不同的气体流速,共计4个样本收集器,共计6个样本采集点,每次样本收集的时间为12h,一天收集两次,早7点到晚7点和晚7点到第二天早7点分别代表白天和晚上,采集3天的样本。
-
引物信息
| 哺乳动物 16S引物 | 16Sman1 | 5’-CGGTTGGGGTGACCTCGGA-3’ |
| 16Sman2 | 5’-GCTGTTATCCCTAGGGTAACT-3’ | |
| 脊椎动物 12S引物 | 12SV05 forward | 5’-TTAGATACCCCACTATGC-3’ |
| 12SV05 reverse | 5’-TAGAACAGGCTCCTCTAG-3’ |
案例6
水生植物多样性调查:eDNA调查京津冀河流中水生植物的多样性及分布[6]
传统的调查方法(TSM)很难对河流中对水生植物多样性进行普查,随着水生植物多样性的不断下降,改进水生植物群落监测方法越来越重要。本研究通过与TSM对比,评价了eDNA对京津冀河流水生植物多样性和空间分布的估算效果。结合这两种方法,共鉴定到157种水生植物,其中包括24种水生植物、61种两栖植物和72种中间植物。eDNA对物种丰富度和丰度的空间格局与TSM结果一致,从山区向农业区再向城市区逐渐下降。与TSM相比,eDNA在每个位点鉴定的物种数量显著增加,并在水生植物中获得显著较高的丰度。本研究结果强调eDNA在评价水生植物多样性的潜力。
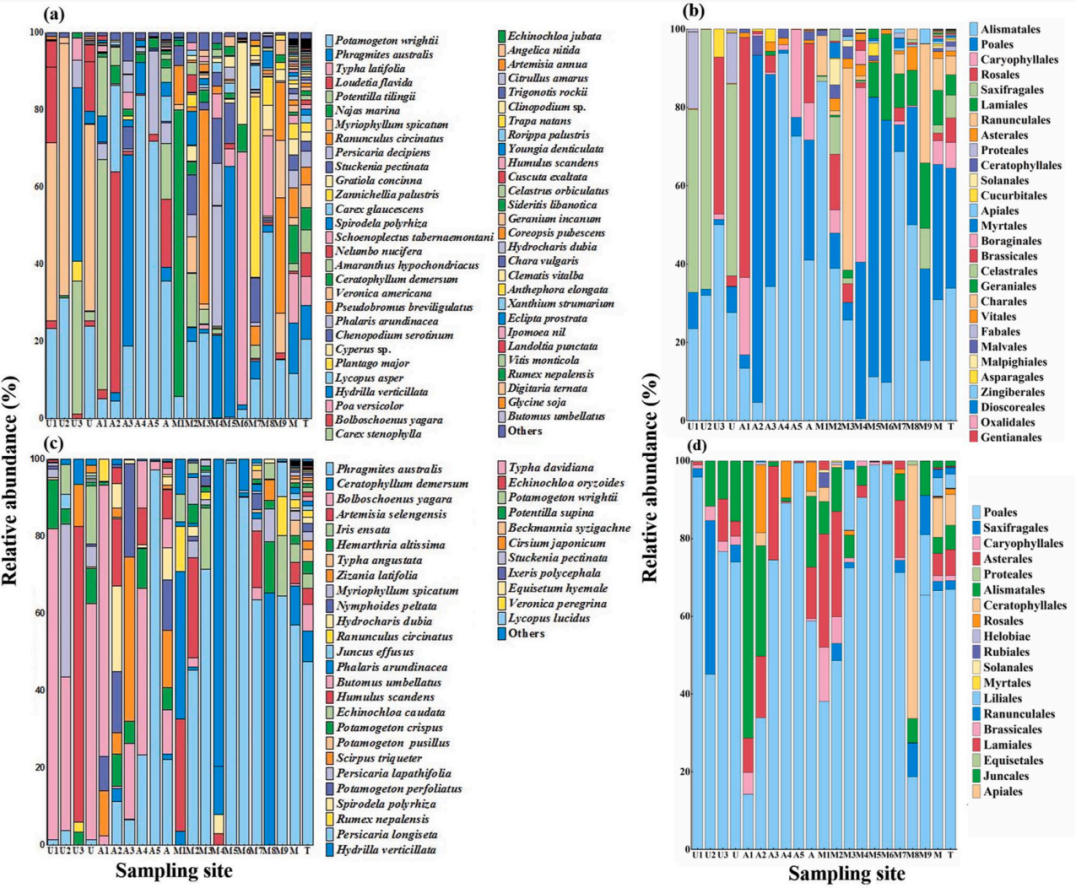
图 eDNA在物种水平(a, c)和目水平(b, d)上每个站点的水生植物物种相对丰度
-
取样方法
在每个地点,使用Peterson抓取器收集表层沉积物顶部0-1 cm的样本,沉积物样本立即放入5 mL无菌离心管中,并在液氮中保存,每个地点重复5次沉积物样本,用于eDNA的提取,在-80°C下保存直到提取。
-
引物信息
| RbclF | 5′-ATGTCACCACAAACAGAGACTAAAGC-3′ |
| RbclR | 5′-GTAAAATCAAGTCCACCRCG-3′ |
案例7
陆地植物多样性调查:现代丝绸之路东段植物物种多样性格局[7]
本研究沿着中国西北荒漠区现代丝绸之路东段进行了植物物种多样性调查,发现在这一区域温度比降水更能影响当地的植物物种多样性。此外,人类活动可能通过引进作物和入侵植物以及消除适应环境的本地植物来改变当地的植物物种多样性格局。研究证明了基于eDNA技术在植物物种多样性调查方面的潜力。

图 基于土壤表层样品的中国西北荒漠区种子植物整体情况
-
取样方法
从阿拉善地区沿河西走廊和新疆塔里木盆地采集表层土样144份。采用五点取样法,每个样方 (1,000 cm×1,000 cm) 采集5份表土样品等量混为一份,样品经65 ℃干燥24小时,以防止DNA的进一步降解,在-80°C下保存直到提取。
-
引物信息
| matK | matK472F | 5′-CCC RTY CAT CTG GAA ATC TTG GTT C-3 |
| matK1248R | 5′-GCT RTR ATA ATG AGA AAG ATT TCT GC-3′ | |
| rbcL | rbcLbF | 5′-AGACCTWTTTGAAGAAGGTTCWGT-3′ |
| rbcLbR | 5′-TCGGTYAGAGCRGGCATRTGCCA-3′ |
通过以上案例,相信大家已经掌握了eDNA的应用方向,那么,eDNA研究过程中一个核心问题就是:引物如何选择?
凌恩已总结好了植物、真菌、原生生物、古菌、细菌常用的eDNA引物(如下表格),以及COI、SSU rRNA、ITS引物的应用方向:
① COI基因可以用于区分不同物种和亚种,但不适合所有真核生物;
② SSU rRNA基因被广泛应用于真核生物分类学研究中,尤其是对于一些难以鉴别的物种;
③ ITS区域也被证明具有较高的进化变化率,可以作为鉴定物种的有效工具;
④ 与SSU rRNA基因相比,COI基因在分析真核微生物的遗传关系方面更有效。
表 eDNA引物列表[8]

此外,eDNA分析过程中比对的数据库也是另一个重要方面,针对地区性特有物种检测,凌恩生物推出了eDNA数据库构建服务,让物种检测更加准确!(2024,eDNA研究新亮点→数据库定制服务)
具体样本、引物以及数据库信息可以后台给我们留言或者联系当地销售哦!期待与您合作!

参考文献
[1]Environmental DNA captures native and non-native fish community variations across the lentic and lotic systems of a megacity.Science Advances, 2022.
[2] Environmental DNA from soil reveals the presence of a“lost”Afrotherian species. Biodiversity and Conservation,2023.
[3] Environmental DNA reveals the genetic diversity and population structure of an invasive species in the Laurentian Great Lakes.PNAS,2023.
[4] Assessing the potential use of environmental DNA for multifaceted genetic monitoring of cetaceans: Example of a wandering whale in a highly disturbed bay area.Ecological Indicators,2023
[5] Airborne environmental DNA captures terrestrial vertebrate diversity in nature.Molecular Ecology Resources,2023
[6] Estimating aquatic plant diversity and distribution in rivers from Jingjinji region, China, using environmental DNA metabarcoding and a traditional survey method. Environmental Research,2021.
[7] What determines plant species diversity along the Modern Silk Road in the east? iMeta,2023.
[8] DNA barcoding, an efective tool for species identifcation:a review.Molecular Biology Reports,2023.