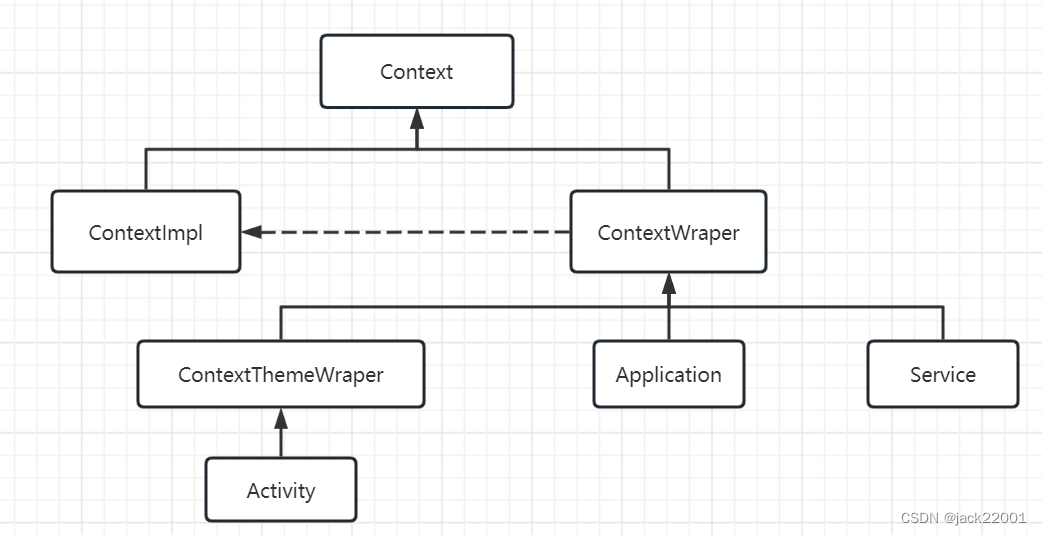
随着测序技术的不断发展,组学研究愈加深入。在微生态研究领域,受限于二代测序读长的扩增子测序技术,迎来了“解放式”全面发展优化的三代测序时代——PacBio全长扩增子测序的优势逐渐显现:凭借其超长读长,高准确率,高分辨率等优势,全长扩增子测序技术在环境保护,医学诊断,畜牧养殖,污水处理等领域发挥了重要作用。这里,为大家分享多篇基于PacBio三代测序平台开展的全长扩增子研究应用,让我们一起深入了解三代测序技术为微生态研究提供的新方法和新思路!
01、环境细菌研究
◆ 文献标题:Microdiversity and phylogeographic diversification of bacterioplankton in pelagic freshwater systems revealed through long-read amplicon sequencing长读扩增子测序分析揭示远洋淡水系统浮游细菌的微多样性和系统地理多样性
◆ 期刊:Microbiome
◆ 发表时间:2021年1月
◆ 内容摘要:
尽管淡水生态系统中栖息地是互不相连的,但淡水生态系统中栖息着世界性浮游细菌谱系。根据16S rRNA基因序列大于97%的相似性来进行谱系划分,但细菌谱系内多样性和系统地理学仍然没有得到解决,而这是理解这些菌谱背后普遍存在的生态进化过程的关键。本研究针对几乎全长的16S rRNA基因和相邻的核糖体内部转录间隔区序列进行了长读长扩增子测序,以揭示日本和欧洲11个深水湖泊中远洋浮游细菌组合的谱系内多样性。研究通过单核苷酸识别分析,且利用鸟枪法宏基因组测序进行验证,发现了11个主要细菌谱系中的每一个的7-10个扩增子序列变异,并提示 不能通过常规方法解决的共域、异地和时间微多样性。鉴定出具有相似谱系内群体组成的样本集群,这一直支持日本和欧洲之间的基因隔离。在区域尺度(长达数百公里),湖泊之间的扩散不太可能成为限制因素,而环境因素或遗传漂变是种群构成的潜在决定因素。不同谱系的微多样性程度不同,这表明高度多样化的谱系,谱系内多样性程度最低的是占优势的次生物种谱系(CL500-11),这表明它们在湖泊间的扩散不受限制,尽管均温层是一个比变温层更孤立的栖息地。
◆ 研究亮点:
新的利用长度长扩增子测序的方法补充了短读长扩增子测序的有限分辨率和基于宏基因组组装的方法的有限灵敏度,突出了淡水浮游细菌谱系普遍存在的复杂生态过程。
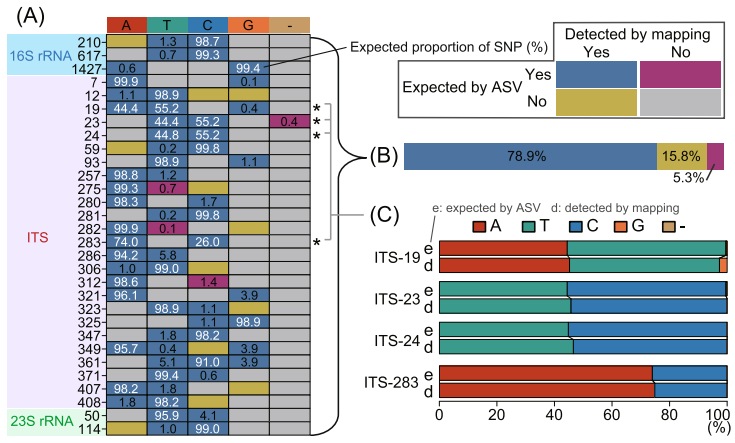
图1. 琵琶湖CL500-11序列基于扩增序列变异(ASV)的单核苷酸多态性(SNPs)与利用宏基因组技术检测到的SNPs的比
02、环境真菌研究
◆ 文献标题:Long- and short-readmetabarcoding technologies reveal similar spatio-temporal structures infungal communities长读长和短读长测序元条形码技术揭示了真菌群落中相似的时空结构
◆ 期刊:Molecular Ecology Resources
◆ 发表时间:2021年3月
◆ 内容摘要:
真菌群落在陆地生态系统中发挥着重要作用,但从环境中确定真菌的种类并开展系统发育定位研究仍存在相当挑战。这项研究中研究者采用四种测序策略研究真菌群落的时空结构:分别用 illumina Miseq、Ion Torrent IonS5和Pacbio RSII平台对ITS2区域(~300-400bp长)扩增子进行测序,另外采用PacbioRSII平台进行ITS全长及LSU部分区域(1200-1600bp长)的扩增子测序。研究发现。由此产生的群落结构和多样性更多依赖于统计方法而非测序技术及平台。使用长度长reads可以从元读取中构建系统发育树,有助于序列的分类鉴定;但同时,长度取存在不同群落去噪算法的问题。对此,研究者提出一个解决方案,即将长读取分成短的同源区去噪后,再重新构建完整的去噪读取。在研究方案选择上,研究者建议,采用混合方法:用短读取实现高采样的数量和深度,用长读取表征本地物种库,以此改进物种识别和系统发育分析。
◆ 研究亮点:
比较不同的测序平台,测序方案及分析方法对样本微生态表征的差异,提出了长读取与短读取混合分析方案以优化真菌微生态的时空结构研究。
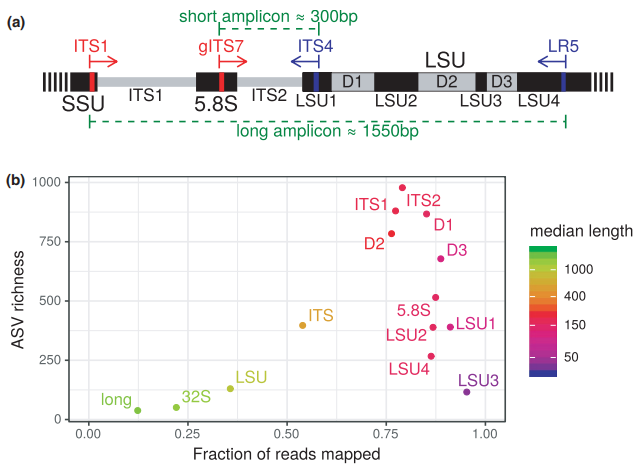
图2. a)真菌ITS扩增区域展视;b)DADA2 ASV 的总数与使用 LSUx 从一组长 PacBio 扩增子序列中提取的不同 rDNA 区域成功映射到 ASV 的解复用读数的比例
03、动物微生态研究
◆ 文献标题:Using PacBio sequencing to investigatethe effects of treatment with lactic acid bacteria or antibiotics on cowendometritis
使用PacBio测序研究乳酸菌和抗生素治疗奶牛子宫内膜炎的效果
◆ 期刊:Electronic Journal of Biotechnology
◆ 发表时间:2021年3月
◆ 内容摘要:
子宫内膜炎是奶牛最常见的疾病,一般使用抗生素进行治疗。乳酸菌既能抑制病原体生长,又有治疗子宫内膜炎的潜力。本研究采用PacBio单分子实时测序技术,对31例子宫内膜炎奶牛子宫粘液样本中微生物群的16S rRNA基因全长进行了测序,其中乳酸菌处理组(实验组)和抗生素对照组(对照组)分别为实验组(E组)和对照组(C组)。比较治疗前后采集的微生物群。结果:两种处理后细菌物种丰富度均显著高于处理前,但细菌多样性差异无显著性。经乳酸菌和抗生素处理后,Lactobacillus helveticus,Lactococcus lactis,Lactococcus raffinolactis,Pseudomonas alcaligenes和Pseudomonasveronii等菌种丰度增加。数量显着减少的细菌种类因奶牛是否使用了乳酸菌或抗生素而有所不同。Staphylococcus equorum和Treponemabrennaborense的丰度在乳酸菌处理后增加,在抗生素处理后降低。根据基于COG的功能宏基因组预测,384个功能蛋白在治疗后有显著差异。E组和C组蛋白表达途径均明显高于治疗前(p<0.05)。
◆ 结论:
本研究发现乳酸菌可用于治疗子宫内膜炎,恢复正常生理状态,同时避免了抗生素治疗带来的有益微生物群丰度降低的缺点,表明乳酸菌具有替代抗生素用于治疗牛子宫内膜炎的潜力。
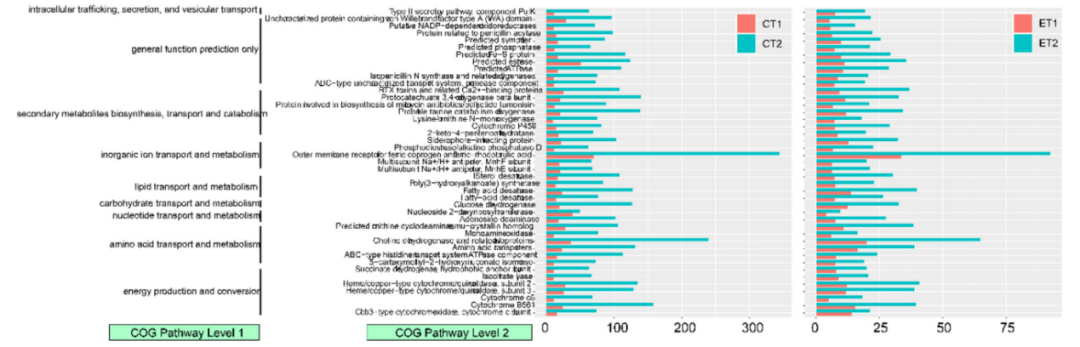
图3. 不同特定COG注释的代谢途径和相应蛋白质的数量
04、昆虫微生态研究
◆ 文献标题:Differences in the intenstinalmicrobiota between insecticide-resistant and -sensitive Aedes albopictus basedon full-length 16S rRNA sequencing.
基于16S rRNA全长测序研究抗杀虫剂及对杀虫剂敏感的白纹伊蚊的肠道微生态差异。
◆ 期刊:Microbiology Open
◆ 发表时间:2021年2月
◆ 内容摘要:
白纹伊蚊的肠道共生菌在宿主对杀虫剂的抗性中发挥潜在作用。在这项研究中,利用16s rRNA全长测序技术,对具有杀虫剂抗性和敏感性白纹伊蚊肠道微生态进行了测序,并分析了两者之间肠道微生物群落的差异。此外,研究在好氧和厌氧环境中使用六种培养基培养和分析共生菌。我们发现两种白纹伊蚊的肠道微生物群的多样性和丰度存在显着差异。发现体外培养的共生菌主要是兼性厌氧菌。米沙雷菌(Serratia oryzae)、朱氏不动杆菌(Acinetobacter junii)等培养菌可能具有促进杀虫剂抗药性发展的作用。研究表明肠道微生态有助于增强白纹伊蚊对杀虫剂的抗性,并指出16s rRNA全长测序对研究蚊子的肠道微生态有分析优势。
◆ 研究亮点:
研究采用16s rDNA全长测序技术对具有杀虫剂抗性和不具杀虫剂抗性的白纹伊蚊的肠道微生态进行测序,基于全长测序的高分辨率的识别出白纹伊蚊肠道微生态中对抗杀虫剂有积极响应的沙雷氏菌和不动杆菌物种,说明16s全长测序可作为研究蚊虫肠道微生态的理想技术。
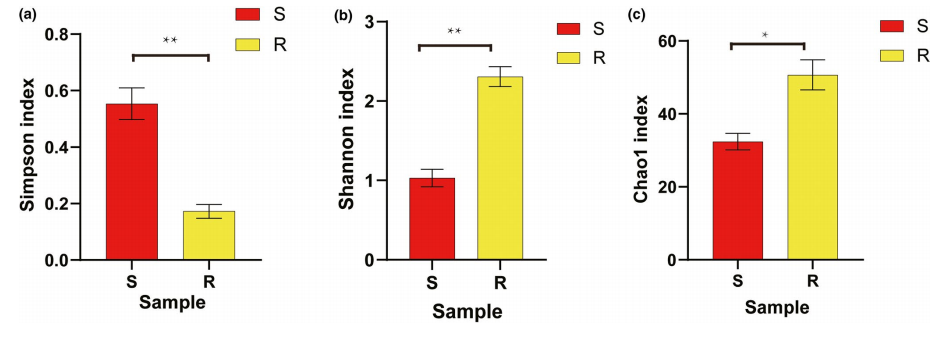
图4.抗药性和敏感型白纹伊蚊肠道微生态的多样性指数差异。a-c分别为Simpson指数、Shannon指数和Chao指数。
05、人体微生态研究
◆ 文献标题:High-Resolution Differentiation ofEnteric Bacteria in Premature Infant Fecal Microbiomes Using a Novel rRNAAmplicon
利用一种新的rRNA扩增子高分辨率鉴定早产儿粪便微生物组的肠道细菌
◆ 期刊:mBio
◆ 发表时间:2021年2月
◆ 内容摘要:
随着微生物群的进化,识别和跟踪微生物菌株是微生物组研究领域的主要挑战。研究者利用了一种新的测序试剂盒,它结合了DNA提取、rRNA操纵子大区域的PCR扩增和下游生物信息学数据分析。来自两个不同新生儿重症监护病房(NICU)的共入院双胞胎的纵向微生物组样本被用跨越16S和23S rRNA基因并比对到新的自定义的16S-23S rRNA数据库的2500个碱基扩增子进行了分析。使用DADA2推断的扩增序列变体(ASV)为区分rRNA变体提供了足够的分辨率,这些菌株与克雷伯氏菌、大肠杆菌和肠杆菌菌株关系密切,但之前未测序,这些菌株是NICU入院后在这些婴儿肠道中定殖的第一批细菌。不同的ASV群体随着时间的推移在双胞胎之间被监测,证明了追踪共生体和病原体的来源和传播的可能性。从长扩增子测序获得的高分辨率分类,可在医院环境中婴儿建立微生物组,在时间和空间上跟踪菌株。
◆ 研究亮点:
本研究突破16s rDNA扩增子全长的局限,充分利用PacBio三代测序的技术优势,将条形码范围扩大到16s+23s的超长区域,在人体微生态研究中实现了菌株级别分辨,显著提高了扩增子研究的分辨率。

图5. 双胞胎A/B和Y/Z的细菌群落构成。研究对两对双胞胎在出生后第1周至第8周(双胞胎A/B)或第9周(双胞胎Y/Z)的粪便样本进行了群落结构组成分析。
从上述5篇近期发表的扩增子研究进展中不难看出,伴随着三代测序技术应用的推广和深入研究者已经不能满足基于短读取的扩增子研究,而更倾向于打破二代测序读长短的瓶颈,将三代的全长扩增子研究应用到更多领域研究,如真菌群落,真核生物群落研究等;并将全长扩增子的范围进一步扩大,不仅仅局限在既往研究的指定经典区域,而将扩增子区域选择范围放得更大(如16s+23S, ITS+LSU等),获得更长的序列信息以换取更高的物种分辨率。
以细菌的16S研究为例,基于16s全长区域,可以通过长度长测序获得更长的序列信息和物种变异位点,在此前提下长读取研究方案可以在同一分类水平下实现更多物种分类,更有研究者基于16S全长测序获得的长序列中的单核苷酸变异信息实现了对菌株的区分,获得详尽的菌株分类信息,在更精细的物种鉴定中显示出绝对优势这一点很大程度满足了微生物群落研究者对物种鉴定精细度的要求。
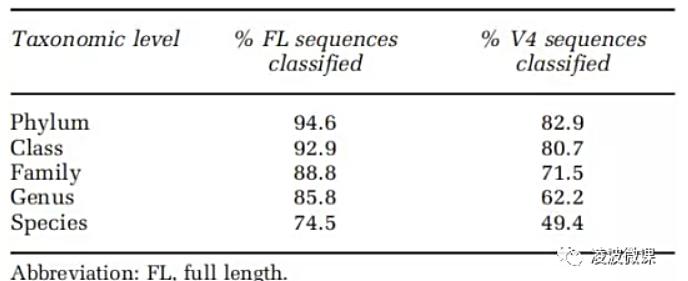
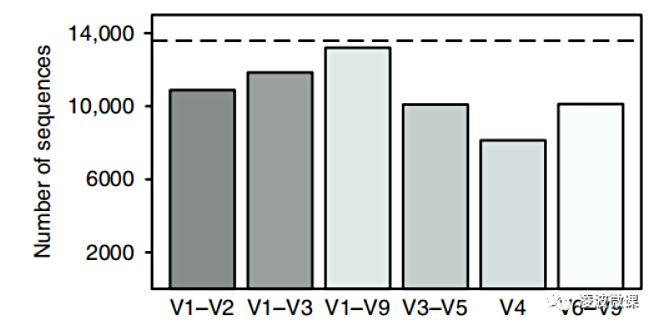
由于16S全长测序获取的长序列信息,可极大程度提升和改善序列不能被识别的状况。有研究者比对了基于16s不同高变区和16s全长研究的差异:不难看出V1-V9区引物很少有序列不能被明确分类,相比之下,仅仅开展V4区的研究,则有极高比例的序列无法实现明确分类。
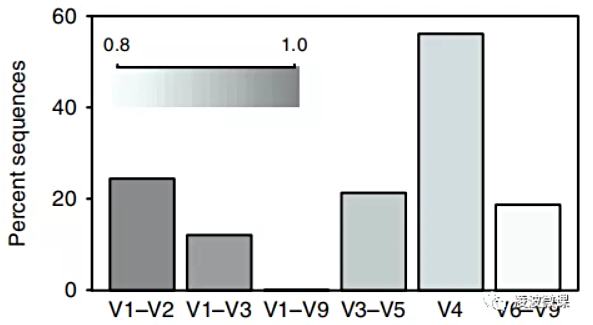
此外,通过聚类OTU,并与模拟物种库进行比较,研究者还发现,V1-V9区研究实现了对样本的最佳还原(下图),物种的识别和定量是最接近模拟物种库原貌的,这一点说明了基于三代平台的16s全长测序在保真效果上的巨大优势。
同时,由于三代测序技术在建库等实验操作过程中无需PCR过程,这就最大程度保持了样本的真实微生态状态,降低了研究结果偏差。
不难看出,伴随着三代长度长测序技术的发展,全长扩增子测序的优势逐渐显现,高分辨率表征微生态群落结构,精细的物种识别和对样本状态的高保证还原。全场扩增子测序技术势必取代基于NGS技术的扩增子研究,成为微生态研究领域的新热点和发展的必然趋势。