1、前言
unetPlusPlus 在unet 的基础上增添了密集连接的结构,有点像densenet网络
因为这种密集连接,unet++可以实现剪枝的轻量化操作。由于本人的没有接触过这种剪枝、蒸馏之类的轻量化方法,所以不多赘述
本章仅仅根据unet++模型实现医学图像分割的任务,为了更好的调节参数(学习率衰减策略、优化器等等),做对比实验,这里统一进行实现
项目下载在最后
2、罗里吧嗦的话
医学图像分割任务中,二值分割任务较多,这里实现的代码仅对二值分割数据处理
2.1 dice
因为之前实现的评估指标都是iou,但之前很多人联系本人都想要dice来评估模型。其实dice和iou指标是一一对应的,两者之间存在函数关系,之前 Unet 代码加个函数就可以简单实现,当然这样计算略显麻烦,可以直接通过TP、FP直接计算dice更好

二值分割的任务,网络的输出可以直接固定为1,这样通过对输出结果进行阈值处理得到的仍然是二值图像。虽然之前使用的都是交叉熵,取不同channel 输出概率最大值的索引当做输出的灰度值,多类别方便
但其实一开始我们分割二值图像的时候,用的就是输出通道为1,然后根据0阈值判断前景和背景的,例如:UNet - 训练数据train_unet train-CSDN博客
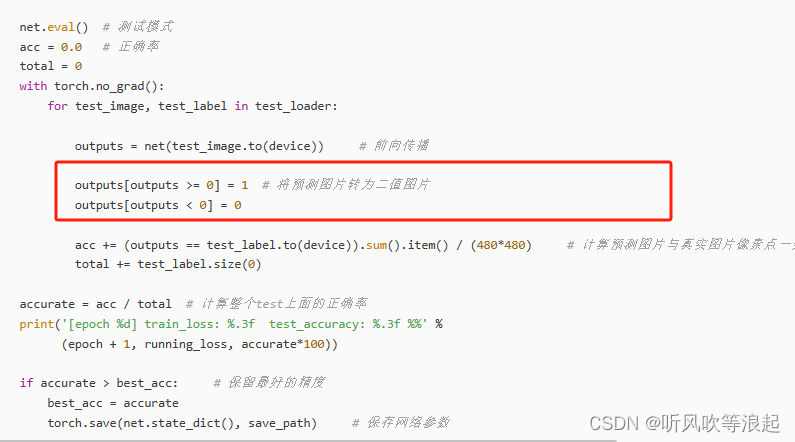
这样好处很多,减少运算量啊、可以用更好的损失函数等等。
本章代码使用的目标函数就是BCE逻辑损失函数
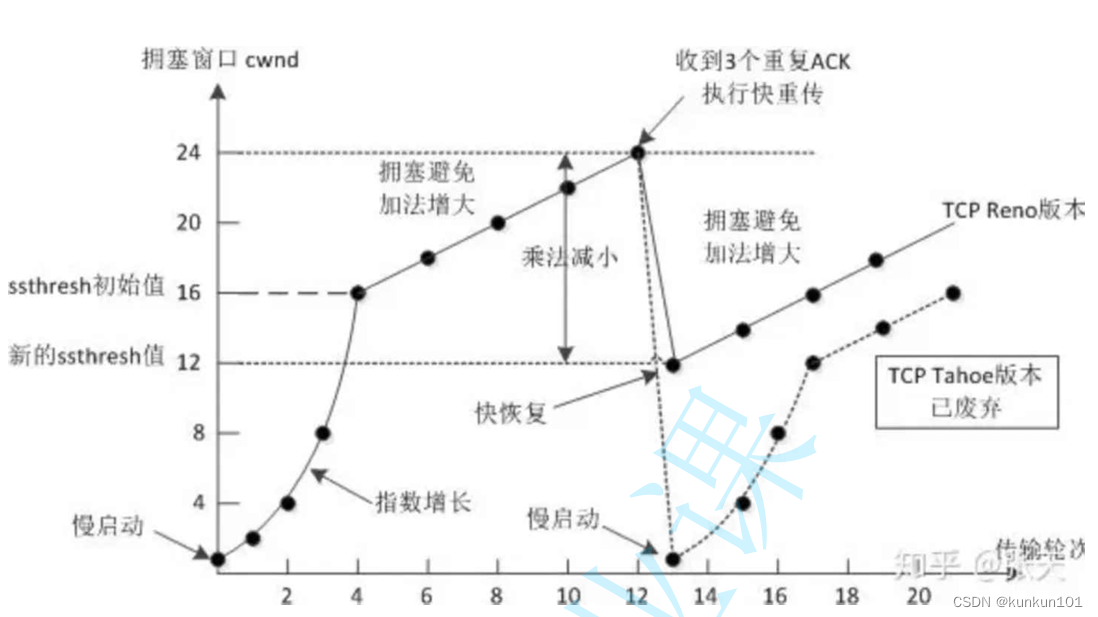
2.2 学习率衰减策略
学习率的衰减策略采用下面三个:
恒定的学习率、step 学习率衰减、cos余弦退火衰减

黄色框是统一的设定的初始学习率
红色框指定选择的哪一种学习效衰减策略
蓝色框是cos衰减的衰减因子,最后的lr = 初始lr * lrf
绿色框是step衰减的epoch、gamma系数。本章中,代码会在第10和第20个epoch的时候,lr衰减0.1倍率
2.3 加载数据的一些问题
我们的分割是有监督学习,也就是image有对应的mask标签,那么如何对应数据和标签很重要。当然实现很简单,基本上数据都是文件名一样或者后缀有一点点不一样的。
这里更改下面参数即可,上面是数据的后缀,下面是mask数据的后缀

在标签为图像的分割数据里,mask灰度值不严谨就很难受。
因为有的任务明明是二值分割,然而有的数据为了方便可视化啊之类的,mask用的是灰度数据,而非0 1这样的阈值图像。当然不管怎么标注,只要标注是对的,都可以根据自定义的数据加载脚本进行数据预处理。
本章处理的很简单,也是阈值处理
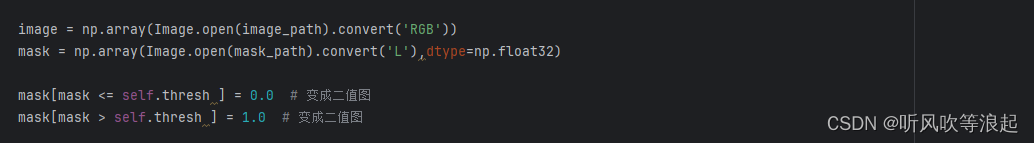
这里的阈值不太好确定,但基本上都是0为背景,其余的灰度值为前景。
所以这里通过下面的参数进行阈值处理
3、测试项目
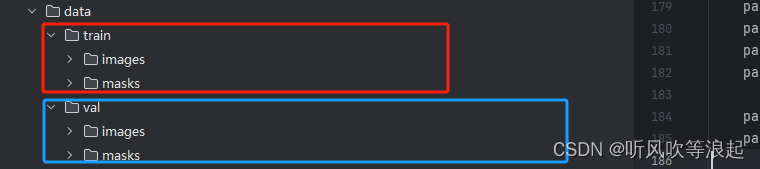
数据集按照下面摆放即可
3.1 训练
训练展示部分如下:
训练过程:
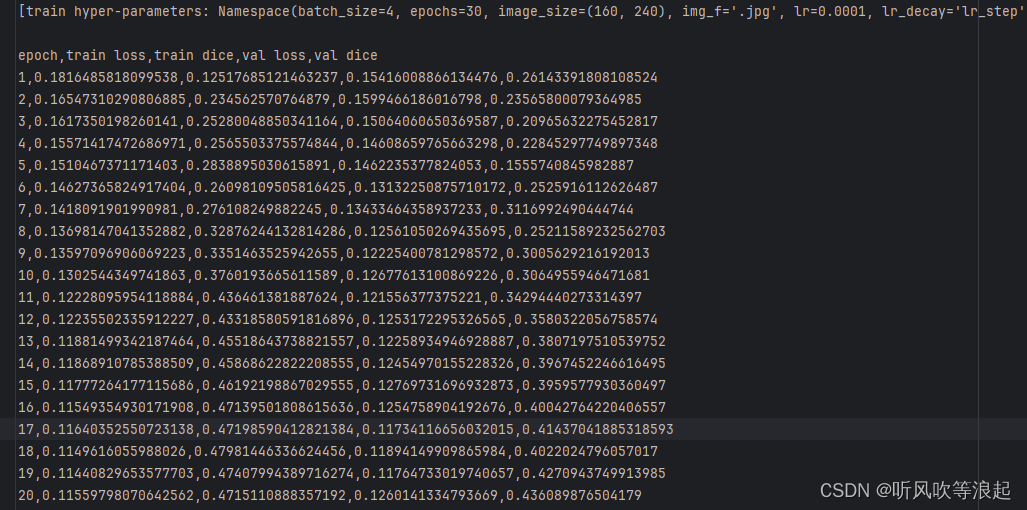
需要注意的是,进度条的loss、dice显示是实时的一个batch的loss和dice
训练一轮的打印是整个数据集的平均loss和dice
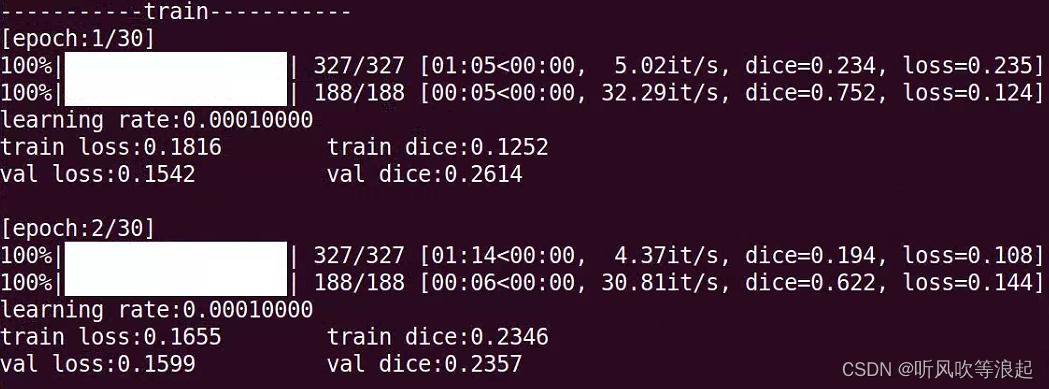
训练结束后:会载入最好权重进行验证

3.2 生成的结果
如下:
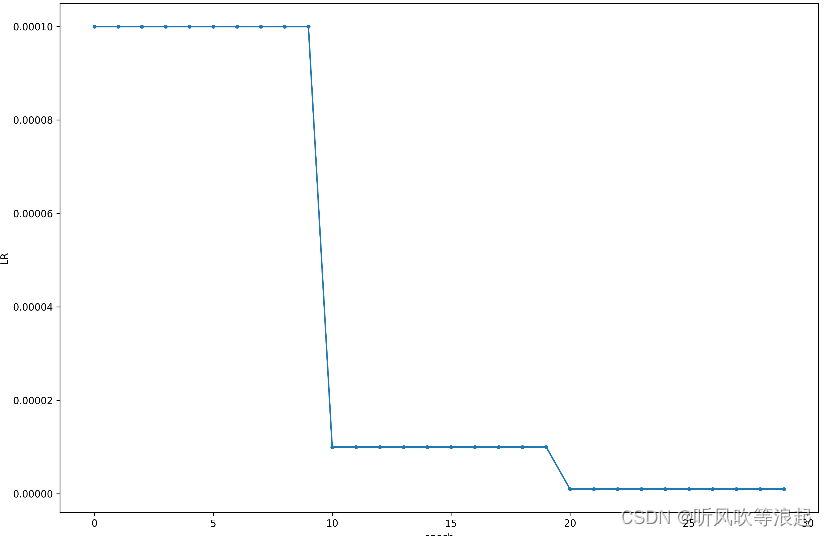
学习率下降图:
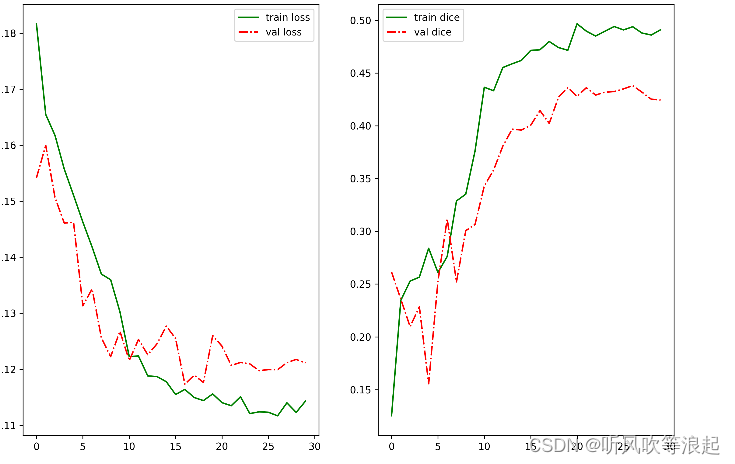
loss和dice图
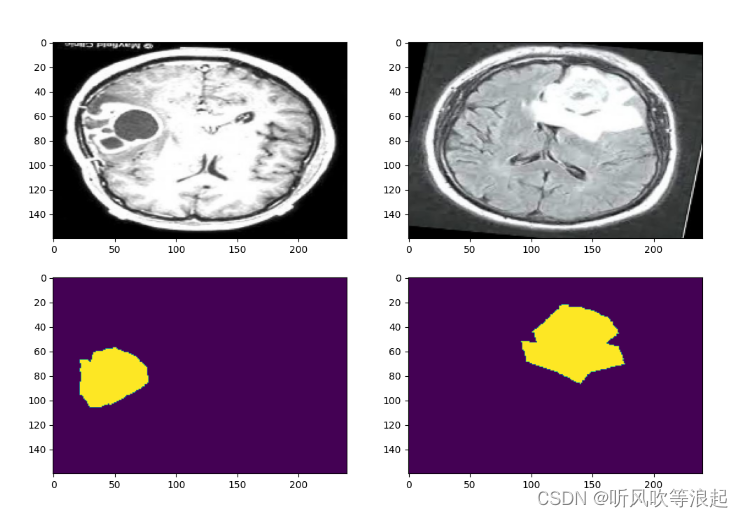
预处理数据结果图:
训练日志:
3.3 推理
需要推理的图像放在inference下即可:
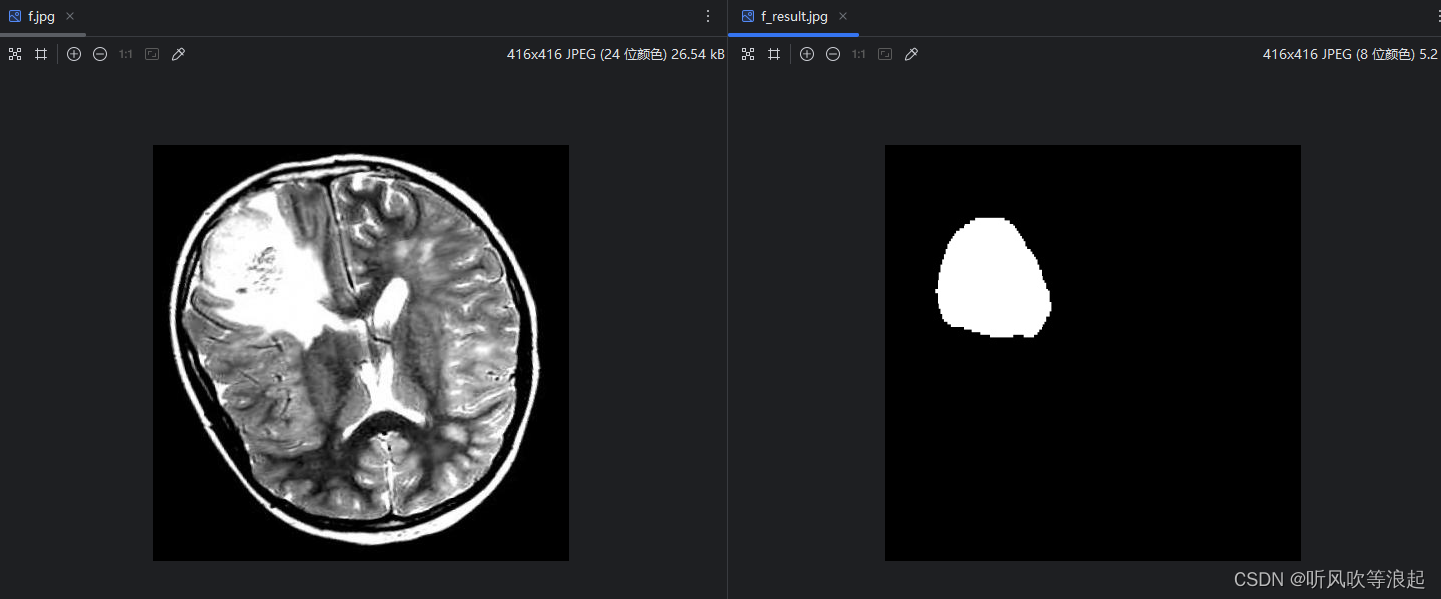
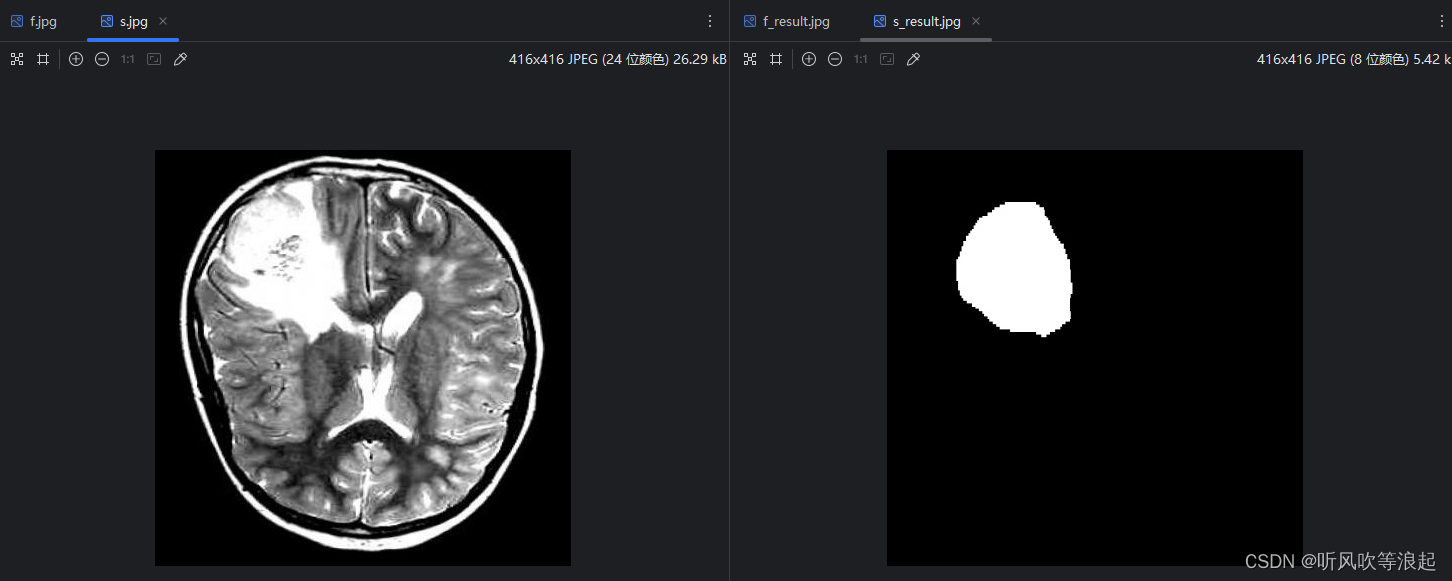
本项目测试的数据为大脑MRI的肿瘤分割:基于UnetPlusPlus网络对大脑肿瘤分割实战【包含数据集、完整代码、训练好的结果、权重文件等等】资源-CSDN文库