近日,凌恩生物客户重庆医科大学在《Water Research》(IF= 13.4)发表研究论文“Supervised machine learning improves general applicability of eDNA metabarcoding for reservoir health monitoring”。该研究主要介绍了一种基于eDNA的机器学习方法,具有广泛的适用性,可用于生物监测,评估水库的健康状况。

传统的生物监测方法存在许多局限性,如:采样和鉴定困难、难以监测微小生物、难以监测季节变化。建立有效和标准化的监测方法对于水库的成功恢复和管理至关重要。环境DNA(eDNA)宏条码测序为生物监测提供了一种很有前途的选择,并可以克服传统形态学生物评估的许多局限性。本文提出了一种基于eDNA的机器学习方法,该方法通过综合评估水库的物理化学和生物指标,开发了一种新的生物完整性指数(Me-IBI),该指数整合了多种营养相互作用和环境信息,可以克服许多传统生物监测方法的局限性。本文详细介绍了该方法的实施过程,包括样本采集、数据分析和模型建立等步骤,为大规模、标准化的生物监测应用奠定了坚实的基础。
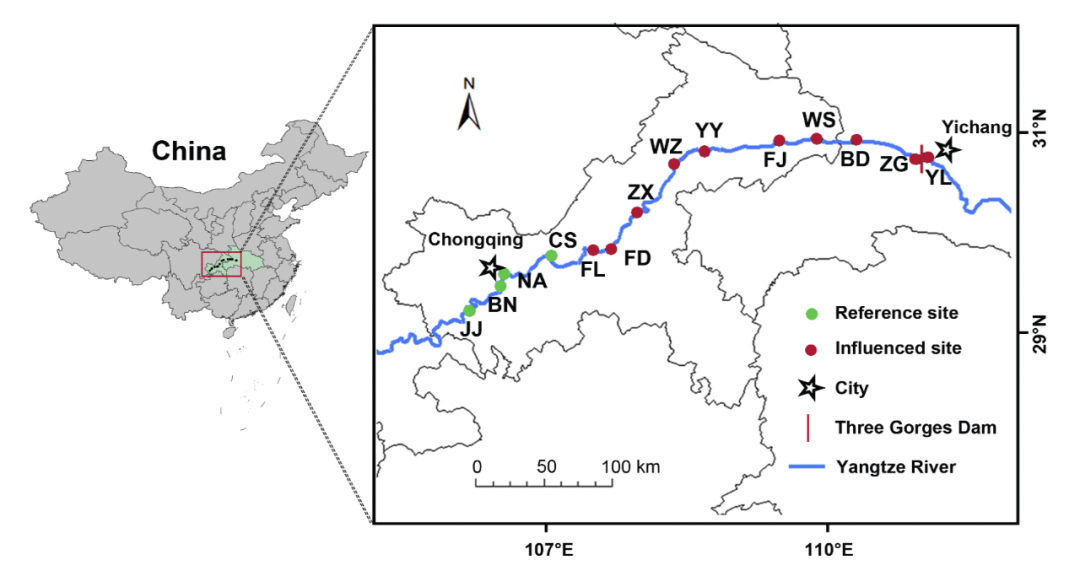
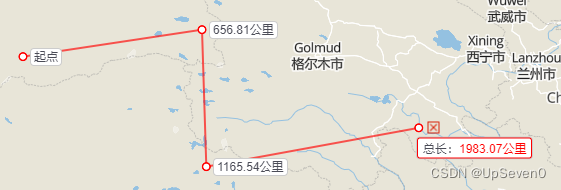
图1 研究采样点

本文研究了三峡水库的水质评估和生态系统健康监测。作者采用了多种方法,包括单因素污染指数和水质指标测定法、eDNA测序法、生物信息学分析、以及机器学习方法。开发了一种基于eDNA的指标(Me-IBI),用于评估水库生态系统的健康状况。结果表明,Me-IBI比物理化学评估更能区分三峡水库的实际健康状况,比较了不同机器学习方法在预测Me-IBI时的性能,即使只有少量的特征,不同的SML算法也可以建立稳定的模型,并获得优异的预测性能。同时探讨了人类活动和营养状况对Me-IBI的影响。最后,提出了一种基于eDNA和机器学习的标准化健康监测方法,可用于监测河流-水库生态系统的健康状况。

图2 三峡水库健康状况综合评价
(a)根据《中国地表水环境质量标准》(GB3838-2002),采用单因素污染指标法(SFPI)对水质进行评价。(b)两个季节TGR 14个采样点(平均±标准差值)计算WQI的变异和健康分类。两个季节TGR 14个采样点(平均±标准差值)计算的Me-IBI的(c)变异和健康分类。(d)根据SFPI、WQI和Me-IBI对两季水质健康评估结果的比较。

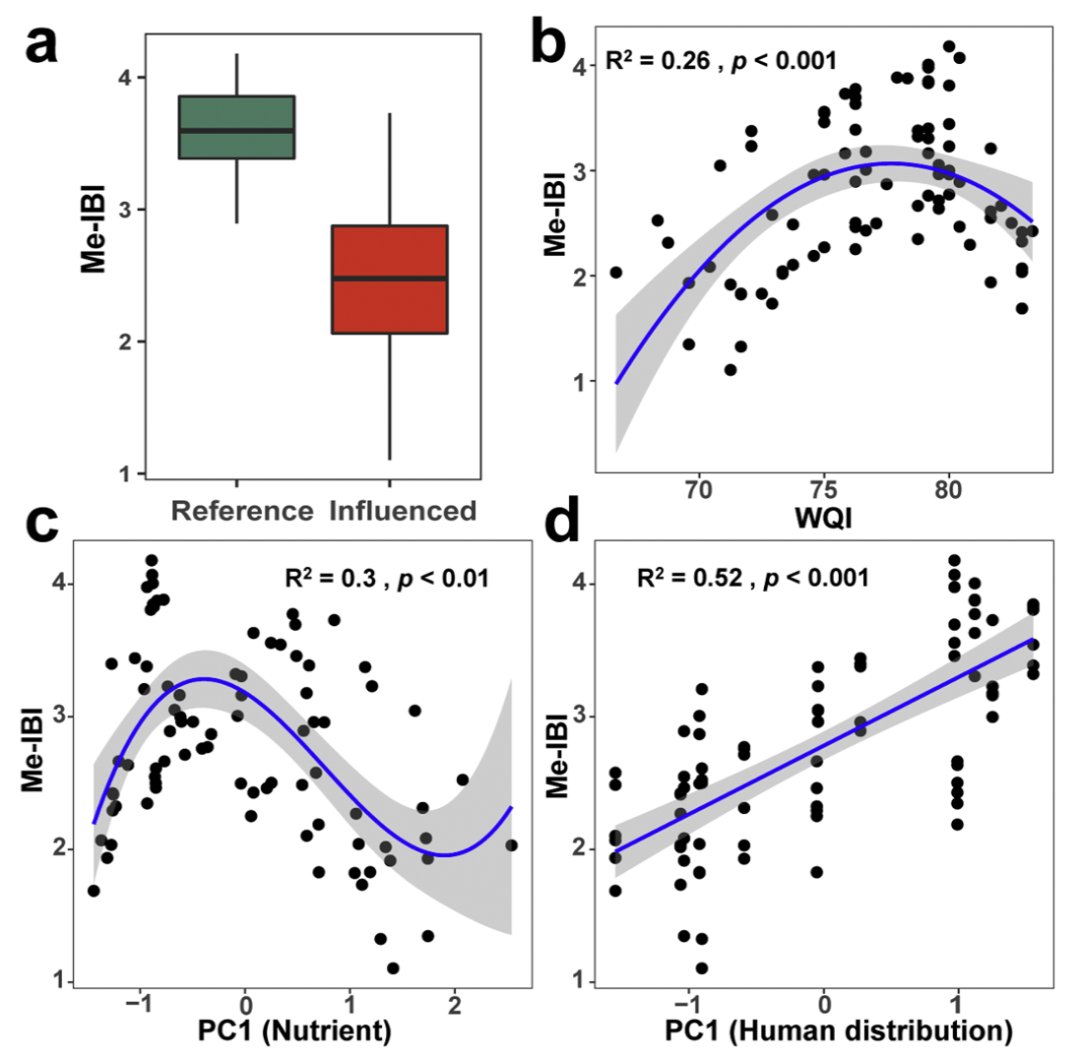
图3 Me-IBI对大坝(a)、WQI (b)、营养状况(PC1表示,c)和人类分布(PC1表示,d)的响应。非线性多项式回归包括95%CI(阴影区域)的WQI(二次)和营养(立方)。线性回归包括人类分布中的95%CI(阴影区域)。

图4 使用随机森林进行特征筛选
(a)确定预测两个季节水库健康状况的所有特征。(b)通过5次重复10次交叉验证确定重要特征的数量。(c)核心特征的确定交叉出现在重要特征下100个重复。


图5 随机森林(RF)和支持向量机(SVM,具有四个核技巧)模型在预测TGR中的Me-IBI时使用三个特征数的预测性能。实际和预测的Me-IBI值之间的线性值r2值,测量实际和预测的Me-IBI值之间的差异,以及测量实际和预测的Me-IBI衍生健康状态之间的一致性的kappa值在箱线图上表示。


图6 使用不同的算法获得的最好的预测模型与三个特征数。获得的参考Me-IBI值绘制在横轴上,而通过无分类法数据获得的预测值绘制在纵轴上。彩色的矩形代表离散的质量分级,从蓝色的“优秀”到红色的“差”。灰色点表示来自训练数据集的预测,而红点表示来自测试数据集的预测。


参考文献
Supervised machine learning improves general applicability of eDNA metabarcoding for reservoir health monitoring. Water Research, 2023.
原文链接
Doi.org/10.1016/j.watres.2023.120686