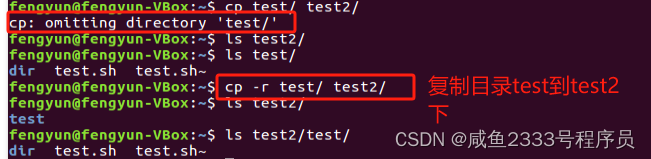
在火山图中,我们有时候会想要标注出自己感兴趣的基因,这个时候该怎么嘞!
还有还有,在添加标签时,可能会遇到元素过多或位置密集导致标签显示不全,或者虽然显示全了但显得密集杂乱,不易阅读的情况。这可咋整捏!
话不多说,直接开干!
(本节内容同时适用于其他散点型图表)
数据处理
今日份绘图所用到的数据是之前的看完还不会来揍/找我 | 差异分析三巨头 —— DESeq2、edgeR 和 limma 包 | 附完整代码 + 注释的结果,有兴趣的小伙伴可以去瞅瞅哟!如果不想再折腾,也是完全OK的!我已经把数据上传到了GitHub,大家可以在公众号后台回复火山图,即可获得存放数据的链接。不过我在分享过程中也会把每一步的输入数据和输出结果进行展示,大家可以作为参考并调整自己的数据格式,然后直接用自己的数据跑,也是没有任何问题的!
# 带标签的火山图 | 标记感兴趣基因
# 加载包,没安装的记得安装一下哟!
library(tidyverse)
library(ggplot2)
library(ggrepel)
# 加载差异分析结果
load("./DEG_limma_voom.Rdata")
head(DEG_limma_voom)
# logFC AveExpr t P.Value adj.P.Val B
# FIGF -5.984847 -0.7193930 -51.67041 1.843289e-309 4.938355e-305 698.5939
# CA4 -6.844833 -2.5701167 -44.96985 3.340380e-261 4.474605e-257 587.5876
# PAMR1 -3.989305 2.3605059 -44.85958 2.161519e-260 1.665003e-256 585.9261
# LYVE1 -4.786578 1.3531474 -44.85132 2.485914e-260 1.665003e-256 585.7724
# CD300LG -6.537456 -0.0898487 -43.57667 6.384798e-251 3.421102e-247 564.1320
# SDPR -4.600471 2.7186631 -43.38389 1.712581e-249 7.646961e-246 560.8745
# 加change列,标记上下调基因,可根据需求设定阈值
logFC = 2.5
P.Value = 0.01
k1 <- (DEG_limma_voom$P.Value < P.Value) & (DEG_limma_voom$logFC < -logFC)
k2 <- (DEG_limma_voom$P.Value < P.Value) & (DEG_limma_voom$logFC > logFC)
DEG_limma_voom <- mutate(DEG_limma_voom, change = ifelse(k1, "down", ifelse(k2, "up", "stable")))
# 再加个基因列,方便后续加标签
DEG_limma_voom$symbol <- rownames(DEG_limma_voom)
head(DEG_limma_voom)
# logFC AveExpr t P.Value adj.P.Val B change symbol
# FIGF -5.984847 -0.7193930 -51.67041 1.843289e-309 4.938355e-305 698.5939 down FIGF
# CA4 -6.844833 -2.5701167 -44.96985 3.340380e-261 4.474605e-257 587.5876 down CA4
# PAMR1 -3.989305 2.3605059 -44.85958 2.161519e-260 1.665003e-256 585.9261 down PAMR1
# LYVE1 -4.786578 1.3531474 -44.85132 2.485914e-260 1.665003e-256 585.7724 down LYVE1
# CD300LG -6.537456 -0.0898487 -43.57667 6.384798e-251 3.421102e-247 564.1320 down CD300LG
# SDPR -4.600471 2.7186631 -43.38389 1.712581e-249 7.646961e-246 560.8745 down SDPR
table(DEG_limma_voom$change)
# down stable up
# 749 25664 378
普通火山图
# 普通火山图
p <- ggplot(data = DEG_limma_voom,
aes(x = logFC,
y = -log10(P.Value))) + # 设置x轴为logFC,y轴为-P.Value的对数值
geom_point(alpha = 0.4, size = 3.5,
aes(color = change)) + # 添加散点,根据change列着色
ylab("-log10(Pvalue)") + # 设置y轴标签
scale_color_manual(values = c("blue4", "grey", "red3")) + # 设置颜色映射,蓝色表示下调,灰色表示稳定,红色表示上调
geom_vline(xintercept = c(-logFC, logFC), lty = 4, col = "black", lwd = 0.8) + # 添加垂直参考线,用于标记logFC阈值
geom_hline(yintercept = -log10(P.Value), lty = 4, col = "black", lwd = 0.8) + # 添加水平参考线,用于标记-P.Value阈值
theme_bw() # 使用网格白底主题
p

光秃秃的火山图,咱们给它装扮一下!
带标签的火山图
按需求添加标签
比如你只想展示你关注的那几个基因,咱们可以这样做!
# 标签添加
# 按需求添加标签
# 创建一个新的列label,并初始化为NA
DEG_limma_voom$label <- NA
# 根据symbol的值,为特定基因添加标签信息
DEG_limma_voom$label[which(DEG_limma_voom$symbol == "MYOC")] <- "MYOC"
DEG_limma_voom$label[which(DEG_limma_voom$symbol == "PALB2")] <- "PALB2"
DEG_limma_voom$label[which(DEG_limma_voom$symbol == "NEK2")] <- "NEK2"
DEG_limma_voom$label[which(DEG_limma_voom$symbol == "ARF1")] <- "ARF1"
# 在普通火山图p的基础上,添加标签,并使用geom_label_repel函数进行标签的绘制
p0 <- p + geom_label_repel(data = DEG_limma_voom, aes(label = label),
size = 4, # 设置标签大小
box.padding = unit(0.5, "lines"), # 设置标签内边距
point.padding = unit(0.8, "lines"), # 设置标签与点的距离
segment.color = "black", # 设置标签边界线颜色
show.legend = FALSE, # 不显示图例
max.overlaps = 10000) # 设置标签重叠的最大次数
p0
# geom_text_repel()和geom_label_repel()这两个函数都可用于添加标签,咱们下面一个一个来看!

看!它们被标记了!你可以标记出任意你感兴趣的基因们!
按条件添加标签
比如在上下调基因中选择显著性top10的基因给它标注出来,或者选择差异倍数top20的基因进行标注啦,又或者其他,条件随你定!
# 按条件添加标签
# 筛选上调中显著性top10的基因
up_data <- filter(DEG_limma_voom, change == 'up') %>% # 从DEG_limma_voom中筛选出上调的基因
distinct(symbol, .keep_all = TRUE) %>% # 去除重复的基因,保留第一个出现的行
top_n(10, -log10(P.Value)) # 选择-P.Value值最大的前10个基因
# 筛选下调中显著性top10的基因
down_data <- filter(DEG_limma_voom, change == 'down') %>% # 从DEG_limma_voom中筛选出下调的基因
distinct(symbol, .keep_all = TRUE) %>% # 去除重复的基因,保留第一个出现的行
top_n(10, -log10(P.Value)) # 选择-P.Value值最大的前10个基因
head(up_data); head(down_data)
# logFC AveExpr t P.Value adj.P.Val B change symbol
# HSD17B6 3.206654 1.840588 25.15742 7.624913e-113 5.081568e-111 246.7745 up HSD17B6
# NEK2 4.747981 3.840978 24.33444 6.029187e-107 3.605535e-105 233.2608 up NEK2
# AC093850.2 6.047425 1.102478 24.22747 3.483702e-106 2.042273e-104 231.3813 up AC093850.2
# CDC25C 3.808098 1.872805 23.89870 7.515027e-104 4.151239e-102 226.1433 up CDC25C
# SPC25 3.088991 2.332539 23.72996 1.173282e-102 6.350181e-101 223.4213 up SPC25
# PKMYT1 4.415991 3.432915 23.22566 4.144003e-99 2.071306e-97 215.2842 up PKMYT1
# logFC AveExpr t P.Value adj.P.Val B change symbol
# FIGF -5.984847 -0.7193930 -51.67041 1.843289e-309 4.938355e-305 698.5939 down FIGF
# CA4 -6.844833 -2.5701167 -44.96985 3.340380e-261 4.474605e-257 587.5876 down CA4
# PAMR1 -3.989305 2.3605059 -44.85958 2.161519e-260 1.665003e-256 585.9261 down PAMR1
# LYVE1 -4.786578 1.3531474 -44.85132 2.485914e-260 1.665003e-256 585.7724 down LYVE1
# CD300LG -6.537456 -0.0898487 -43.57667 6.384798e-251 3.421102e-247 564.1320 down CD300LG
# SDPR -4.600471 2.7186631 -43.38389 1.712581e-249 7.646961e-246 560.8745 down SDPR
# 使用geom_text_repel()函数添加标签,常规样式,默认参数配置
p1 <- p + # 基于普通火山图p
geom_text_repel(data = up_data, aes(x = logFC, y = -log10(P.Value), label = symbol)) + # 添加上调基因的标签
geom_text_repel(data = down_data, aes(x = logFC, y = -log10(P.Value), label = symbol)) # 添加下调基因的标签
p1

再来看看带框框的标签!
# 使用geom_label_repel()函数添加标签,带边框样式,默认参数配置
p2 <- p + # 基于普通火山图p
geom_label_repel(data = up_data, aes(x = logFC, y = -log10(P.Value), label = symbol)) + # 添加上调基因的标签
geom_label_repel(data = down_data, aes(x = logFC, y = -log10(P.Value), label = symbol)) # 添加下调基因的标签
p2

俺觉得这种的更好看!个人观点,仅供参考!最终解释权归小蛮要所有!
标签重叠怎么办
哎嘿!上面这俩图我们可以看出,并不是所有期望的基因都被成功标注,这是为啥捏!因为它们太挤了,都重叠在一起了,那要怎么办呢?咱们看下面!
# 基因太挤怎么办!
# 以我喜欢的geom_label_repel为例
# 强调显示上调基因的散点并添加带边框的标签
p3 <- p + # 基于普通火山图p
geom_point(data = up_data, # 添加强调显示的散点,仅限于上调基因
aes(x = logFC, y = -log10(P.Value)),
color = 'red3', size = 4.5, alpha = 0.2) + # 设置散点的颜色、大小和透明度
geom_label_repel(data = up_data, # 添加带边框的标签,仅限于上调基因
aes(x = logFC, y = -log10(P.Value), label = symbol),
seed = 233, # 设置随机数种子,用于确定标签位置
size = 3.5, # 设置标签的字体大小
color = 'black', # 设置标签的字体颜色
min.segment.length = 0, # 始终为标签添加指引线段
force = 2, # 设置标签重叠时的排斥力
force_pull = 2, # 设置标签与数据点之间的吸引力
box.padding = 0.4, # 设置标签周围的填充量
max.overlaps = Inf) # 设置排斥重叠过多标签的阈值为无穷大,保持始终显示所有标签
p3

# 上面是只有上调基因,咱们把下调也加上!当然,大家也可以在前面先对数据进行整合后再画图!
p4 <- p + # 基于普通火山图p
geom_point(data = up_data, # 添加强调显示的上调基因的散点
aes(x = logFC, y = -log10(P.Value)),
color = 'red3', size = 4.5, alpha = 0.2) + # 设置散点的颜色、大小和透明度
geom_label_repel(data = up_data, # 添加带边框的标签,仅限于上调基因
aes(x = logFC, y = -log10(P.Value), label = symbol),
seed = 233, # 设置随机数种子,用于确定标签位置
size = 3.5, # 设置标签的字体大小
color = 'black', # 设置标签的字体颜色
min.segment.length = 0, # 始终为标签添加指引线段
force = 2, # 设置标签重叠时的排斥力
force_pull = 2, # 设置标签与数据点之间的吸引力
box.padding = 0.4, # 设置标签周围的填充量
max.overlaps = Inf) + # 设置排斥重叠过多标签的阈值为无穷大,保持始终显示所有标签
geom_point(data = down_data, # 添加强调显示的下调基因的散点
aes(x = logFC, y = -log10(P.Value)),
color = 'blue4', size = 4.5, alpha = 0.2) + # 设置散点的颜色、大小和透明度
geom_label_repel(data = down_data, # 添加带边框的标签,仅限于下调基因
aes(x = logFC, y = -log10(P.Value), label = symbol),
seed = 233, # 设置随机数种子,用于确定标签位置
size = 3.5, # 设置标签的字体大小
color = 'black', # 设置标签的字体颜色
min.segment.length = 0, # 始终为标签添加指引线段
force = 2, # 设置标签重叠时的排斥力
force_pull = 2, # 设置标签与数据点之间的吸引力
box.padding = 0.4, # 设置标签周围的填充量
max.overlaps = Inf) # 设置排斥重叠过多标签的阈值为无穷大,保持始终显示所有标签
p4

# 再看看不添加指引线的效果
# 创建p5火山图,强调显示上调和下调基因的散点并添加带边框的标签,去掉指引线段
p5 <- p + # 基于普通火山图p
geom_point(data = up_data, # 添加强调显示的上调基因的散点
aes(x = logFC, y = -log10(P.Value)),
color = 'red3', size = 4.5, alpha = 0.2) + # 设置散点的颜色、大小和透明度
geom_label_repel(data = up_data, # 添加带边框的标签,仅限于上调基因
aes(x = logFC, y = -log10(P.Value), label = symbol),
seed = 233, # 设置随机数种子,用于确定标签位置
size = 3.5, # 设置标签的字体大小
color = 'black', # 设置标签的字体颜色
min.segment.length = Inf, # 改为Inf去掉指引线段
force = 2, # 设置标签重叠时的排斥力
force_pull = 2, # 设置标签与数据点之间的吸引力
box.padding = 0.4, # 设置标签周围的填充量
max.overlaps = Inf) + # 设置排斥重叠过多标签的阈值为无穷大,保持始终显示所有标签
geom_point(data = down_data, # 添加强调显示的下调基因的散点
aes(x = logFC, y = -log10(P.Value)),
color = 'blue4', size = 4.5, alpha = 0.2) + # 设置散点的颜色、大小和透明度
geom_label_repel(data = down_data, # 添加带边框的标签,仅限于下调基因
aes(x = logFC, y = -log10(P.Value), label = symbol),
seed = 233, # 设置随机数种子,用于确定标签位置
size = 3.5, # 设置标签的字体大小
color = 'black', # 设置标签的字体颜色
min.segment.length = Inf, # 改为Inf去掉指引线段
force = 2, # 设置标签重叠时的排斥力
force_pull = 2, # 设置标签与数据点之间的吸引力
box.padding = 0.4, # 设置标签周围的填充量
max.overlaps = Inf) # 设置排斥重叠过多标签的阈值为无穷大,保持始终显示所有标签
p5

标签太乱怎么办
现在,咱们已经学会了展示全部目标标签的方法。但是还存在一个问题,就是当我们需要展示的目标标签过多时,无论我们是否添加指引线,指引线的长度可能会不一致,导致整体看起来比较混乱(其实我觉得还好,一般也不需要展示过多吧!不然怎么着都丑哈哈哈)。在这种情况下,咱们就可以尝试把标签垂直整齐排列,以提高可读性和美观性。
# 对齐目标标签
p6 <- p + # 基于普通火山图p
geom_point(data = up_data, # 添加强调显示的上调基因的散点
aes(x = logFC, y = -log10(P.Value)),
color = 'red3', size = 4.5, alpha = 0.2) + # 设置散点的颜色、大小和透明度
geom_label_repel(data = up_data, # 添加带边框的标签,仅限于上调基因
aes(x = logFC, y = -log10(P.Value), label = symbol),
seed = 233, # 设置随机数种子,用于确定标签位置
size = 3.5, # 设置标签的字体大小
color = 'black', # 设置标签的字体颜色
min.segment.length = 0, # 始终为标签添加指引线段
force = 3, # 设置标签重叠时的排斥力
force_pull = 2, # 设置标签与数据点之间的吸引力
box.padding = 0.4, # 设置标签周围的填充量
max.overlaps = Inf, # 设置排斥重叠过多标签的阈值为无穷大,保持始终显示所有标签
segment.linetype = 3, # 设置线段类型为虚线
segment.color = 'black', # 设置线段颜色
segment.alpha = 0.5, # 设置线段不透明度
nudge_x = 8 - up_data$logFC, # 调整标签x轴起始位置
direction = "y", # 按y轴调整标签位置方向,若想水平对齐则为x
hjust = 1) + # 对齐标签:0右对齐,1左对齐,0.5居中
geom_point(data = down_data, # 添加强调显示的下调基因的散点
aes(x = logFC, y = -log10(P.Value)),
color = 'blue4', size = 4.5, alpha = 0.2) + # 设置散点的颜色、大小和透明度
geom_label_repel(data = down_data, # 添加带边框的标签,仅限于下调基因
aes(x = logFC, y = -log10(P.Value), label = symbol),
seed = 233, # 设置随机数种子,用于确定标签位置
size = 3.5, # 设置标签的字体大小
color = 'black', # 设置标签的字体颜色
min.segment.length = 0, # 始终为标签添加指引线段
force = 10, # 设置标签重叠时的排斥力
force_pull = 2, # 设置标签与数据点之间的吸引力
box.padding = 0.4, # 设置标签周围的填充量
max.overlaps = Inf, # 设置排斥重叠过多标签的阈值为无穷大,保持始终显示所有标签
segment.linetype = 3, # 设置线段类型为虚线
segment.color = 'black', # 设置线段颜色
segment.alpha = 0.5, # 设置线段不透明度
nudge_x = -8 - down_data$logFC, # 调整标签x轴起始位置
direction = "y", # 按y轴调整标签位置方向,若想水平对齐则为x
hjust = 1) # 对齐标签:0右对齐,1左对齐,0.5居中
p6

文末碎碎念
那今天的分享就到这里啦!我们下期再见哟!
最后顺便给自己推荐一下嘿嘿嘿!
如果我的分享对你有用的话,欢迎关注点赞在看转发分享阿巴阿巴阿巴阿巴巴巴!这可是我的第一原动力!
蟹蟹你们的喜欢和支持!!!
啊对!如果小伙伴们有需求的话,也可以加入我们的交流群:一定要知道 | 永久免费的生信交流群终于来啦!
还有兴趣的话,也可以看看我掏心掏肺的干货满满 | 给生信小白的入门小建议 | 掏心掏肺版!绝对干货满满!
如果有小伙伴对付费分析有需求的话,可以看看这里:个性化科研服务 | 付费分析试营业正式启动啦!定制你的专属生信分析!可提供1v1答疑!
入群链接后续可能会不定期更新,主要是因为群满换码或是其他原因,如果小伙伴点开它之后发现,咦,怎么失效啦!不要慌!咱们辛苦一下动动小手去主页的要咨询那里,点击进交流群即可入群!

参考资料
- https://mp.weixin.qq.com/s/qZke0E4f1m22dSNnhzzrGw
- https://mp.weixin.qq.com/s/TWI-Tt741Gqe9ERzZr23yg
- https://cloud.tencent.com/developer/article/1486128