🌞欢迎来到AI+医学的世界
🌈博客主页:卿云阁💌欢迎关注🎉点赞👍收藏⭐️留言📝
🌟本文由卿云阁原创!
📆首发时间:🌹2024年3月17日🌹
✉️希望可以和大家一起完成进阶之路!
🙏作者水平很有限,如果发现错误,请留言轰炸哦!万分感谢!
目录
CYPs功能和抑制剂
常见的机器学习算法举例
KNN实现鸢尾花分类
回归问题
随机森林
论文翻译
代码实现
数据集来源
下载初试文件
整理文件
论文地址:
gkaa166.pdf (silverchair.com)
CYPs功能和抑制剂
细胞色素P450(cytochromeP450或CYP450,简称CYP450)属于单氧酶的一类,。它参与内源性物质和包括药物、环境化合物在内的外源性物质的代谢。
根据氨基酸序列的同源程度,其成员又依次分为家族、亚家族和酶个体三级。细胞色素P450酶系统可缩写为CYP,其中家族以阿拉伯数字表示,亚家族以大写英文字母表示,酶个体以阿拉伯数字表示,如CYP2D6、CYP2C19、CYP3A4等。人类肝细胞色素P450酶系中至少有9种P450与药物代谢相关。
细胞色素P450主要分布在内质网和线粒体内膜上,研究表明细胞色素P450是药物代谢过程中的关键酶,而且对细胞因子和体温调节都有重要影响。
常见的机器学习算法举例
KNN实现鸢尾花分类
from sklearn.neighbors import KNeighborsClassifier from sklearn.datasets import make_moons from sklearn.model_selection import train_test_split from sklearn.metrics import accuracy_score iris = datasets.load_iris() iris_X = iris.data #导入数据 iris_y = iris.target #导入标签 X_train,X_test,y_train,y_test = train_test_split(iris_X,iris_y,train_size=0.3,random_state=20) model = KNeighborsClassifier() model.fit(X_train, y_train) # 训练 y_pred = model.predict(X_test) accuracy_score(y_pred, y_test) # 评估回归问题
这里使用的数据是美国波士顿的房价数据集。这个数据集有 13 个特征变量,目标变量是 5.0 ~ 50.0 的数值。简单起见,这里介绍一元回归的评估方法,所以我们只使用 13 个特征变量中的“住宅平均房间数”(列名为 RM )。from sklearn import datasets data = datasets.load_boston() X = data.data[:, [5,]] y = data.target from sklearn.linear_model import LinearRegression model_lir = LinearRegression() model_lir.fit(X, y) y_pred = model_lir.predict(X) print(model_lir.coef_) print(model_lir.intercept_) %matplotlib inline import matplotlib.pyplot as plt fig, ax = plt.subplots() ax.scatter(X, y, color='pink', marker='s', label='data set') ax.plot(X, y_pred, color='blue', label='LinearRegression') ax.legend() plt.show()
随机森林
from sklearn.datasets import load_wine from sklearn.ensemble import RandomForestClassifier from sklearn.model_selection import train_test_split from sklearn.metrics import accuracy_score # 读取数据 data = load_wine() X_train, X_test, y_train, y_test = train_test_split( data.data, data.target, test_size=0.3) model = RandomForestClassifier() model.fit(X_train, y_train) # 训练 y_pred = model.predict(X_test) accuracy_score(y_pred, y_test) # 评估
论文翻译
摘要:细胞色素P450酶(CYPs)影响药物代谢,CYPs抑制分类的预测模型基于成熟的机器学习方法。该模型在交叉验证和外部验证集上都得到了验证,并取得了良好的性能。
SuperCYPsPred
材料与方法
软件实现
SuperCYPsPred 数据存储在关系 MySQL 中数据库。RDKit 包 (http://www.rdkit.org/) 用于处理数据库中的化学信息。ChemDoodle Web 组件 (https://web.chemdoodle.com/),服务器中使用了用于化学接口的开源 JavaScript 库。网站后端是使用 PHP 和 Python 构建;Web 访问是通过Apache HTTP 服务器。Redis 用于排队和评估 API 请求 。服务器已经过测试在最新版本的 Mozilla Firefox、Google Chrome 上和 Apple Safari。
输入和输出
SuperCYPsPred 的用户界面易于使用自我信息。Web 服务器为用户提供四种方式
提交小分子。用户可以上传标准分子文件,分子名称,绘制分子或输入化合物的 SMILES(简化分子输入线输入系统)字符串。或者,用户可以选择不同的模型或所有模型进行预测,包括不同的分子指纹。结果显示在浏览器中的表格格式和雷达图,包括具有理化性质的分子结构以及训练集中最相似的三个分子这为该决定做出了最重要的贡献的CYP预测模型。用户可以在结果部分访问结果,以防预测结果无法立即显示。这些预测结果也是显示为比较平均置信度的雷达图每个训练集中活性化合物的分数模型,到输入化合物的模型。通过Web 服务器上可用的 DDI 检查器用户可以在给定组合时了解 DDI 的可能性的药物以及获取有关替代药物的信息可以用于相同的治疗效果。
图 1.用作应用案例的示例化合物(舍曲林)的图示。舍曲林是起始化合物;用户可以选择
MACCS 或 Morgan 指纹或两者兼而有之。在这种情况下,两者都被选中。显示的结果显示了细胞色素抑制曲线对于五种主要亚型。结果页面还包括有关类似化合物的信息,以及文献和整体雷达中报告的已知CYP情节。DDI 基质显示舍曲林的细胞色素-药物相互作用,当与药物(如西沙必利或布洛芬)联合使用时可以导致与副作用的主要交互(有关详细信息,请查看服务器上的 DDI 示例)。数据集
模型的训练集是从文献和两个不同的数据库中收集的。使用针对五种主要cyp亚型的体外生物发光试验进行定量高通量筛选的总共17143种受试物质来自PubChem Bioas- say数据库,.有关细胞色素P450酶的许多信息是使用我们的内部数据挖掘平台从文献中提取的,并存储在SuperCYP数据库中.该数据库目前包含1170种药物的信息,已知有3800多种相互作用。从最终数据集中移除了无机化合物、盐和混合物以及分类为非决定性的条目。对于五种色谱图中的每一种,将化合物分为训练集和外部验证集,保持活性物质(抑制剂)和非活性物质(非抑制剂)的比例恒定(参见S1补充数据)。
对超原型模型的评估
使用10倍CLUSTER交叉验证对每种模型进行了验证。使用不同的采样方法对数据进行分割,并保持所有折叠中活性和非活性物质的比例不变。
代码实现
数据集来源
PubChem (nih.gov)
下载初试文件
整理文件
删除无关信息,只保留这三行
只保留有活性和无活性
Inactive变成0,active变成1,根据5中不同类型分成5个表格
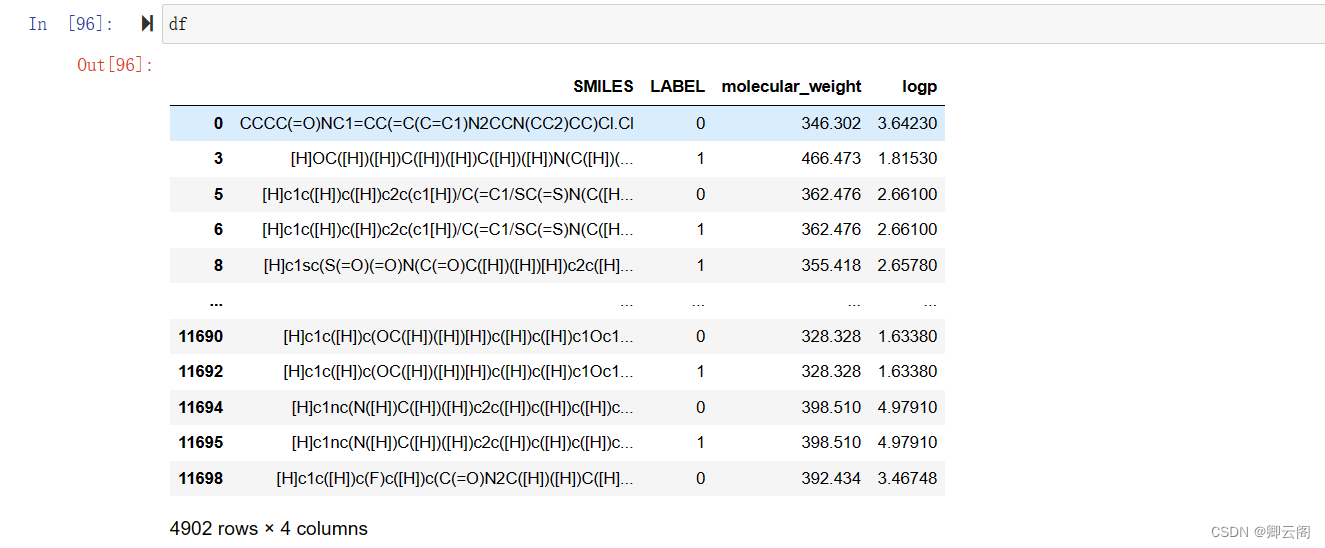
现在我们只研究p450-cyp3a4,修改名称,方便操作(SMILES,LABEL)
读取文件
import pandas as pd df = pd.read_csv('1.csv')
数据预处理
#删除空值 df=df.dropna()standardiser标准化分子
pip install -i https://pypi.tuna.tsinghua.edu.cn/simple standardiserfor i in df.index: try: smi = df.loc[i,'SMILES'] mol = Chem.MolFromSmiles(smi) mol = Chem.AddHs(mol) parent = standardise.run(mol) mol_ok_smi = Chem.MolToSmiles(parent) df.loc[i,'SMILES'] = mol_ok_smi print(i,'done') except standardise.StandardiseException as e: logging.warning(e.message)去重
df.drop_duplicates(inplace=True)计算分子量和logp
# 计算分子量 molweight = [] for smi in df['SMILES'].values: molweight.append(Descriptors.MolWt(Chem.MolFromSmiles(smi))) df['molecular_weight'] = molweight # 计算LogP logp = [] for smi in df['SMILES'].values: logp.append(Descriptors.MolLogP(Chem.MolFromSmiles(smi))) df['logp'] = logp
删除MW大于1000的分子保存清洗后的数据
df = df[df['molecular_weight'] <=1000] df.shape df.to_csv('2.csv',index=None)从SMILES列中的每个SMILES字符串中提取Morgan指纹,并将其添加到名为
fingerprint的新列中。建立模型完成预测from rdkit import Chem from rdkit.Chem import AllChem from rdkit import DataStructs import pandas as pd from sklearn.model_selection import train_test_split from sklearn.ensemble import RandomForestClassifier from sklearn.metrics import accuracy_score # 读取CSV文件 df = pd.read_csv('2.csv') # 提取分子指纹 fps = [] for smi in df['SMILES']: mol = Chem.MolFromSmiles(smi) fp = AllChem.GetMorganFingerprintAsBitVect(mol, 2, nBits=1024) # 使用Morgan指纹,半径为2,位数为1024 fps.append(fp) # 将分子指纹转换为数组形式 X = [list(fp.ToBitString()) for fp in fps] # 提取标签 y = df['LABEL'].values # 划分训练集和测试集 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) # 建立随机森林分类器模型 clf = RandomForestClassifier(n_estimators=100, random_state=42) # 训练模型 clf.fit(X_train, y_train) # 预测测试集 y_pred = clf.predict(X_test) # 计算准确率 accuracy = accuracy_score(y_test, y_pred) print('准确率:', accuracy)from rdkit import Chem from rdkit.Chem import MACCSkeys import pandas as pd from sklearn.model_selection import train_test_split from sklearn.svm import SVC from sklearn.metrics import accuracy_score # 读取CSV文件 df = pd.read_csv('2.csv') # 提取分子指纹 fps = [] for smi in df['SMILES']: mol = Chem.MolFromSmiles(smi) fp = MACCSkeys.GenMACCSKeys(mol) # 使用MACCS指纹 fps.append(fp) # 将分子指纹转换为数组形式 X = [list(fp.ToBitString()) for fp in fps] # 提取标签 y = df['LABEL'].values # 划分训练集和测试集 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) # 建立支持向量机分类器模型 clf = SVC(kernel='rbf', random_state=42) # 训练模型 clf.fit(X_train, y_train) # 预测测试集 y_pred = clf.predict(X_test) # 计算准确率 accuracy = accuracy_score(y_test, y_pred) print('准确率:', accuracy)from rdkit import Chem from rdkit.Chem import MACCSkeys import pandas as pd from sklearn.model_selection import train_test_split from sklearn.ensemble import RandomForestClassifier from sklearn.metrics import accuracy_score # 读取CSV文件 df = pd.read_csv('2.csv') # 提取分子指纹 fps = [] for smi in df['SMILES']: mol = Chem.MolFromSmiles(smi) fp = MACCSkeys.GenMACCSKeys(mol) # 使用MACCS指纹 fps.append(fp) # 将分子指纹转换为数组形式 X = [list(fp.ToBitString()) for fp in fps] # 提取标签 y = df['LABEL'].values # 划分训练集和测试集 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42) # 建立随机森林分类器模型 clf = RandomForestClassifier(n_estimators=100, random_state=42) # 训练模型 clf.fit(X_train, y_train) # 预测测试集 y_pred = clf.predict(X_test) # 计算准确率 accuracy = accuracy_score(y_test, y_pred) print('准确率:', accuracy)
【bioinformation 10】ADMET-CYPs抑制剂预测实战
news2026/2/16 23:22:12
本文来自互联网用户投稿,该文观点仅代表作者本人,不代表本站立场。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如若转载,请注明出处:http://www.coloradmin.cn/o/1526193.html
如若内容造成侵权/违法违规/事实不符,请联系多彩编程网进行投诉反馈,一经查实,立即删除!相关文章
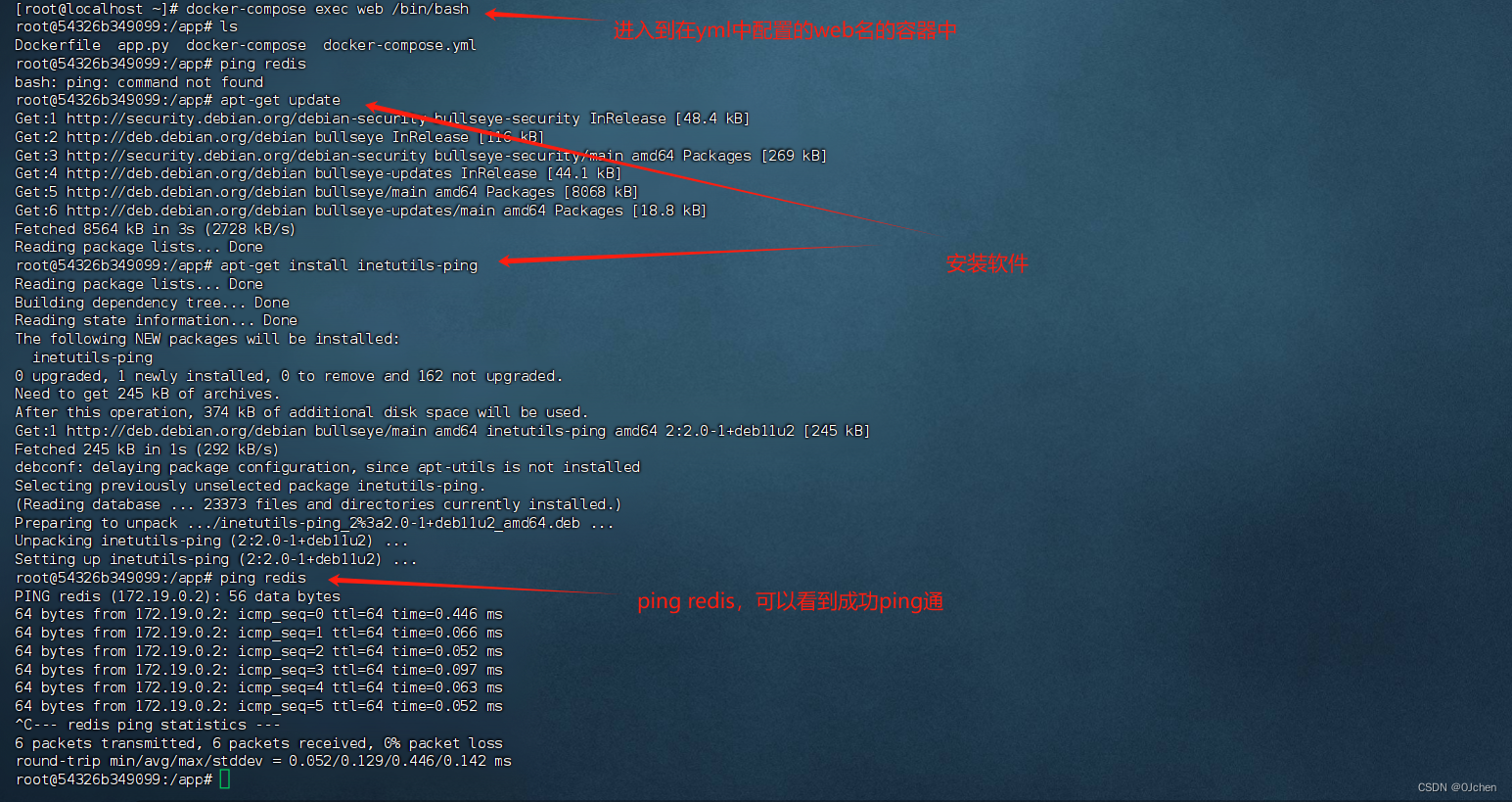
/usr/local/bin/docker-compose: line 1: Not: command not found
安装docker-compose 检查是否安装成功 docker-compose --version 出错
/usr/local/bin/docker-compose: line 1: Not: command not found 检查下载连接是否正确
官网 https://dockerdocs.cn/compose/install/ 根据官网上连接下载 发现下载不了 在版本前加个V 就可以解决 版…
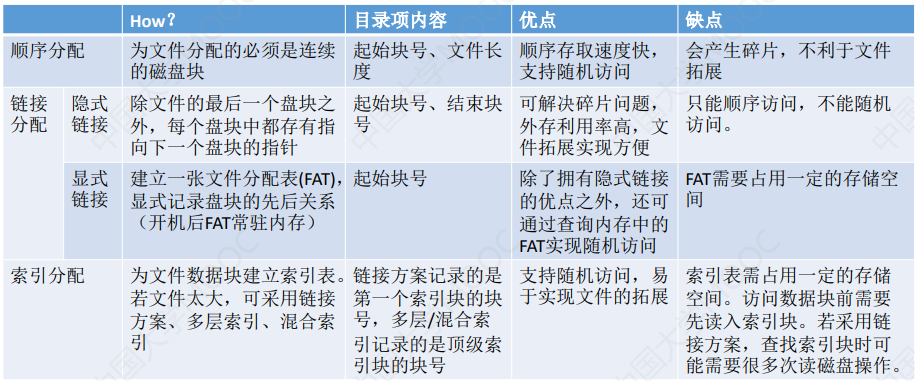
4.1_4 文件的物理结构
文章目录 4.1_4 文件的物理结构(一)文件块、磁盘块(二)文件分配方式——连续分配(三)文件分配方式——链接分配(1)链接分配——隐式链接(2)链接分配——显式链…
JETSON 配置并跑通 NanoDet
JETSON 配置 NanoDet 文章目录 JETSON 配置 NanoDetNanoDet 介绍源码环境搭建及测试配置 NanoDet 的环境环境配置过程中遇到的问题:环境配置完毕验证 NanoDet NanoDet 介绍
可以参考这个博客:NanoDet:这是个小于4M超轻量目标检测模型
源码 …
什么是网站?为什么要搭建网站?
网站:简单来说,网站就是通过互联网来展示信息的页面集合。它可以在电脑或者手机上打开,提供各种功能,比如查看新闻、购买商品、搜索信息等。 一、建网站的目的:展示个人或企业的存在 网站建设的首要目的之一是展示个人…
23-分支和循环语句_习题练习
1、转换以下ASClI码为对应字符并输出他们:73,32,99, 97,110,32,100,111,32,105,116,33
输入:无
输出:一行输出转换题目中给出的所有ASClI码对应的字符,无需以空格隔开。
输入:
int main()
{int i 0;int arr[] { …
加拿大光量子计算公司Xanadu入局英国多企业量子合作计划
内容来源:量子前哨(ID:Qforepost) 编辑丨慕一 编译/排版丨沛贤
深度好文:1200字丨8分钟阅读
英国航空发动机制造商罗尔斯罗伊斯(Rolls-Royce)、英国量子计算公司Riverlane和加拿大量子计算公…
【Nutx3】middleware目录介绍
简言
记录下nuxt3middleware目录的使用方法。
middleware
middleware是存放路由中间件的文件目录。 路由中间件有三种:
匿名(或内联)路由中间件直接在页面中定义。已命名的路由中间件,放在 middleware/ 中,页面使用…
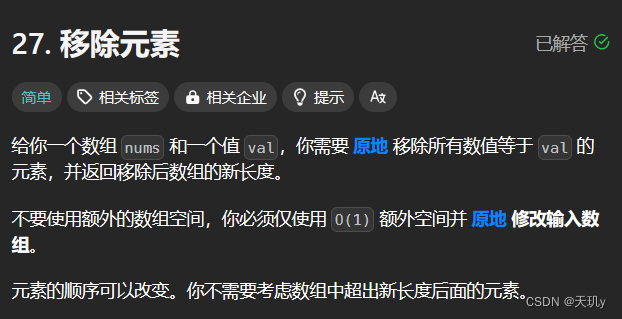
leetcode代码记录(移除元素
目录 1. 题目:2. 我的代码:小结: 1. 题目: 给你一个数组 nums 和一个值 val,你需要 原地 移除所有数值等于 val 的元素,并返回移除后数组的新长度。
不要使用额外的数组空间,你必须仅使用 O(1)…
Docker入门二(应用部署、迁移与备份、DockerFile、docker私有仓库、Docker-Compose)
文章目录 一、应用部署1.MySQL部署2.Redis部署3.Nginx部署 二、迁移与备份1.容器做成镜像2.镜像备份和恢复(打包成压缩包) 三、DockerFile0.镜像从哪里来?1.什么是DockerFile2.DockerFile 构建特征3.DockerFile命令描述4.构建一个带vim的centos镜像案例5…
网安渗透攻击作业(4)
Unload-labs-01 function checkFile() { var file document.getElementsByName(upload_file)[0].value; if (file null || file "") { alert("请选择要上传的文件!"); return false; } //定义允许上传的文件类型 v…
面渣逆袭:Java基础面试题,46道Java基础八股文(4.8万字,30+手绘图)
1、什么是 Java? Java是一种广泛使用的编程语言,由Sun Microsystems(现为Oracle Corporation的一部分)在1995年首次发布。它是一种面向对象的语言,这意味着它支持通过类和对象的概念来构造程序。 Java设计有一个核心理…
软件工程-第5章 结构化设计
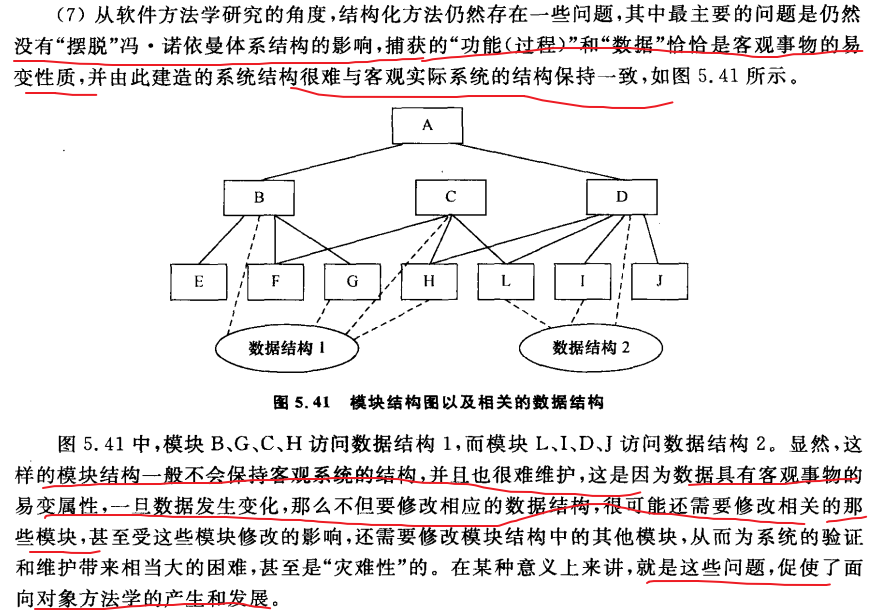
5.1 总体设计的目标及其表示方法 5.2 总体设计 变换设计基本步骤:
第1步:设计准备--复审并精华系统模型;
第2步:确定输入、变换、输出这三部分之间的边界;
第3步:第一级分解--系统模块结构图顶层和第一层…
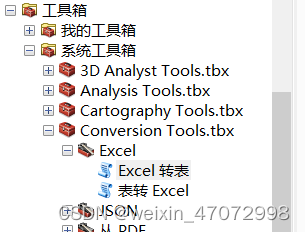
arcgis数据导出到excel
将arcgis属性数据导出到excel:
1) 工具箱\系统工具箱\Conversion Tools.tbx\Excel\Excel 转表 2)用excel打开导出的图层文件中后缀为.dbf的数据(方便快捷,但是中文易乱码)
GPT-5:人工智能的下一个前沿即将到来
当我们站在人工智能新时代的门槛上时,GPT-5即将到来的呼声愈发高涨且迫切。作为革命性的GPT-3的继任者,GPT-5承诺将在人工智能领域迈出量子跃迁式的进步,其能力可能重新定义我们与技术的互动方式。
通往GPT-5之路
通往GPT-5的旅程已经标记着…
鸿蒙Harmony应用开发—ArkTS声明式开发(容器组件:FolderStack)
FolderStack继承于Stack(层叠布局)控件,新增了折叠屏悬停能力,通过识别upperItems自动避让折叠屏折痕区后移到上半屏 说明: 该组件从API Version 11开始支持。后续版本如有新增内容,则采用上角标单独标记该内容的起始版本。 子组件…
探秘atoi与atof的模拟之路:从原理到实践的全能指南!
目录 编辑
一.atoi及atof库函数的工作原理
1.1atoi
1.2atof
1.3使用时的注意事项
注意事项
1. 检查输入字符串是否为 NULL
2. 检查字符串是否仅包含有效的数字字符
3. 检查转换结果是否在预期范围内
4. 使用更健壮的替代函数
二. 模拟实现atoi和atof
2.1模拟 atoi…
罗马不是一天建成的:DevOps 转型分步指南
“罗马不是一天建成的”这句格言非常适合 DevOps。许多公司都渴望通过“闪电战”获得市场主导地位的即时满足感,但DevOps 的真正成功是一场马拉松,而不是短跑。它需要致力于建立可持续的 DevOps 文化——一种从头开始促进协作、自动化和安全性的文化。
…
计算机设计大赛 题目:基于深度学习的中文对话问答机器人
文章目录 0 简介1 项目架构2 项目的主要过程2.1 数据清洗、预处理2.2 分桶2.3 训练 3 项目的整体结构4 重要的API4.1 LSTM cells部分:4.2 损失函数:4.3 搭建seq2seq框架:4.4 测试部分:4.5 评价NLP测试效果:4.6 梯度截断…
Linux编程4.8 网络编程-建立连接
1、服务器端 #include <sys/types.h> #include <sys/socket.h>int listen(int sockfd, int backlog);返回:成功返回0,出错返回-1。参数:sockfd:套接字的文件描述符backlog:定义了sockfd的挂起连接队列可能增长的最大长度。…
使用 ZipArchiveInputStream 读取压缩包内文件总数
读取压缩包内文件总数
简介
ZipArchiveInputStream 是 Apache Commons Compress 库中的一个类,用于读取 ZIP 格式的压缩文件。在处理 ZIP 文件时,编码格式是一个重要的问题,因为它决定了如何解释文件中的字符数据。通常情况下,Z…