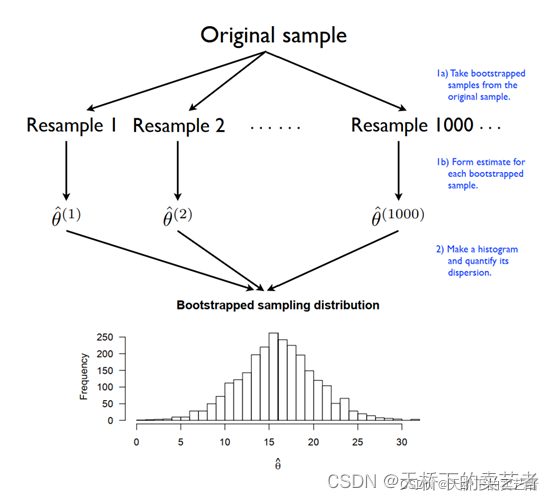
bootstrap自采样目前广泛应用与统计学中,其原理很简单就是通过自身原始数据抽取一定量的样本(也就是取子集),通过对抽取的样本进行统计学分析,然后继续重新抽取样本进行分析,不断的重复这一过程N(大于500次以上)次,然后得到N个统计结果,然后进行区间分析,得到最终结果。
上一章我们简单介绍了BOOT重抽样获取回归方程系数95%可信区间,可能大家对BOOT重抽样的用处感觉还不是很明显。BOOT重抽样在我们统计中处理数据还是很有用的,本期我们来介绍一下怎么使用BOOT重抽样获取cox回归方程C-index(C指数)可信区间,这也是一个粉丝向我问的问题,我觉得蛮有典型性和实用性的,因此就拿出来讲讲。首先我们看看什么是C-index(C指数),C-index,C指数即一致性指数(concordance index),用来评价模型的预测能力。c指数是指所有病人对子中预测结果与实际结果一致的对子所占的比例。我们在既往的文章《手把手教你使用R语言建立COX回归并画出列线图(Nomogram)》中已经介绍了怎么计算C指数,今天我们继续以原来文章的数据和方法为例进行视频演示。
R语言使用BOOT重抽样获取cox回归方程C-index(C指数)可信区间
代码
library(survival)
library(rms)
library(boot)
bc<-cancer
bc <- na.omit(bc)
# inst: 机构代码,time: 以天为单位的生存时间,status: 状态:审查状态 1=审查,2=死亡,
# age: 年龄,sex: 男=1 女=2,ph.ecog:由医师评定的 ECOG 表现评分。
# ph.karno:由医师评定的 Karnofsky 表现评分(差=0-好=100),pat.karno:由患者评定的 Karnofsky 性能评分
# ,meal.cal:用餐时消耗的卡路里,wt.loss:过去六个月的体重减轻
bc$sex<-as.factor(bc$sex)
f <- cph(Surv(time, status) ~ age + sex + ph.ecog + pat.karno +wt.loss,
x=T, y=T, surv=T, data=bc)
rcorrcens(Surv(time, status) ~ predict(f), data = bc)
#C.index
C.index=1-0.344
##粉丝提供的
c_index <- function(formula, data, indices) {
tran.data <- data[indices,]
vali.data <- data[-indices,]
fit <- coxph(formula, data=tran.data)
result<-survConcordance(Surv(vali.data$time,vali.data$death)~predict(fit,vali.data))
index<-as.numeric(result$concordance)
return(index)
}
##我改良的
c_index <- function(data,indices){
dat <- data[indices,]
fit<- cph(Surv(time, status) ~ age + sex + ph.ecog + pat.karno +wt.loss,
x=T, y=T, surv=T, data=bc)
pr1<-predict(fit,newdata=dat)
Cindex=rcorrcens(Surv(time, status) ~ pr1, data =dat)[1]
Cindex=1-Cindex
Cindex
}
#调试一下
c_index(bc,1:100)
###
results <- boot(data=bc, statistic=c_index, R=500)
#如果你想查看每个抽样的结果
results$t
##抽样分布
plot(results)
#计算可信区间
boot.ci(results,conf = 0.95)
#( 0.5977, 0.7104 )