前言:
最近在阅读文献的时候发现2篇文章,是采用T1w/T2w 比值表征髓磷脂,有点感兴趣,因此尝试了一下文献所提出的方法。(https://www.ncbi.nlm.nih.gov/pmc/articles/PMC9247578/ https://www.ncbi.nlm.nih.gov/pmc/articles/PMC10028664/) 髓鞘形成是皮层下白质的一个显著特征,但大脑皮层的灰质也包含大量髓鞘轴突,深层皮质的髓鞘磷脂密度高于浅层皮质层的。先前的研究表明,T1w/T2w比率的区域差异与组织学定义的皮质区域的髓磷脂含量匹配,并增强对髓鞘信号强度的敏感性,同时减少受试者间信号强度偏差,可以用作检测与疾病相关的髓鞘形成程度变化。
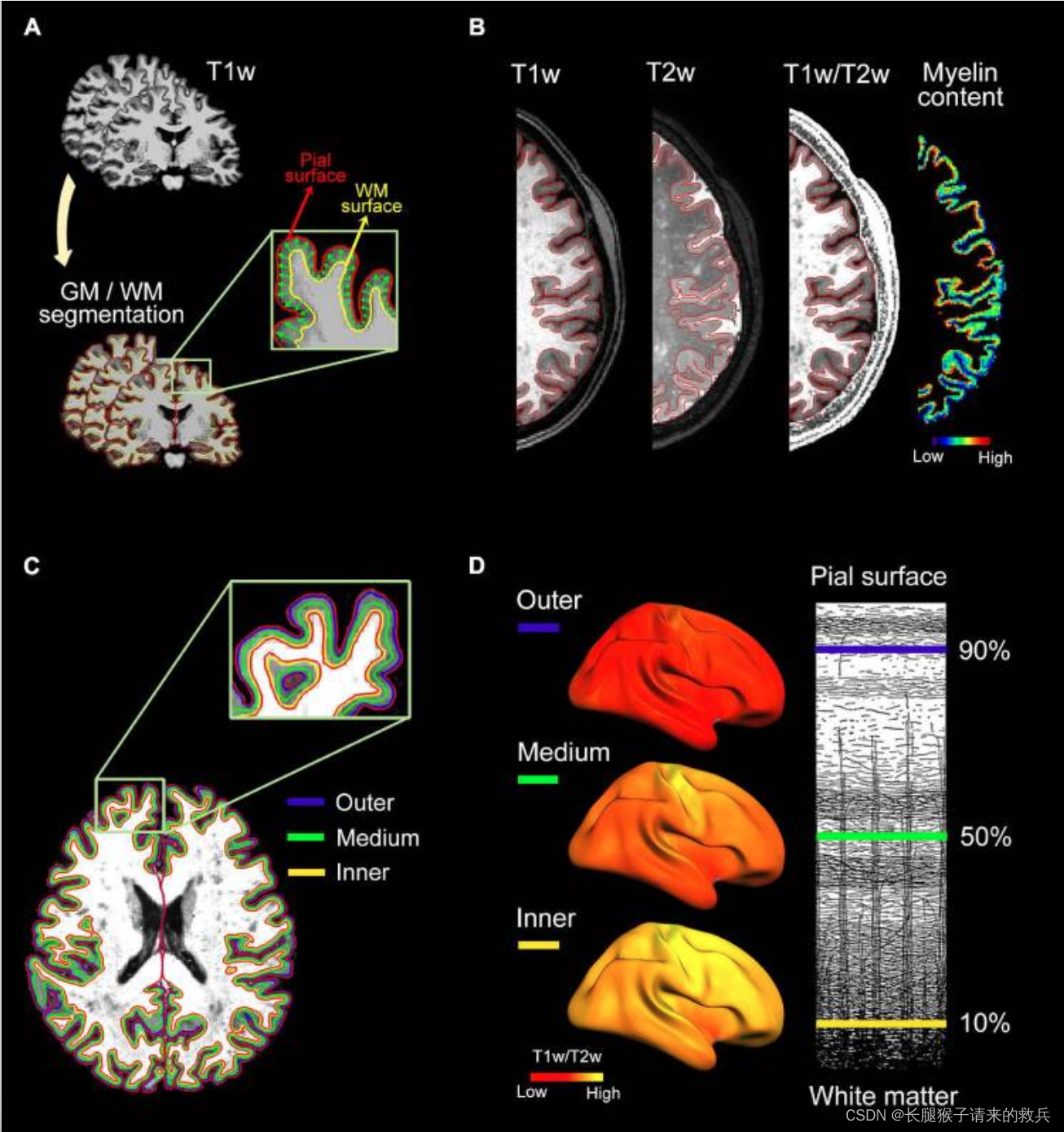
根据文献的流程图,可以把整个处理过程分成4部分,分别是freesurfer分割T1像,将T2像配准到T1像得到T1w/T2w像,对T1w/T2w像进行部分容积效应校正(Partial Volume Effect Correction, PVEC),将校正后的T1w/T2w投射到平均皮质表面fsaverage
一、分割T1像
参考之前的freesurfer部分的博客
export SUBJECTS_DIR=/home/brainer/YSY/FCD/recon
ls *nii |parallel --jobs 20 recon-all -s {.} -i {} -all -qcache #并行处理
二、将T2像配准到T1像
这里采用的是freesurfer的bbregister命令
https://surfer.nmr.mgh.harvard.edu/fswiki/bbregister

export SUBJECTS_DIR=/home/brainer/YSY/FCD/recon
cd /home/brainer/YSY/FCD/recon/sub001
mkdir T2
cd T2
bbregister --s sub001 --mov $T2path/T2w.nii.gz --t2 --reg T2w2T1.dat --o T2w2T1.nii.gz --fslmat T2w2T1.mat #配准
freeview -viewport cor -v T2w2T1.nii.gz --surface ../surf/lh.white:edgecolor=yellow --surface ../surf/rh.white:edgecolor=yellow
#检查配准
cd ../mri
mri_convert T1.mgz ../T2/T1.nii.gz
cd ../T2
mris_calc --output T1divT2.nii.gz T1.nii.gz div T2w2T1.nii #获得T1w/T2w像
三、部分容积效应校正
这里采用的是freesurfer的mri_gtmpvc命令
https://surfer.nmr.mgh.harvard.edu/fswiki/PetSurfer
#Partial Volume Correction
mkdir -p sub001/T2/pvc
export SUBJECTS_DIR=/home/brainer/YSY/FCD/recon
cd ..recon/sub001/T2
gtmseg --s sub001
mri_coreg --s sub001 --mov T1divT2.nii.gz --reg T1divT2.reg.lta
mri_gtmpvc --i T1divT2.nii.gz --reg T1divT2.reg.lta --psf 0 --seg gtmseg.mgz \
--mgx .01 --o sub001/pvc/pvcgtmpvc.output --opt 3 --save-input --no-rescale --threads 3
四、投射到平均皮质表面fsaverage
export SUBJECTS_DIR=/home/brainer/YSY/FCD/recon
cd ..recon/sub001/T2
mri_vol2surf --mov mgx.ctxgm.nii.gz --reg aux/bbpet2anat.lta --hemi lh \
--projfrac 0.5 --o lh.mgx.ctxgm.fsaverage.sm00.nii.gz --cortex --trgsubject fsaverage
#投射到软脑膜和白质表面中间层(50%)
#--projfrac-avg .1 .9 .4 投射到软脑膜和白质表面中间层(10%,50%,90%)
五、后续可根据需求进行后续的分析
- Surface-based analysis
- Subcortical volume-based analysis
- mSCN network analysis




![[职场] 应聘销售的简历怎么写 #职场发展#笔记](https://img-blog.csdnimg.cn/img_convert/25791407876f5f98a5bdcf792c49ce2c.jpeg)





![[OPEN SQL] 删除数据](https://img-blog.csdnimg.cn/direct/07afe4f4e12948bd98921590d2ef57a8.png)