2023年10月9日,诺华公司的Lingling Shen和He Wang在Chemrxiv上发表了文章《Pocket Crafter: A 3D Generative Modeling Based Workflow for the Rapid Generation of Hit Molecules in Drug Discovery》,介绍了他们分子生成在hit finding项目应用中的pipeline: Pocket Crafter,及其实际应用效果。

Pocket Crafter以Pocket2Mo作为3D的分子生成工具,然后经过化学物理的过滤器,类药过滤器,以及SAR富集,对top的分子进行骨架聚类,获得虚拟的多样性骨架。
在他们的WDR5体系中,基于获得的虚拟骨架在诺华库中进行有针对性的搜索后,获得了一组重点关注的 2029 个化合物。经过试验测定,获得了一个新颖的活性分子。
Pocket Crafter 成功构建了一个有效的端到端 3D 生成分子的实际应用的工作流程原型,用于探索新的化学骨架,代表了早期药物发现中识别新型活性化合物的一种有前途的方法。
这也是分子生成AI方法新的应用模式。
1. Pocket Crafter流程
Pocket Crafter的流程结构如下图:
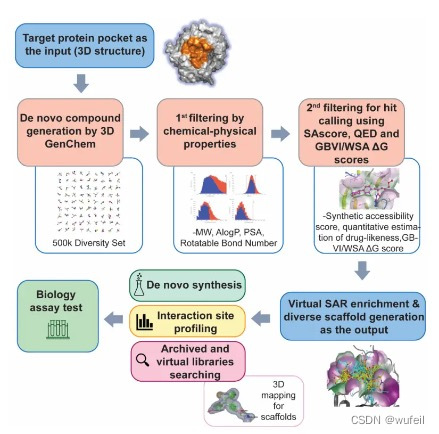
首先,将蛋白的三级结构作为输入,利用Pocket2Mol模型,3D生成binder。
然后将生成的分子经过物理-化学过滤器,然后是hit过滤器(QED,可合成性SA,与口袋的结合力等),然后进行虚拟的SAR分析,提取多样性骨架。
Pocket Crafter最终输出的是:能结合在口袋上的多样性的分子骨架;对应的相互作用模式;虚拟预测的可合成性,可用在化合物库中检索的分子。
1.1 Pocket2Mol生成分子
使用github中的Pocket2Mol代码,每次采样生成2000个分子,重复进行300次,每次使用不同的随机种子。
最终,获得了50W个的de novo的多样性的虚拟分子。
可想而知,虽然是在口袋条件下,但是生成的分子重复率仍然很低(1-50w/2000/300,约为17%),可见适合一个口袋的化学空间是多巨大的,对如此巨大的化学空间的检索,显然不是人能做的,只有CADD和AIDD才能完成。
案例中,WDR5 WBM口袋已知小分子binder的如下图(已知的binder,作为口袋参考):

1.2 化学物理性质过滤
使用Pipeline Pilot软件,包括:校验分子,保证分子的原子类型,化学键,电荷正确。然后计算分子的分子质量,AlogP,分子极性表面积,可旋转键数量,按照“Molecular_weight < 800, AlogP < 7, AlogP > -1, Molecular_PolarSurfaceArea (Å2 ) < 125, and Num_RotatableBonds < 12”的条件,进行过滤。
1.3 Hit相关的性质过滤
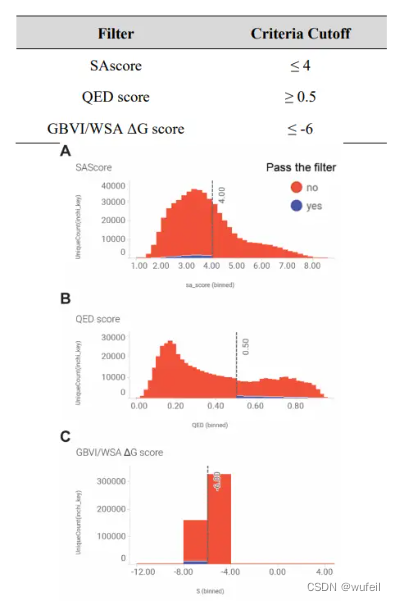
将能通过上述化学物理性质filter的分子,使用rdkit计算QED,SA分数。仅保留SA_score<4, QED>=0.5的分子。
使用“Bemis-Murcko Assemblies”片段化方法,用 Murcko 片段 SMILES 片段化并注释所有分子(即,提取骨架)。
随后,基于Murcko SMILES的ECFP_6描述符,使用最大相异性方法选择聚类中心,最大距离为0.625,对分子进行聚类。
可惜作者没有给出最后产生了多少个类别,一共有多少分子通过了这些filters(文章中没有具体指明数量,预印本有卖描述错误)。
化学物理性质过滤器以及Hit相关性质过滤的Pipeline Pilot流程如下图:

将能通过上述filters的分子,使用MOE计算GBVI/WSA ΔG 分数,评估pocket2Mol生成构象与口袋的结合自由能。
注意,之前使用docking进行构象重新生成,docking使用默认参数,分子使用QuickPrep快速准备,晶体结构中的原来的小分子作为对接中心。
注意,这里并没有使用docking分数作为阈值打分。
1.4 虚拟 SAR 富集和多样性骨架生成
将能经过上述两步filters的分子,称为虚拟的hits(virtual hits)。
关于GBVI/WSA ΔG 分数,作者在流程中会尝试使用不同的阈值,从-6开始,每间隔0.01逐个尝试。
作者是使用了Fisher's 抽取测试,按照每一个分子是否是hit,以及是否属于某个类别,进行P值统计。
将P值<=0.005的类的Top N个分子作为Hit SAR 骨架。
虚拟 SAR 富集分析使能够识别哪些化学基团和结构最有可能成为口袋的真正binder,其动态 SAR 范围适合药物化学家进一步优化。
1.5 分子库的检索
经过SAR富集的分子数量还是很多,考虑到合成的复杂性,要直接合成也是不可能的。作者使用检索内部分子库的方式。
他们使用的诺华内部的3~4百万的多样性的分子库,以及100亿级别的按需合成的Enamine库。使用ROCS (Rapid Overlay of Chemical Structures)以及机器学习的分子检索工具,基于形状,静电,药效团,以及其他的2D,3D的特征,从分子库中检索分子。
作者将之前选出的top 100-200个虚拟hit,使用ROCS工具,利用Tanimoto Combo score 阈值为1.0,从诺华内部的多样性分子库中检索分子。流程如下图:

将检索到的分子,重新计算GBVI/WSA ΔG score,使用之前的阈值,做筛选,同时这些分子还要经过诺华内部的溶解度预测机器学习模型。
注意,上述工作,pocket2mol使用了10个节点,CPU部分使用了1-200个节点,花费了1个星期时间。这个算力,还真不是一般公司可以提供的。
随后,会经过QC质量检测,HTRF活性检测,DSF检测,已获得筛选出来分子的活性。
2. 实验结果
pocket2mol一共生成了543,491个有效分子。
作者比较了pocket2mol生成的分子与诺华内部多样分子库的化学空间,pocket2mol生成的分子多样性要更大一些,也许这就是AIDD分子生成的意义,遍历了更大的化学空间。如下图。

经过物理化学filters的分子一共有352,820个。相关性质分布如下图:
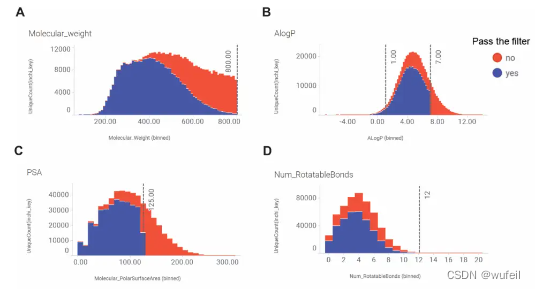
352,820个分子类hit性质如下图。经过类hit filters(SA分数,QED分数等)以后,共计只有9,531。分子数量在类药filters大规模下降了。
毕竟对于现在的AI模型来说,生成分子的类药性真的是一个硬伤。
经过SAR富集分析和分子库检索以后,考虑溶解度,分子库中质量,选择了2029个分子进行实验测试,测试结果如下图。
湿实验获得7个阳性hit,对应的阳性率为0.345%,Hit Rate较HTRF分子库直接实验筛选,提升了12.8倍。


在7个阳性hit中,三个分子的IC50分别为35.6, 27.5 and 28.5 μM,他们的WDR5-MYC HTRF dose-response curve (DRC)如下图,对照实验结果表明这三个分子影响了WDR5 and MYC 相互作用。
DSF实验有明显温度漂移,也暗示着这些小分子是结合在WDR5蛋白上。
同时,对接分析结果表明,这些分子的结合模式与已知的binder结合模式不相同(对接结果,没有晶体)。

但是,基于Pocket Crafter流程筛选出来的活性hit,活性要低于之前湿实验的结果。但是,不管怎么说,获得了一个之前湿实验没有获得新骨架。
3. 总结
在换句话说,Pocket Crafter代表了AIDD领域的分子生成的新应用场景。
以往,AI生成的分子往往被抱着怀疑的态度,因为生成的分子在被合成不是每一个都有活性,确切的说,是几乎都没有活性,或者活性远低于参考分子。这是药化团队或者公司成本是不能接受的,因此,分子生成的工业应用,非常困难。
Pocket Crafter的例子呢,则提供了一种新的分子生成应用方式,用于生成新的骨架,然后再去检索分子库。
似乎,直接检索分子库做docking更好一些?当然,也有可能在这个体系中,docking无法识别分子活性,甚至分类都做不出来。这估计也是为什么在流程中,作者没有使用docking打分作为阈值的原因。