一、问题假设
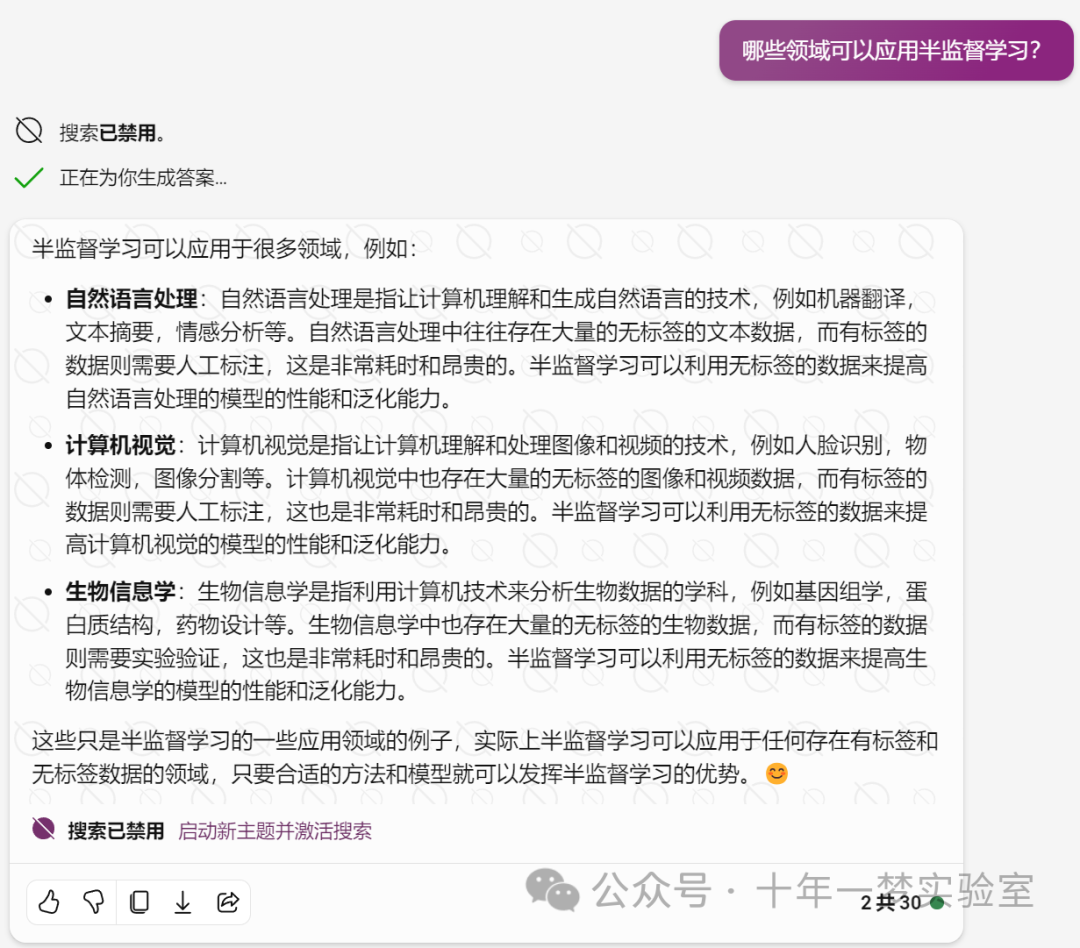
要利用无标签样本进行训练,必须对样本的分布进行假设?


二、启发式算法



自训练和协同训练是两种常用的半监督学习的方法,它们的主要区别在于使用的模型的数量和类型。
自训练:自训练是一种使用单个模型的半监督学习的方法,它的过程是先用有标签的数据训练一个初始的模型,然后用这个模型对无标签的数据进行预测,选择一些预测结果最有信心的数据作为新的有标签的数据,加入到原来的有标签的数据集中,再用这个扩充的数据集训练一个新的模型,重复这个过程直到满足某个终止条件。自训练的优点是简单和高效,但是缺点是可能会放大模型的初始偏差,导致错误的标签传播。
协同训练:协同训练是一种使用多个模型的半监督学习的方法,它的过程是先用有标签的数据训练两个或多个不同的模型,然后用这些模型分别对无标签的数据进行预测,选择一些预测结果最有信心且不一致的数据作为新的有标签的数据,分别加入到对应的有标签的数据集中,再用这些扩充的数据集训练新的模型,重复这个过程直到满足某个终止条件。协同训练的优点是能够利用模型之间的多样性和互补性,减少错误的标签传播,但是缺点是需要设计合适的模型和选择合适的数据,否则可能会降低协同训练的效果。
三、生成模型



使用高斯混合模型的半监督学习生成模型的原理


这是一个半监督学习的示例,使用高斯混合模型(GMM)来对数据进行聚类和分类
# 导入numpy库,用于进行数值计算和矩阵操作
import numpy as np
# 导入sklearn库中的make_blobs函数,用于生成模拟数据
from sklearn.datasets import make_blobs
# 导入sklearn库中的GaussianMixture类,用于创建和训练GMM模型
from sklearn.mixture import GaussianMixture
# 生成数据,其中有三个类别,每个类别有两个特征
# X是一个1000行2列的矩阵,表示1000个数据点的特征值
# y是一个长度为1000的向量,表示每个数据点的真实类别
X, y = make_blobs(n_samples=1000, n_features=2, centers=3, random_state=0)
# 随机选择一部分数据作为标记数据,其余的作为未标记数据
# n_labeled是标记数据的数量,这里设为100
n_labeled = 100
# indices是一个长度为1000的向量,表示对数据点的随机排列
indices = np.random.permutation(X.shape[0])
# X_labeled是一个100行2列的矩阵,表示标记数据的特征值
X_labeled = X[indices[:n_labeled]]
# y_labeled是一个长度为100的向量,表示标记数据的真实类别
y_labeled = y[indices[:n_labeled]]
# X_unlabeled是一个900行2列的矩阵,表示未标记数据的特征值
X_unlabeled = X[indices[n_labeled:]]
# 用标记数据来初始化GMM的参数,假设有三个高斯分布
# gmm是一个GaussianMixture对象,表示GMM模型
gmm = GaussianMixture(n_components=3, random_state=0)
# gmm.means_init是一个3行2列的矩阵,表示GMM模型的初始均值
# 这里用标记数据的均值来初始化GMM模型的均值
gmm.means_init = np.array([X_labeled[y_labeled == i].mean(axis=0) for i in range(3)])
# gmm.fit是一个方法,用于根据标记数据来训练GMM模型
# 这里用标记数据的特征值和类别来训练GMM模型
gmm.fit(X_labeled, y_labeled)
# 用未标记数据来更新GMM的参数,用EM算法来迭代优化
# gmm.fit是一个方法,用于根据未标记数据来更新GMM模型
# 这里用未标记数据的特征值来更新GMM模型
gmm.fit(X_unlabeled)
# 用更新后的GMM来计算每个数据点的后验概率
# probs是一个1000行3列的矩阵,表示每个数据点属于每个高斯分布的后验概率
# gmm.predict_proba是一个方法,用于根据GMM模型来计算后验概率
# 这里用GMM模型和所有数据的特征值来计算后验概率
probs = gmm.predict_proba(X)
# 用后验概率来对数据进行分类,将数据分配给后验概率最大的高斯分布所对应的类别
# y_pred是一个长度为1000的向量,表示每个数据点的预测类别
# probs.argmax是一个方法,用于沿着指定的轴返回最大值的索引
# 这里用后验概率的最大值所在的列的索引来表示预测类别
y_pred = probs.argmax(axis=1)
# 计算分类的准确率
# accuracy是一个浮点数,表示预测类别和真实类别的一致比例
# np.mean是一个函数,用于计算平均值
# 这里用预测类别和真实类别的相等情况的平均值来表示准确率
accuracy = np.mean(y_pred == y)
# print是一个函数,用于输出结果
# 这里用字符串格式化的方法,将准确率保留两位小数并输出
print(f"Accuracy: {accuracy:.2f}")输出:Accuracy: 0.92
四、低密度分割
半监督支持向量机

SSVM

# 导入相关的库
import numpy as np
from sklearn.datasets import make_blobs
from sklearn.svm import SVC
# 生成数据,其中有三个类别,每个类别有两个特征
X, y = make_blobs(n_samples=1000, n_features=2, centers=3, random_state=0)
# 随机选择一部分数据作为标记数据,其余的作为未标记数据
n_labeled = 100
indices = np.random.permutation(X.shape[0])
X_labeled = X[indices[:n_labeled]]
y_labeled = y[indices[:n_labeled]]
X_unlabeled = X[indices[n_labeled:]]
# 用标记数据来训练一个初始的SVM分类器,假设有三个类别
svm = SVC(C=1.0, kernel='rbf', gamma='auto', probability=True)
svm.fit(X_labeled, y_labeled)
# 用未标记数据来改进SVM分类器,用自训练的方法
n_iter = 10 # 迭代次数
n_add = 10 # 每次迭代添加的数据个数
for i in range(n_iter):
# 用SVM分类器来对未标记数据进行预测,得到每个数据点属于每个类别的概率
probs = svm.predict_proba(X_unlabeled)
# 选择预测结果最有信心的一部分数据,即概率最大的数据
max_probs = probs.max(axis=1)
max_indices = max_probs.argsort()[-n_add:]
# 将这些数据加入到标记数据中,用其预测的类别作为标签
X_labeled = np.concatenate([X_labeled, X_unlabeled[max_indices]])
y_labeled = np.concatenate([y_labeled, probs[max_indices].argmax(axis=1)])
# 用这些数据来重新训练SVM分类器
svm.fit(X_labeled, y_labeled)
# 从未标记数据中移除这些数据
X_unlabeled = np.delete(X_unlabeled, max_indices, axis=0)
# 用改进后的SVM分类器来对所有数据进行分类
y_pred = svm.predict(X)
# 计算分类的准确率
accuracy = np.mean(y_pred == y)
print(f"Accuracy: {accuracy:.2f}") # 输出 0.9TSVM Transductive SVM

import numpy as np
import sklearn.svm as svm
import time
class TransductiveSVM(svm.SVC):
def __init__(self,kernel="rbf",Cl=1,Cu=0.01,gamma=0.1,X2=None):
'''
Initial TSVM
Parameters
----------
kernel: kernel of svm
Cl: Penalty Inductive SVM
Cu: Penalty Unlabeled set
gamma: gamma for rbf kernel
X2: Unlabeled set(only features)
np.array, shape:[n2, m], n2: numbers of samples without labels, m: numbers of features
'''
self.Cl=Cl
self.Cu=Cu
self.kernel = kernel
self.gamma=gamma
self.clf=svm.SVC(C=self.Cl,kernel=kernel,gamma=self.gamma,probability=True)
self.Yresult=None
self.X2=X2
def fit(self, X1, Y1):
'''
Train TSVM by X1, Y1, X2(X2 is passed on init)
Parameters
----------
X1: Input data with labels
np.array, shape:[n1, m], n1: numbers of samples with labels, m: numbers of features
Y1: labels of X1
np.array, shape:[n1, ], n1: numbers of samples with labels
'''
t=time.time()
X2=self.X2
Y1[Y1!=+1]=-1
Y1 = np.expand_dims(Y1, 1)
ratio=sum(1 for i in Y1 if i==+1)/len(X1)
num_plus=int(len(X2)*ratio) #number of positive example as describe in joachims svm
N = len(X1) + len(X2)
sample_weight = np.zeros(N)
sample_weight[:len(X1)] = self.Cl
self.clf.fit(X1, Y1,sample_weight=sample_weight[:len(X1)]) #classify the num_plus examples with the highest value with +1, other -1
Y2=np.full(shape=self.clf.predict(X2).shape,fill_value=-1)
Y2_d = self.clf.decision_function(X2)
index=Y2_d.argsort()[-num_plus:][::-1]
for item in index:
Y2[item]=+1
#INIT CMINUS E CLUS
#C_minus=.00001
C_minus=.00001
C_plus=.00001*(num_plus/(len(X2)-num_plus))
for i in range(len(Y2)):
if(Y2[i]==+1):
sample_weight[len(X1)+i]=C_plus
else:
sample_weight[len(X1)+i]=C_minus
Y2 = np.expand_dims(Y2, 1)
X3 = np.vstack((X1, X2))
Y3 = np.vstack((Y1, Y2))
k=0
while (C_minus<self.Cu or C_plus<self.Cu): #LOOP 1
self.clf.fit(X3, Y3, sample_weight=sample_weight)
Y3 = Y3.reshape(-1)
#slack=Y3*(self.clf.decision_function(X3))
slack = Y3*self.clf.decision_function(X3)
idx=np.argwhere(slack<1)
eslackD=np.zeros(shape=slack.shape)
for index in idx:
eslackD[index]=1-slack[index]
eslack2=np.zeros(shape=Y2.shape)
eslack=eslackD[:len(X1)] #EPSILON OF LABELLED DATA
eslack2=eslackD[len(X1):] # EPSILON FOR UNLABELED DATA
condition=self.checkCondition(Y2,eslack2) #CONDITION OF LOOP
l=0
while(condition): #LOOP 2
l+=1
i,j=self.getIndexCondition(Y2,eslack2) #TAKE A POSITIVE AND NEGATIVE SET
#print("Switching at loop "+str(k)+"."+str(l)+" index: "+str(i)+" "+str(j))
#print("Switching values: "+str(eslack2[i])+" "+str(eslack2[j]))
Y2[i]=Y2[i]*-1 #SWITCHING EXAMPLE
Y2[j]= Y2[j]*-1
sample_weight[len(X1)+i],sample_weight[len(X1)+j]=sample_weight[len(X1)+j],sample_weight[len(X1)+i] #UPDATE THE WEIGHT
Y3=np.concatenate((Y1,Y2),axis=0)
self.clf.fit(X3, Y3, sample_weight=sample_weight) #TRAINING WITH NEW LABELLING
Y3 = Y3.reshape(-1)
#slack =Y3*(self.clf.decision_function(X3))
slack = Y3*self.clf.decision_function(X3)
idx = np.argwhere(slack < 1)
eslackD = np.zeros(shape=slack.shape)
for index in idx:
eslackD[index] = 1 - slack[index]
eslack = eslackD[:len(X1)]
eslack2 = np.zeros(shape=Y2.shape)
eslack2 = eslackD[len(X1):]
condition = self.checkCondition(Y2, eslack2)
k+=1
#print(eslack2)
C_minus=min(2*C_minus,self.Cu)
C_plus=min(2*C_plus,self.Cu)
#print("Loop "+str(k)+" Ctest="+str(self.Cu)+" Cplus="+str(C_plus)+" Cminus="+str(C_minus))
for i in range(len(Y2)):
if (Y2[i] == 1):
sample_weight[len(X1)+i] = C_plus
else:
sample_weight[len(X1)+i] = C_minus
self.Yresult=Y2
Y3 = np.concatenate((Y1, Y2), axis=0)
Y3=Y3.reshape(-1)
end=time.time()
print("The training finish in "+str(end-t)+" seconds")
return self
def checkCondition(self,Y,slack):
'''
Check condition of the loop 2
Parameters
----------
Y: labels of X2
np.array, shape:[n1, ], n1: numbers of samples with semi-labels
slack: slack variable for unlabeled set
np.array, shape:[n1, ], n1: numbers of with semi-labels
'''
condition=False
M=len(Y)
for i in range(M):
for j in range(M):
if((Y[i]!=Y[j]) and (slack[i]>0) and (slack[j]>0) and ((slack[i]+slack[j])>2.001)):
condition=True
return condition
return condition
def getIndexCondition(self,Y,slack):
'''
Get index that satisfies condition of loop 2
Parameters
----------
Y: labels of X2
np.array, shape:[n1, ], n1: numbers of samples with semi-labels
slack: slack variable for unlabeled set
np.array, shape:[n1, ], n1: numbers of with semi-labels
'''
M=len(Y)
for i in range(M):
for j in range(M):
if(Y[i]!=Y[j] and slack[i]>0 and slack[j]>0 and (slack[i]+slack[j]>2.001)):
return i,j
def predict(self, X):
return self.clf.predict(X)
def predict_proba(self):
return self.clf.predict_proba()
def decision_function(self, X):
return self.clf.decision_function(X)
def getResult(self):
return self.Yresult
if __name__ == '__main__':
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score, precision_score, recall_score,roc_curve,f1_score
X,y = load_breast_cancer(return_X_y=True)
y[y==0]=-1
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.985)
print(X_train.shape,X_test.shape)
gammas=[0.001,0.01,0.2,0.4,0.8,1.5,3]
cls=[1,2,5,10,20]
c=1
g=0.0001
clf1 = svm.SVC(C=c,kernel="linear",gamma=g)
#y_train=np.ravel(y_train)
clf1.fit(X_train, y_train)
y_svm = clf1.predict(X_test)
f2 = accuracy_score(y_true=y_test, y_pred=y_svm)
print("ACCURACY SVM " + str(f2))
print("F1 SVM ",f1_score(y_true=y_test,y_pred=y_svm))
print(" ")
#print(clf1.coef_[0])
clf=TransductiveSVM(kernel="linear",Cl=c,Cu=0.5,X2=X_test,gamma=g)
#y_train=np.ravel(y_train)
clf.fit(X_train,y_train)
y_predicted=clf.predict(X_test)
f = accuracy_score(y_true=y_test, y_pred=y_predicted)
print("ACCURACY TSVM " + str(f))
print("F1 TSVM " + str(f1_score(y_true=y_test,y_pred=y_predicted)))
#print(clf.clf.coef_[0])
#print(y_svm)输出:
(8, 30) (561, 30)
ACCURACY SVM 0.8645276292335116
F1 SVM 0.9025641025641026
The training finish in 4.932965517044067 seconds
ACCURACY TSVM 0.8413547237076648
F1 TSVM 0.8593996840442337
五、基于图的算法


使用Python实现的基于图的半监督学习算法的示例代码,它使用了标签传播(Label Propagation)的方法:
# 导入相关的库
import numpy as np
from sklearn.datasets import make_blobs
from sklearn.metrics.pairwise import rbf_kernel
# 生成数据,其中有三个类别,每个类别有两个特征
X, y = make_blobs(n_samples=1000, n_features=2, centers=3, random_state=0)
# 随机选择一部分数据作为标记数据,其余的作为未标记数据
n_labeled = 100
indices = np.random.permutation(X.shape[0])
X_labeled = X[indices[:n_labeled]]
y_labeled = y[indices[:n_labeled]]
X_unlabeled = X[indices[n_labeled:]]
# 用标记数据和未标记数据构建一个图,用高斯核函数作为相似度度量,用k近邻图作为图结构
k = 10 # 近邻的个数
sigma = 1.0 # 高斯核函数的参数
# 计算数据点之间的高斯核矩阵
K = rbf_kernel(X, gamma=1.0 / (2 * sigma ** 2))
# 对每个数据点,只保留最近的k个邻居的相似度,其余的设为0,得到一个稀疏的相似度矩阵
W = np.zeros_like(K)
for i in range(X.shape[0]):
# 找到第i个数据点的最近的k个邻居的索引
neighbors = K[i].argsort()[-(k + 1):-1]
# 将这些邻居的相似度保留,其余的设为0
W[i, neighbors] = K[i, neighbors]
# 使相似度矩阵对称,即如果i和j是邻居,那么j和i也是邻居
W = np.maximum(W, W.T)
# 用标记数据的类别信息来初始化图中的节点的标签,用一个矩阵Y表示,其中Y[i, j]表示第i个数据点属于第j个类别的概率
n_classes = len(np.unique(y)) # 类别的个数
Y = np.zeros((X.shape[0], n_classes)) # 初始化标签矩阵
# 对于标记数据,用one-hot编码表示其类别,即Y[i, j] = 1当且仅当第i个数据点属于第j个类别
for i in range(n_labeled):
Y[indices[i], y_labeled[i]] = 1
# 对于未标记数据,用均匀分布表示其类别,即Y[i, j] = 1 / n_classes
Y[indices[n_labeled:]] = 1.0 / n_classes
# 用图中的边来传播节点的标签,用标签传播的方法
alpha = 0.99 # 平滑系数,介于0和1之间,越接近1表示越依赖于未标记数据的信息,越接近0表示越依赖于标记数据的信息
n_iter = 10 # 迭代次数
# 在每次迭代中,用以下的公式来更新标签矩阵:Y = alpha * W * Y + (1 - alpha) * Y
for i in range(n_iter):
# 计算W * Y,即用每个数据点的邻居的标签的加权平均来更新其标签
WY = W.dot(Y)
# 将标记数据的标签保持不变,即用(1 - alpha) * Y来表示
WY[indices[:n_labeled]] = Y[indices[:n_labeled]]
# 用alpha * W * Y + (1 - alpha) * Y来更新标签矩阵
Y = alpha * WY + (1 - alpha) * Y
# 对每个数据点,将其标签归一化,使得其概率之和为1
Y = Y / Y.sum(axis=1, keepdims=True)
# 用收敛后的标签矩阵来对数据进行分类,即将数据分配给概率最大的类别
y_pred = Y.argmax(axis=1)
# 计算分类的准确率
accuracy = np.mean(y_pred == y)
print(f"Accuracy: {accuracy:.2f}") # Accuracy: 0.91六、半监督深度学习



ladderNet网络