PCA(Principal Component Analysis),中文名称:主成分分析。迄今为止最流行的降维算法。
假设 n 维空间中的一个单位立方体,易知:一维空间中该立方体中任意两点的距离不超过 1 1 1,二维空间中该立方体中任意两点的距离不超过 2 \sqrt{2} 2,三维空间中该立方体中任意两点的距离不超过 3 \sqrt{3} 3,一百万维空间中该立方体中任意两点的距离不超过 1000000 = 1000 \sqrt{1000000}=1000 1000000=1000。明明是边长为 1 1 1 的单位立方体,一百万维空间中的最大距离却有 1000 1000 1000,这说明高维空间中的数据是非常稀疏的。
PCA 可以识别哪些维度上的信息量最高(主要成分),并将数据投影到那些有着最高信息量维度表示的平面上。PCA 使用 SVD (奇异值分解)找到数据的主要成分。
比如说有 100 个三维空间中的数据
A
100
×
3
A_{100 \times 3}
A100×3(假设数据以原点为中心,如果不是先将数据居中,这是为了消除数据的均值影响,确保主成分的计算是基于数据的奇异性而不是均值),对其使用 SVD 分解:
U
100
×
100
⋅
S
100
×
3
⋅
V
3
×
3
T
=
A
100
×
3
U_{100 \times 100} \cdot S_{100 \times 3} \cdot V^T_{3\times 3} = A_{100 \times 3}
U100×100⋅S100×3⋅V3×3T=A100×3
其中
U
,
V
U,V
U,V 分别是左右奇异阵,为正交阵,
S
S
S 是奇异值阵,为对角阵。
要将数据降为多少维,就保留 V V V 的前几列,再将 A A A 与其相乘,就得到了降维后的数据。
如将三维空间数据集
A
A
A 降为
2
2
2 维,就保留
V
V
V 的前两列,设
W
=
V
(
:
,
1
:
2
)
W = V(:,1:2)
W=V(:,1:2)
则降维后的数据为:
A
r
e
d
u
c
e
d
=
A
⋅
W
A_{reduced} = A \cdot W
Areduced=A⋅W
将降维后的数据恢复原来的维度:
A
r
e
c
o
v
e
r
e
d
=
A
r
e
d
u
c
e
d
⋅
W
T
A_{recovered} = A_{reduced} \cdot W^T
Arecovered=Areduced⋅WT
恢复后的
A
r
e
c
o
v
e
r
e
d
≠
A
A_{recovered} \ne A
Arecovered=A,因为
W
⋅
W
T
≠
I
W \cdot W^T \ne I
W⋅WT=I。这会损失掉
A
A
A 的一部分信息。
另外可以通过
S
S
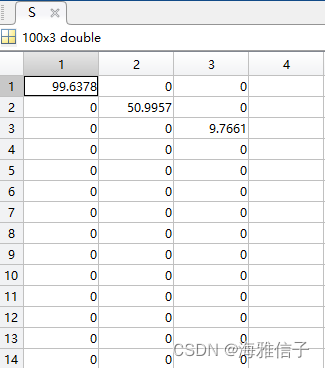
S 矩阵知道降维后的数据保留了多少信息量。比如说
S
S
S 矩阵为
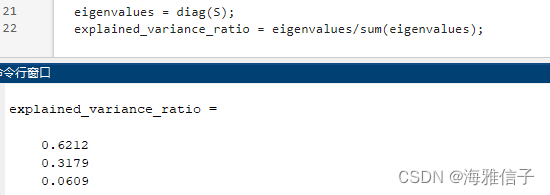
即每一维的信息比为
将
A
A
A 降为
2
2
2 维,降维后的数据保留了
A
A
A 中
93.91
%
93.91\%
93.91% 的信息。
代码如下:
% created by hyacinth on 2024/1/9
clc
clear
close all
%%
A1 = 10*randn(100,1);
A2 = 5*randn(100,1);
A3 = randn(100,1);
A = [A1,A2,A3];
A = A - mean(A);
[U,S,V] = svd(A);
newdim = 2;
W = V(:,1:newdim);
A_reduced = A*W;
A_recovered = A_reduced*W';
eigenvalues = diag(S);
explained_variance_ratio = eigenvalues/sum(eigenvalues);
info_ratio = sum(explained_variance_ratio(1:newdim));
disp("the retained information ratio is : "+num2str(info_ratio))