单细胞超大数据集的热图怎么画?昨天刚做完展示20万单细胞的热图要这么画吗? 今天就有人发消息问我为啥他画出来的热图有问题。
问题起源
昨天分享完 20万单细胞的热图要这么画吗?,就有人问为啥他的数据会出错。我们先来看下他的数据
-
数据输入部分
cluster_gene_stat=FindAllMarkers(d.all,only.pos = TRUE,logfc.threshold = 0.4)head(cluster_gene_stat)table(cluster_gene_stat$cluster)DimPlot(d.all)d.all$orig.ident=Idents(d.all)table(d.all$orig.ident)Idents(d.all)=paste0('cluster',d.all$orig.ident)a=AverageExpression(d.all,return.seurat = TRUE)a$orig.ident=rownames(a@meta.data)head(a@meta.data)head(markers)markers=cluster_gene_statDoHeatmap(a,draw.lines = FALSE, slot = 'scale.data',assay = 'SCT',features = markers %>%group_by(cluster) %>%dplyr::slice_max(avg_log2FC,n = 5) %>% .$gene )
-
画图所需数据
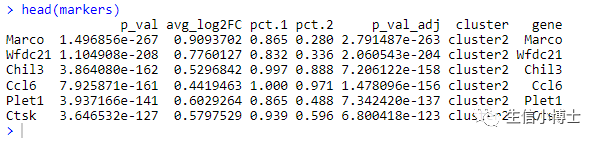
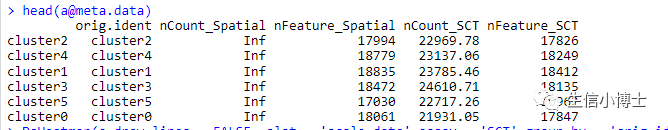
他画出来的图是这样:
markers$cluster=paste0("cluster",markers$cluster)markers$cluster=factor(markers$cluster,levels = unique(markers$cluster))DoHeatmap(a,draw.lines = FALSE, slot = 'scale.data',assay = 'SCT',features = markers %>%group_by(cluster) %>%dplyr::slice_max(avg_log2FC,n = 5) %>% .$gene )
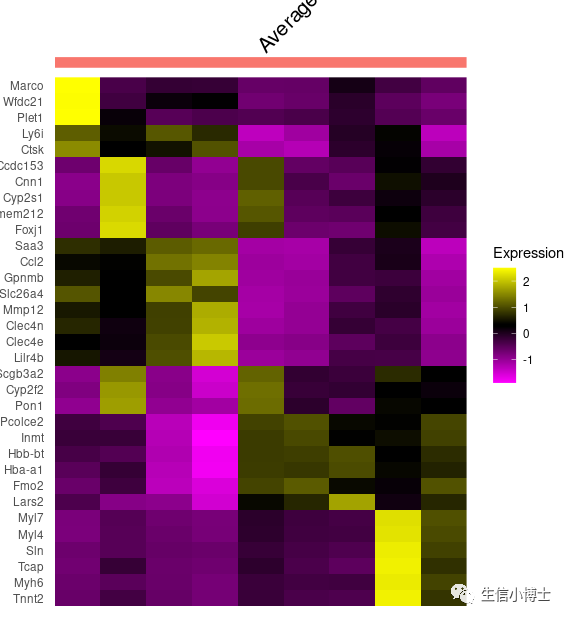
-
和我的图20万单细胞的热图要这么画吗? 区别在于没有列名,图注也不完整,且热图色块是乱序的
解决方法
-
1. 方倒是简单,重新添加列名即可添加即可
Idents(d.all)=paste0('cluster',d.all$orig.ident)a=AverageExpression(d.all,return.seurat = TRUE)a$orig.ident=rownames(a@meta.data)DoHeatmap(a,draw.lines = FALSE, slot = 'scale.data',assay = 'SCT',group.by = 'orig.ident',features = markers %>%group_by(cluster) %>%dplyr::slice_max(avg_log2FC,n = 5) %>% .$gene )
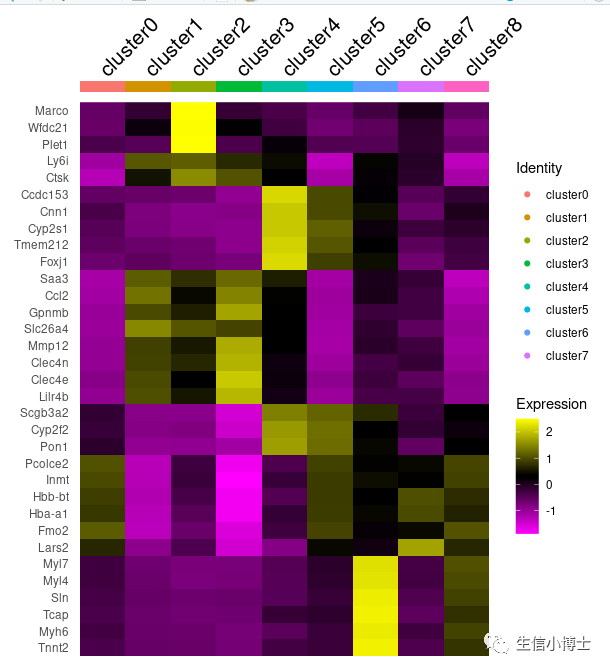
-
2. 调整因子顺序,让热图中的色块更好看些
markers$cluster=factor(markers$cluster,levels = paste0('cluster',seq(0,8,1)) )head(markers)head(a@meta.data)DoHeatmap(a,draw.lines = FALSE, slot = 'scale.data',assay = 'SCT',group.by = 'orig.ident',features = markers %>%group_by(cluster) %>%dplyr::slice_max(avg_log2FC,n = 5) %>% .$gene )
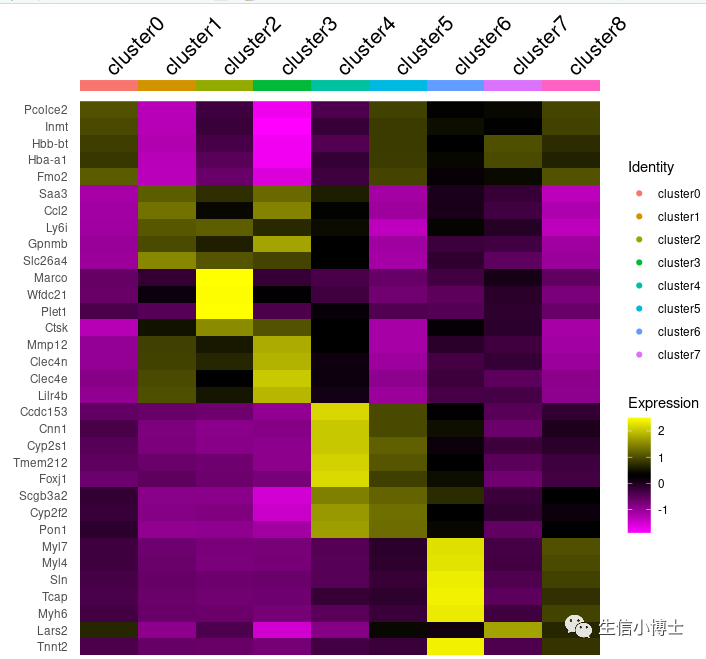
但是你会发现 右边地图注少了cluster8。
为什么cluster8会缺失?我觉得这是bug,盲猜可能由于markers内的cluster8对应地基因在a里是na
-
3. 自己手动添加图注cluster8即可
DoHeatmap(a,features = markers %>%group_by(cluster) %>%dplyr::slice_max(avg_log2FC,n = 5) %>% .$gene,draw.lines = FALSE,group.by = 'orig.ident') +ggplot2:: scale_color_discrete(name = "Identity", labels = paste0('cluster',seq(0,8,1)) )
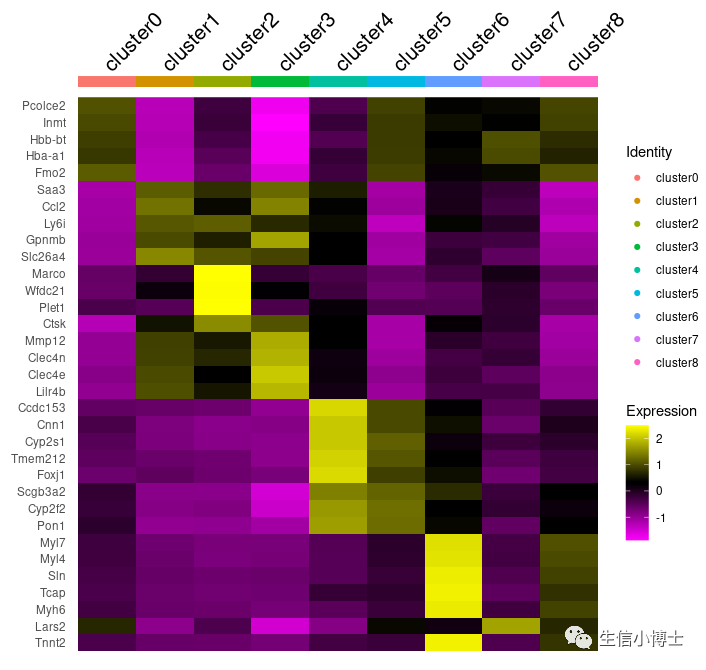
-
3. 如果热图中想多加几个基因的话
DoHeatmap(a,features = markers %>%group_by(cluster) %>%dplyr::slice_max(avg_log2FC,n = 15) %>% .$gene,draw.lines = FALSE,group.by = 'orig.ident') +ggplot2:: scale_color_discrete(name = "Identity", labels = paste0('cluster',seq(0,8,1)) )
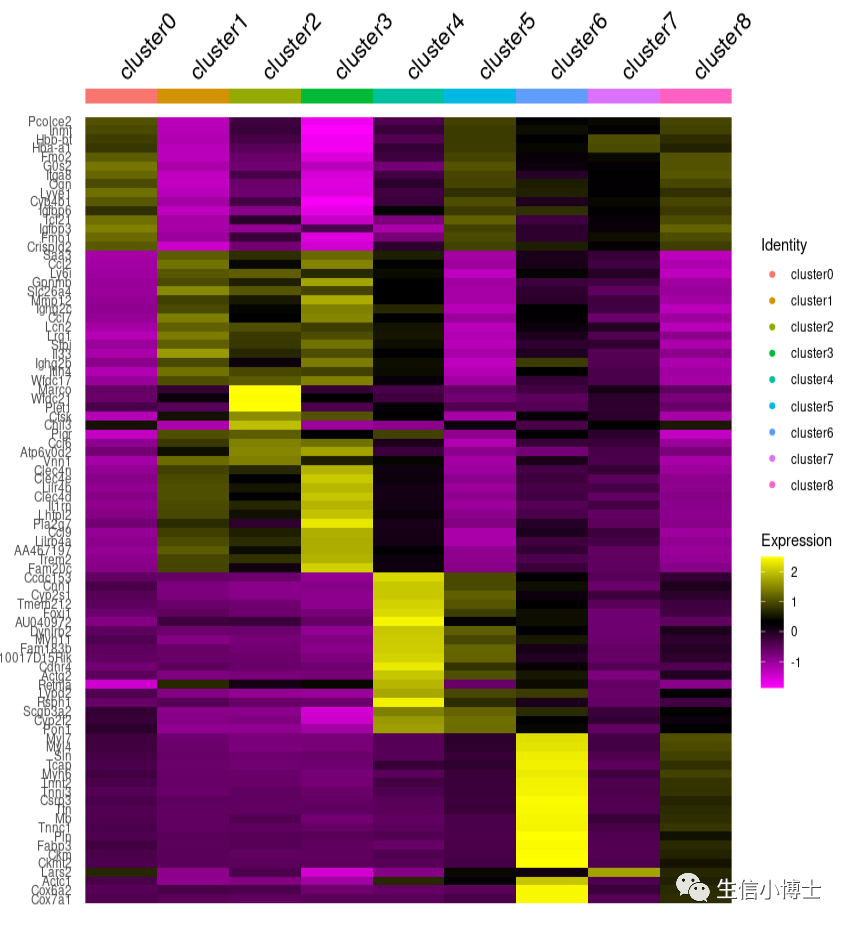
只能这样了,这里所有热图都挺丑的,瘸子里面选巧匠吧

画外音:
这种热图不好看的根本原因在于数据的问题。提示我们需要合并、删除某些cluster,然后再来画热图会更好看。
如果你不会合并、删除某些cluster。可以先看看我之前的直播:
直播四-单细胞个性化注释、细分亚群并把细分亚群放回总群
如果还是不懂,找机会再直播一次~