1、基于metawrap环境计算bin丰度(推荐)

MetaWRAp(Metagenomic Workflow for Assembly, binning, and annotation)是一个用于处理宏基因组学数据的工具,包括元组装、分箱(binning)、基因组注释等功能。要基于 MetaWRAp 计算宏基因组分箱结果的丰度,你可以按照以下步骤进行:
-
安装 MetaWRAp:首先确保已经安装了 MetaWRAp 工具。你可以通过官方的 GitHub 页面(https://github.com/bxlab/metaWRAP)或者相关文档找到安装说明。
-
准备输入数据:准备你的宏基因组学数据,包括原始测序数据(fastq 或者 fasta 格式)以及进行元组装得到的 contigs 或 scaffolds。
-
运行 MetaWRAp:使用 MetaWRAp 中的相应工具来计算 bins 的丰度。在 MetaWRAp 中,你可以使用
metabat2_wf或maxbin2_wf等工作流来执行分箱操作,并获得 bins 的丰度信息。
示例命令(以 MetaBAT2 为例):
source activate metawrap
#示例:
metawrap metabat2_wf -o output_directory -t num_threads assembly.fasta reads_1.fastq,reads_2.fastq
metawrap quant_bins \
-t 32 \
-o Bin_quant/ \
-b Bin/ \
-a final.contigs.fa \
Clean_data/H*.fastq
# 参数:
# -t # 线程
# -o # 输出文件夹(自动创建)
# -b # Bin所在文件夹
# -a # contig组装结果
# 最后是fastq/a(初始数据)文件所在文件夹其中:
-o output_directory:指定输出目录。-t num_threads:指定线程数。assembly.fasta:是你的组装后的 contigs 或 scaffolds 文件。reads_1.fastq,reads_2.fastq:是原始测序数据(paired-end reads)。
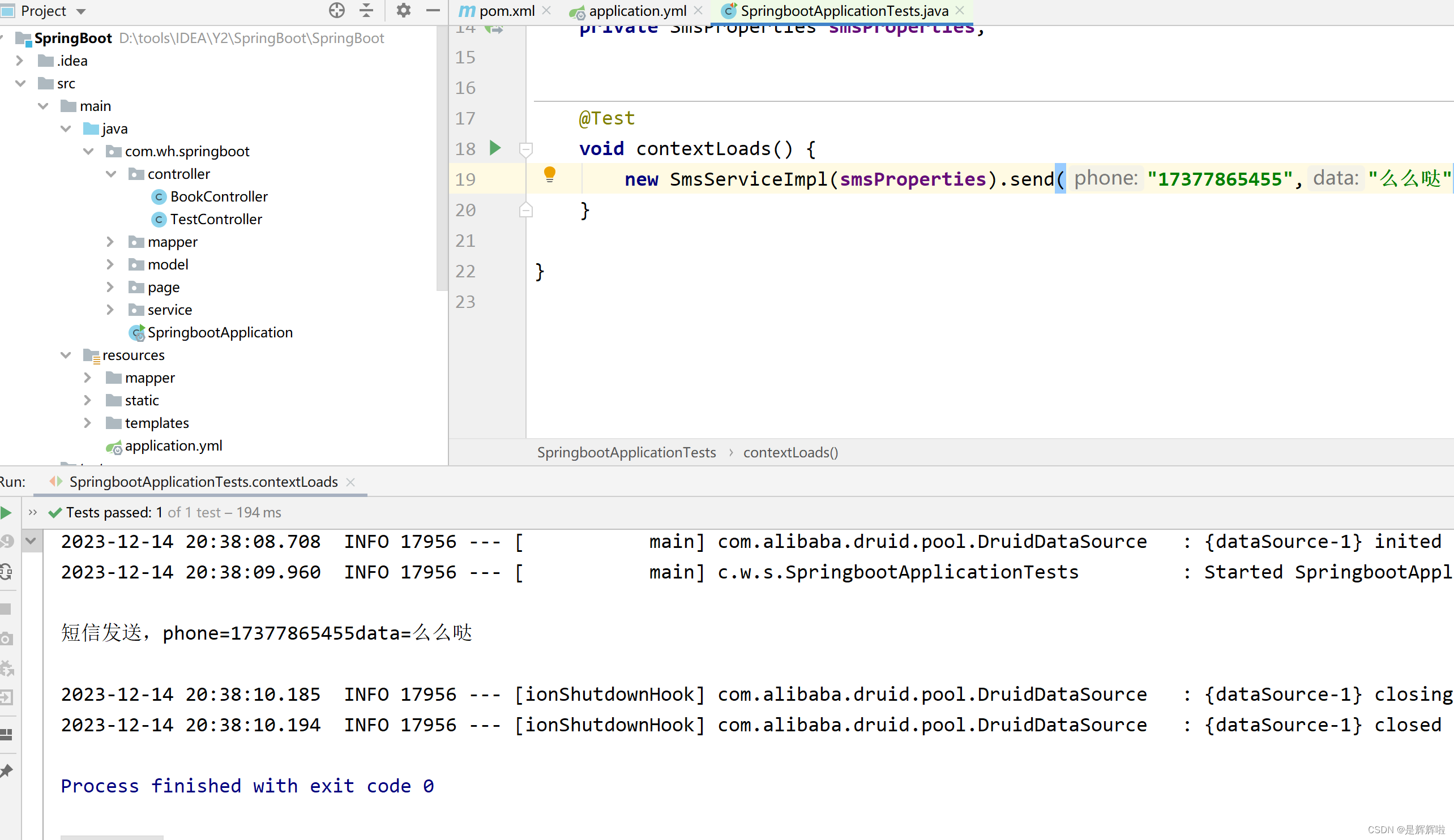
这个挺好的就是直接出图了,相应数据结果直接在bin_quant文件夹。
2、基于coverm环境计算bins丰度

CoverM 是一个用于基因组学和宏基因组学研究的工具,用于评估基因组或分箱(bins)的覆盖度和丰度。以下是使用 CoverM 计算 bins 丰度的一般步骤:
-
安装 CoverM:首先需要安装 CoverM 工具及其相关依赖。你可以从 CoverM 的官方 GitHub 页面(https://github.com/wwood/CoverM)获取安装说明。
-
准备输入数据:准备输入数据,这通常是包含基因组组装结果的 FASTA 格式文件。这可能是来自于元组装工具(如MetaBAT、MaxBin、CONCOCT等)输出的 bins。
-
运行 CoverM:使用 CoverM 来计算 bins 的丰度。下面是一个示例命令:
coverm contig \
--genome-fasta input_bins.fasta \
--min-read-aligned-percent 95 \
--min-read-aligned-length 1000 \
--min-covered-fraction 0.5 \
--min-base-quality 0 \
--min-read-length 100 \
--reference-fasta reference_sequences.fasta \
--output-dir output_directory
这里的参数解释如下:
--genome-fasta:指定输入的 bins 文件。--min-read-aligned-percent:指定至少需要覆盖的 reads 百分比。--min-read-aligned-length:指定最小的 reads 长度。--min-covered-fraction:指定一个 contig 被认为是被覆盖的最小比例。--min-base-quality:指定 reads 的最小质量。--min-read-length:指定最小的 reads 长度。--reference-fasta:可选参数,用于提供参考序列。--output-dir:指定输出目录。
同样超级简单,直接上代码:
mamba activate coverm
# 建议sh脚本,采用nohup运行,这个还是要耗点时间的
for i in $(ls bins); \
do \
coverm genome \
-d $i/metabat2_bins \
-x fa \
-t 80 \
-c read_qc/${i}/*.fastq \
> coverm/${i}.mtbt2.bins.out ;\
done