步骤学习:
1,基因筛选:
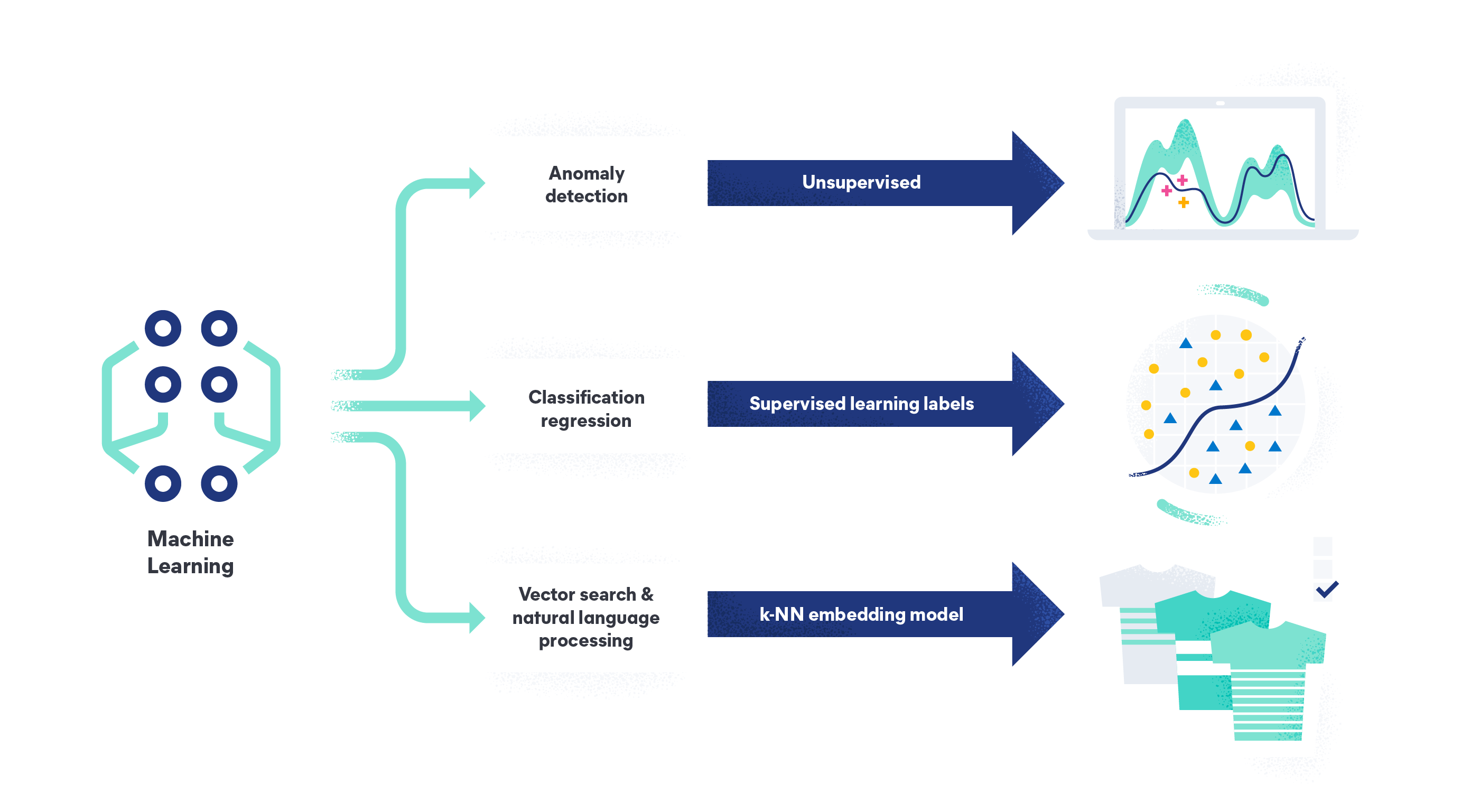
作者使用先前研究得出的 244 肿瘤免疫相关基因(244里有AB两个细分亚集),对特征基因进行缩减。从多个免疫治疗队列中获取这些基因的重要性特征。(TCGA-SKCM、GSE78220、GSE115821、GSE35640、IMvigor210、Snyder et al.)
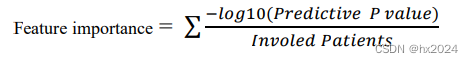
每个基因在每个治疗队列中预测免疫治疗反应的显著性差异P值,取−𝑙𝑜𝑔10P除每个队列的样本数量。然后把每个基因所有队列的“特征值”相加即为每个基因的特征值。(P越小,特征值越大)
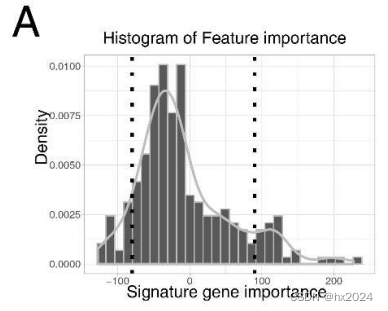
特征基因筛选:Genes with feature importance lesser than -90 and larger than +80。(基因的正负号应该是)
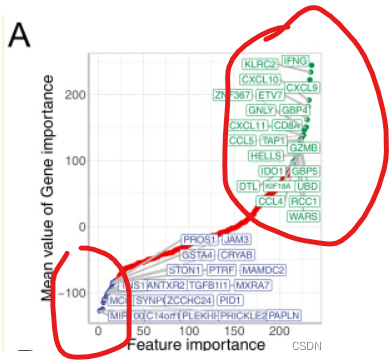
右上绿色正相关,左下蓝色负相关
这个图纵坐标为计算的基因值,横坐标为基因顺序(按直方图顺序)
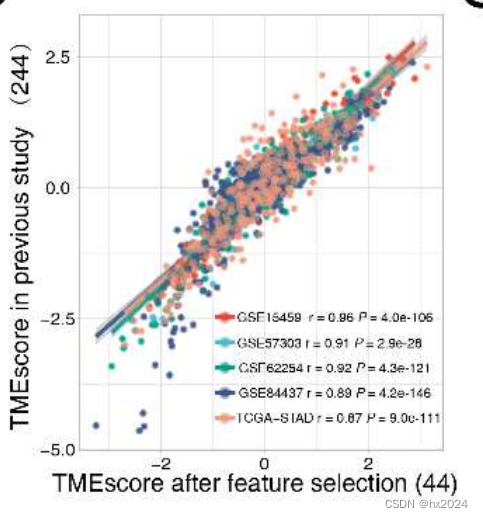
降维后的基因PCA得分与原来的基因集PCA得分相关性高。
2,建模
分别对A和B基因集进行PCA算法,以PC1为模型得分。
TMEscore = ∑ PC1i – ∑PC1j,where i is the signature score of clusters whose Cox coefficient is positive, and j is the expression level of the gene whose Cox coefficient is negative.
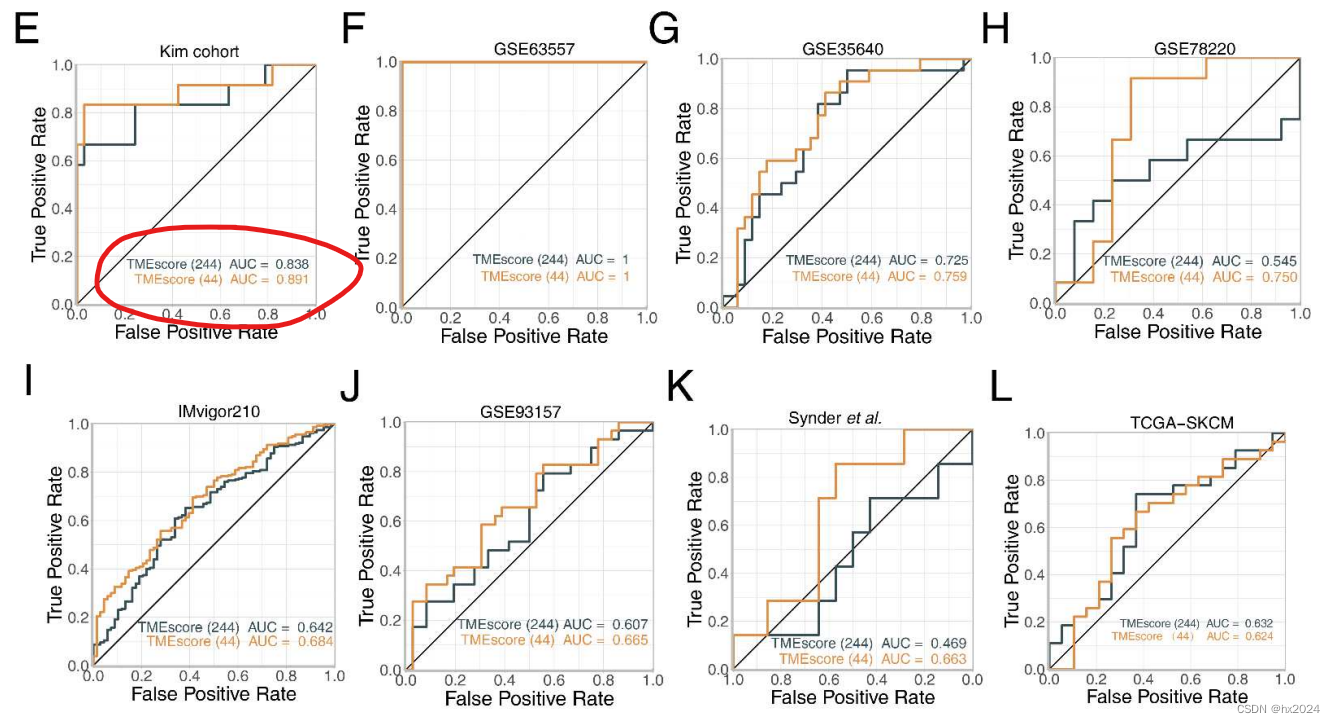

244基因构建的PCA模型性能与44基因构建的模型相比较。
(44-gene模型更好,可能是模型构建的基因经过降维后,混杂的基因更少性能更佳)
3,模型性能比较
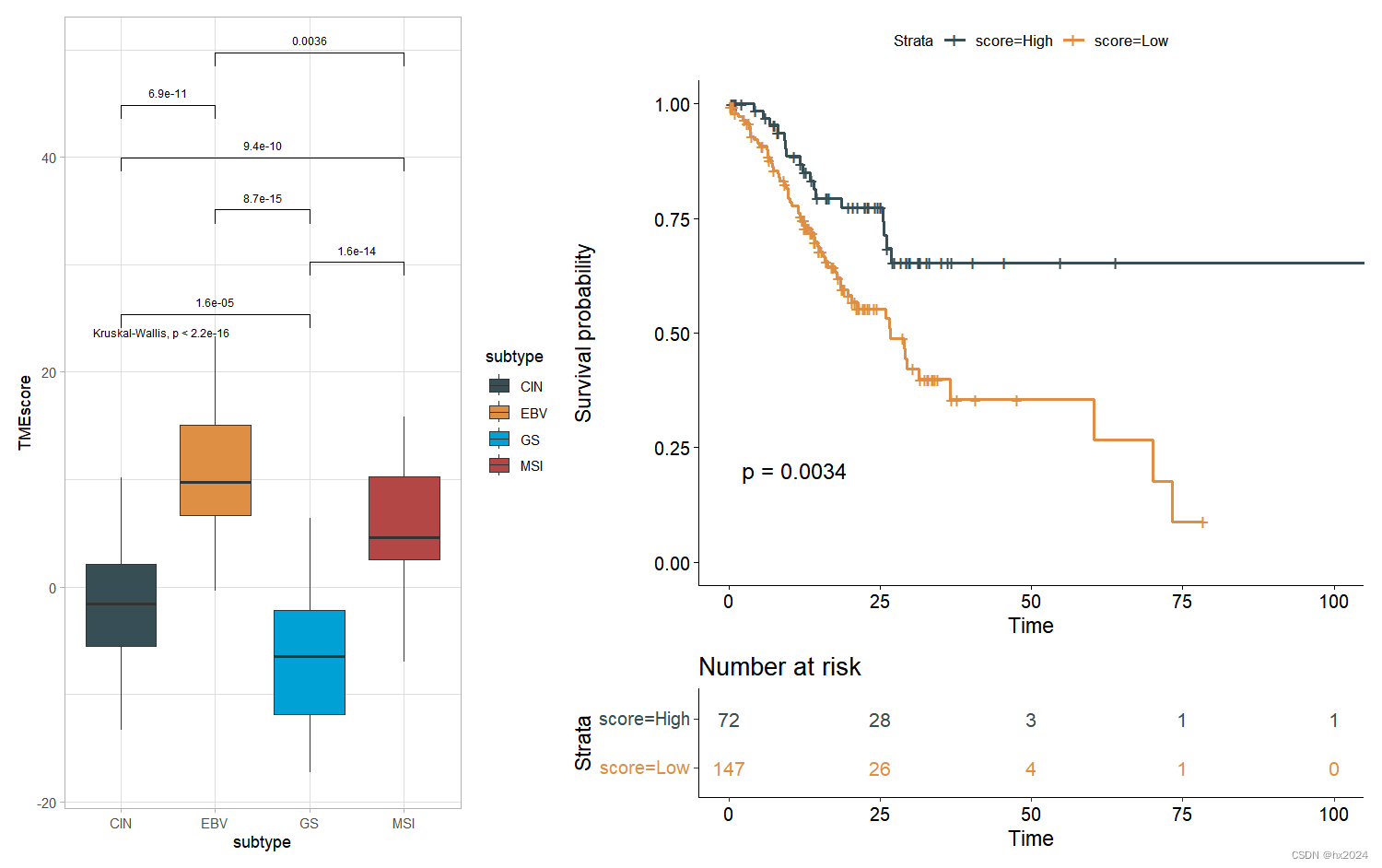
TMEscore 在 mGC 中比 MSI、EBV 和 TMB 准确识别更多的患者
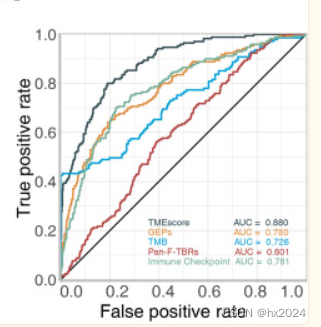
使用
DongqiangZeng0808/TMEscore: Calculating Tumor microenvironment score (github.com)
数据:
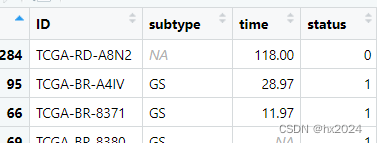
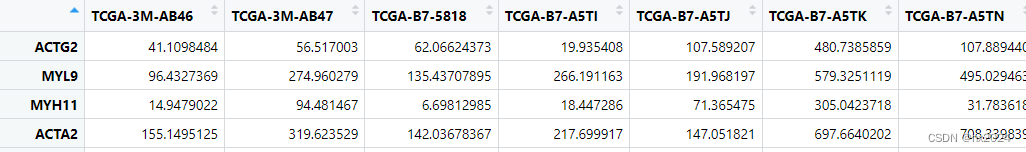
rm(list = ls())
library('TMEscore')
library("ggplot2")
library("patchwork")
eset <- eset_stad
pdata <- pdata_stad
tmescore<-tmescore(eset = eset_stad, #expression data
pdata = pdata_stad, #phenotype data
method = "PCA", #default
classify = T) #if true, survival data must be provided in pdata
head(tmescore)
结果:
#remove observation with missing value
tmescore<-tmescore[!is.na(tmescore$subtype),]
p<-ggplot(tmescore,aes(x= subtype,y=TMEscore,fill=subtype))+
geom_boxplot(notch = F,outlier.shape = 1,outlier.size = 0.5)+
scale_fill_manual(values= c('#374E55FF', '#DF8F44FF', '#00A1D5FF', '#B24745FF'))
comparision<-combn(unique(as.character(tmescore$subtype)), 2, simplify=F)
p1<-p+theme_light()+
stat_compare_means(comparisons = comparision,size=2.5)+
stat_compare_means(size=2.5)
# survival analysis
colnames(tmescore)[which(colnames(tmescore)=="TMEscore_binary")]<-"score"##修改列名
fit<- survfit(Surv(time, status) ~ score, data = tmescore)
p2<-ggsurvplot(fit,
conf.int = FALSE,
palette = c('#374E55FF', '#DF8F44FF'),
risk.table = TRUE,
pval = TRUE,
risk.table.col = "strata")
p2<-list(p2)
p2 <- arrange_ggsurvplots(p2, print = FALSE, ncol = 1, nrow = 1)
# print plots
(p1|p2)+plot_layout(ncol = 2, widths = c(1,2))
作者提供的基因ACRG有244个,TCGA数据只有235个。其中缺失的部分基因是因为多基因名,GEO数据库的探针和TCGA对应的基因名没有对应。可以共同转换为ENS后进行比对。
参考文献:
1:Tumor Microenvironment Characterization in Gastric Cancer Identifies Prognostic and Immunotherapeutically Relevant Gene Signatures
2:Tumor microenvironment evaluation promotes precise checkpoint immunotherapy of advanced gastric cancer