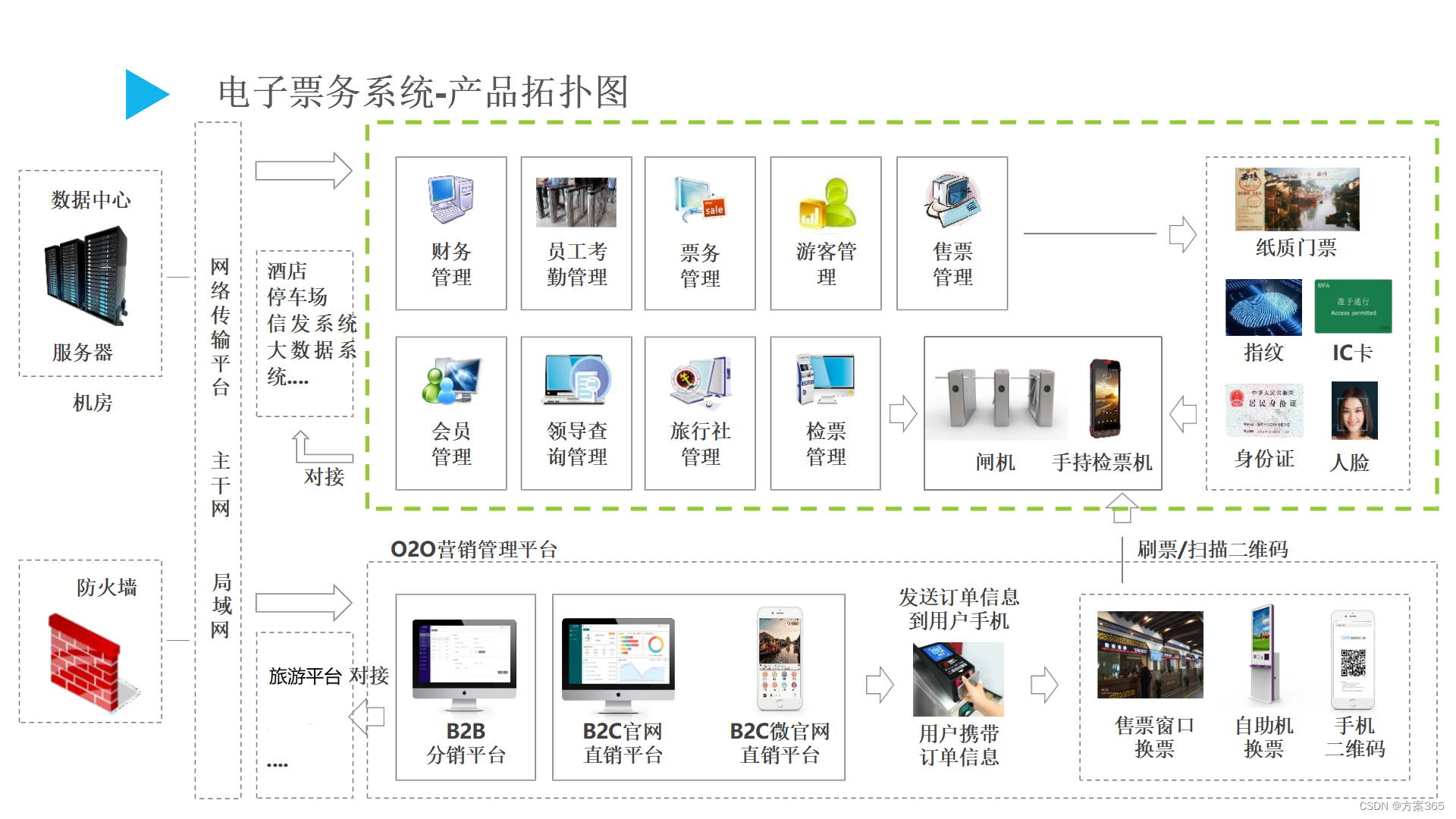
目前植物单细胞转录组测序主流平台的输入都是原生质体悬液或者细胞核悬液。

那么到底采用哪种更好呢?
啥?
成年人少做选择?
看看账上,摸摸兜里,得有财力支持啊!

本期,聊聊这个主题,有错的话请读者留言批评。
首先,上个表:
表1 植物单细胞转录组方法学对比
| 项目 | 提(抽)核 snRNA-Seq | 原生质体(解离)scRNA-Seq |
| 细胞大小 | 对细胞直径无限制 | 细胞最大直径<40微米 |
| 细胞类型全面性 | 更全面,破碎提核法对不同类型细胞通用性较强 | 有偏好,酶解较难的细胞类型无法富集获取 |
| 实验操作 | 较个性化,不同物种不同组织可优化处理条件;实验周期相对较短 | 极个性化,需要根据不同物种不同组织类型摸索处理条件;实验周期相对较长 |
| 样本要求及适应范围 | 较低,同普通转录组,鲜样或者冻存等都适用,支持范围广 | 较高,需鲜样,支持范围窄 |
| 基因表达信息全面性 | 仅细胞核内 | 整个细胞内 |
| 对基因表达的影响 | 液氮速冻,应激变化小 | 酶解消化处理,应激变化较大 |
| 获得的RNA测序信息 | 前体mRNA占比高 | 成熟mRNA为主 |
一、细胞大小
植物细胞通常大小10-100微米(图1)。目前主流单细胞转录组平台芯片因孔径限制通常要求输入的细胞大小在40um以内,超过此大小的细胞类型存在无法富集的极高风险,此外质检对细胞的结团率等指标也有一定的要求。而植物细胞核直径一般小于20微米,因此提核法对细胞大小几无限制(图2)。
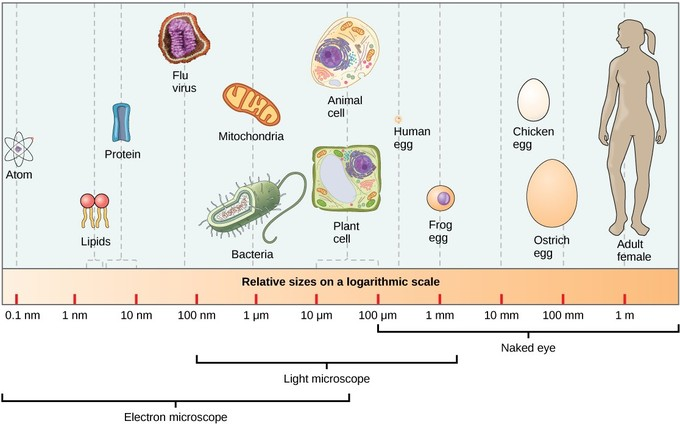
图1 动植物细胞大小示意图
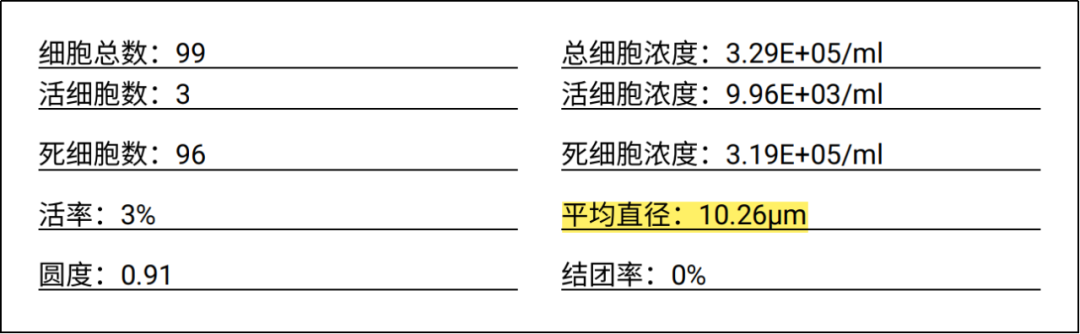
图2 某高等植物种子提核质控检测(高脂肪高蛋白样本类型)
二、细胞类型
植物不同于动物,细胞有细胞壁,主要组成为纤维素和果胶(真菌为几丁质、多糖等),且不同物种不同组织不同生长阶段酶解制备原生质体难易不同,那些不易解离的细胞容易丢失(例如内层组织中柱、胚乳细胞等),而一些较为敏感的细胞可能会因为解离过度而破碎,总之解离可能无法有效获取到标的组织中的所有细胞类型,对结果的准确性有一定的影响[1]。因此我们会看到目前采用原生质体样本的研究,其样本材料绝大多数为幼嫩鲜样。
提核法采用机械破碎细胞获取细胞核,没有酶解法的偏向性,理论上所有的细胞类型均能有效回收,结果更全面完整(尤其是稀有细胞类型)。提核法通用性较强,凌恩生物仍在针对不同样本(例如高蛋白高脂肪高淀粉高次生代谢物等)持续优化处理条件。
另外需要指出的是,有些样本类型因其特殊性,只能采用提核法处理。例如发表于《Developmental Cell》的论文研究水稻雌蕊的发育轨迹[2],作者指出因雌蕊富含多种多糖胶质成分而无法制备原生质体,以植物花粉为研究对象面临同样的情况[3]。作为一种富含蜡质的木本植物,研究者指出荔枝在技术上很难制备原生质体,其对花蕾进行酶解处理后发现大量的细胞残骸、破裂的原生质体和未能消化的细胞壁,转而选择提核制备[4]。

图3 水稻雌蕊snRNA-Seq研究

图4 拟南芥花粉snRNA-Seq研究

图5 荔枝花蕾snRNA-Seq研究
三、样本要求
制备原生质体需要鲜样(植物里尚未看到冷冻复苏做单细胞的,如果这么干第一成本很高,第二效果也不确定),对于样本的采集、保存、运输要求较高。
提核法除鲜样外,冻存等样本同样适用(核膜比细胞膜更皮实,冻存后细胞膜破损而核膜能保持完整),因此提核法的样本要求跟普通转录组一致,样本支持范围更广。
四、基因及其表达
原生质体法可检测细胞内全部的mRNA,而提核法只检测细胞核内的mRNA,因此提核法检测到的前体mRNA(未剪切的转录本,包含内含子)占比会较高。
多项对比研究表明,两种方法学的一致性较好。

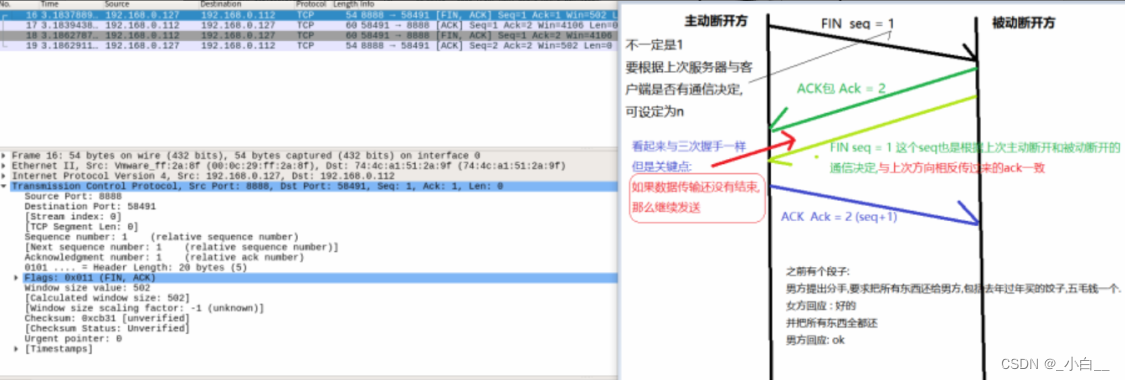
该文报道了拟南芥根组织中单细胞核RNA测序(snRNA-Seq)和单细胞核ATAC测序(snATAC-Seq)[5]。通过与已发表的基于原生质体scRNA-Seq比较,验证了snRNA-Seq的可靠性与一致性。此外,snRNA-Seq的结果揭示了之前scRNA-Seq并未鉴定到的新的细胞类型(一个细胞簇独有,两个细胞簇高占比)。
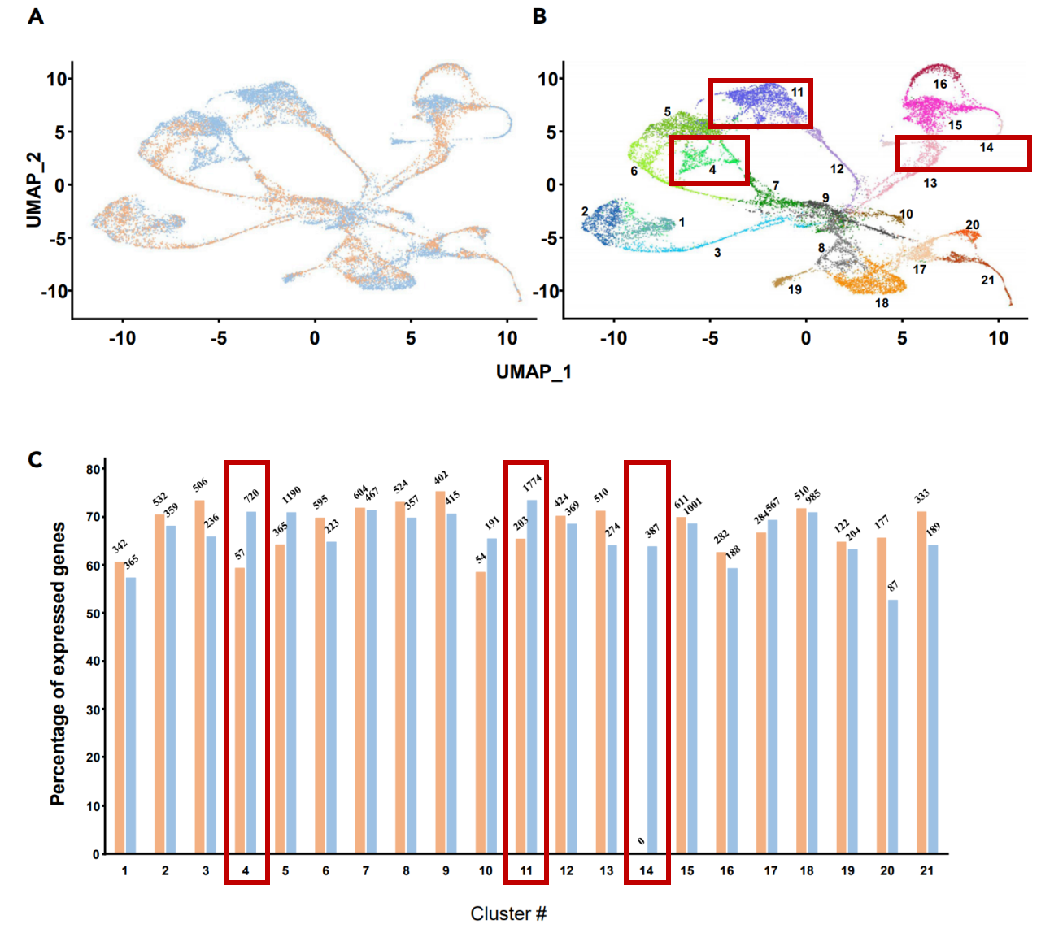
图6 拟南芥原生质体(橙色)和提核(蓝色)单细胞转录组的比对分析(Cluster14为snRNA-Seq独有;Cluster4和11源自snRNA-Seq占比超过89%)
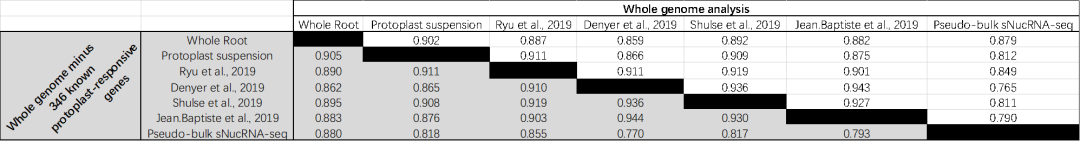
图7 拟南芥snRNA-Seq数据与公开发表的scRNA-Seq数据相关性分析
(Spearman’s Rank Correlation Coefficient)

另外这篇文章也同样比较了两种方法学,得到了类似的结论(部分指标snRNA-Seq甚至更优)。该文验证了非流式分选提核法单细胞转录组的鲁棒性(robustness),不论是在细胞分类还是标记基因鉴定上均与原生质体法一致[6]。
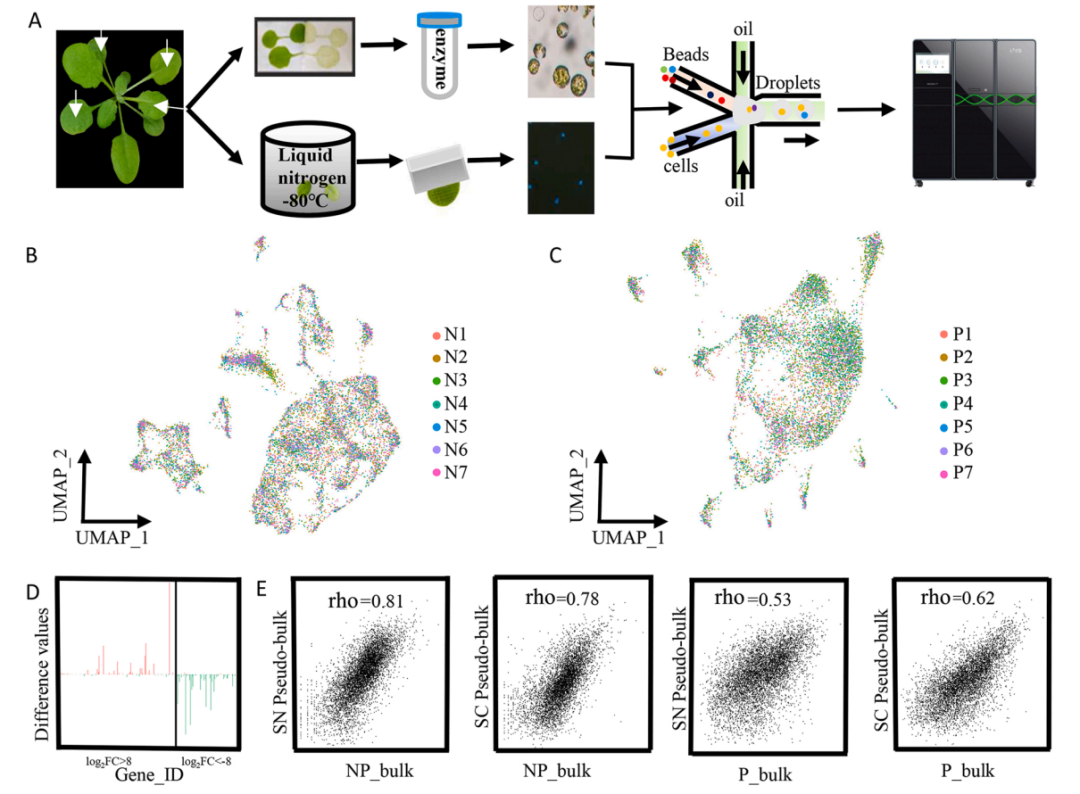
图8 拟南芥snRNA-Seq与scRNA-Seq比较研究
有一点需要提示,前面提到植物的细胞壁主要成分是纤维素和果胶,制备原生质体通常使用纤维素酶和果胶酶进行处理,相当于给细胞施加胁迫,可能会通过基因的表达变化做出一系列的应激反应以适应该胁迫,存在人为因素引入转录偏差的可能性。因此对于特定研究目的的实验,不能采用原生质体法。而提核法采用液氮速冻,细胞的应激变化小,更能反应细胞基因表达的真实情况。
例如刚刚发表在《Nature Plants》上的苜蓿单细胞转录组研究,作者清晰的写明对于早期共生互作研究,原生质体法并不可取,原文如下:“…However, the plant cell wall must be enzymatically digested to obtain free single cells, a process called protoplasting. Unfortunately, the expression levels of many genes, especially those involved in defence and stress responses, change during this treatment, therefore making protoplasting undesirable for studying genes that participate in early symbiotic interactions. Single-nucleus RNA sequencing (snRNA-seq) has offered a route to bypass the prerequisite of protoplasting for single-cell sequencing and shown great promise……”[7]。
小结
相比于基于原生质体的植物scRNA-Seq,基于提核的植物snRNA-Seq具有不怎么挑样本,样本寄送有干冰说走就走,实验简单易上手,多个方面不偏不倚,结果不差可能还有惊喜等众多优点。
随着越来越多的snRNA-Seq牛文发表,小编送它八个字:趋势已现,未来可期。
1. Shaw, R., X. Tian, and J. Xu, Single-Cell Transcriptome Analysis in Plants: Advances and Challenges. Mol Plant, 2021. 14(1): p. 115-126.
2. Li, C., et al., Single-nucleus sequencing deciphers developmental trajectories in rice pistils. Dev Cell, 2023. 58(8): p. 694-708 e4.
3. Ichino, L., et al., Single-nucleus RNA-seq reveals that MBD5, MBD6, and SILENZIO maintain silencing in the vegetative cell of developing pollen. Cell Rep, 2022. 41(8): p. 111699.
4. Yang, M.C., et al., Single-nucleus RNA sequencing and mRNA hybridization indicate key bud events and LcFT1 and LcTFL1-2 mRNA transportability during floral transition in litchi. J Exp Bot, 2023. 74(12): p. 3613-3629.
5. Farmer, A., et al., Single-nucleus RNA and ATAC sequencing reveals the impact of chromatin accessibility on gene expression in Arabidopsis roots at the single-cell level. Mol Plant, 2021. 14(3): p. 372-383.
6. Wang, K., et al., An optimized FACS-free single-nucleus RNA sequencing (snRNA-seq) method for plant science research. Plant Sci, 2023. 326: p. 111535.
7. Liu, Z., et al., Single-nucleus transcriptomes reveal spatiotemporal symbiotic perception and early response in Medicago. Nat Plants, 2023. 9(10): p. 1734-1748.