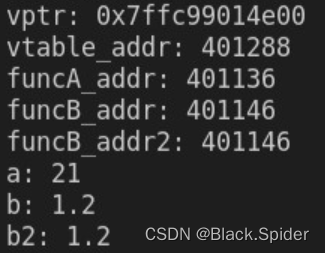
在这篇文章:【3D 图像分割】基于 Pytorch 的 VNet 3D 图像分割2(基础数据流篇) 的最后,我们提到了:
在采用vent模型进行3d数据的分割训练任务中,输入大小是16*96*96,这个的裁剪是放到Dataset类里面裁剪下来的image和mask。但是在训练时候发现几个问题:
- 加载数据耗费了很长时间,从启动训练,到正式打印开始按batch循环,这段时间就有30分钟
batch=64, torch.utils.data.DataLoader里面的num_workers=8,训练总是到8的倍数时候,要停顿较长时间等待- 4个GPU并行训练的,GPU的利用率长时间为0,偶尔会升上去,一瞬间又为0
free -m查看的内存占用,发现buff和cache会逐步飙升,慢慢接近占满。
请问出现这种情况,会是哪里存在问题啊?模型是正常训练和收敛拟合的也比较好,就是太慢了。分析myDataset数据读取的代码,有几个地方可能是较为耗时,和占用内存的地方:
getAnnotations函数,需要从csv文件中获取文件名和结节对应坐标,最后存储为一个字典,这个是始终要占着内存空间的;getNpyFile_Path函数,dataFile_paths和labelFile_paths都需要调用,有些重复了,这部分的占用是可以降低一倍的;get_annos_label函数,也是一样的问题,有些重复了,这部分的占用是可以降低一倍的。
上面这几个函数,都是在类的__init__阶段就完成的,这种多次的循环,可能是在开始batch循环前这部分时间,耗费时间的主要原因;其次,由于重复占用内存,进一步加剧了性能降低,使得后续的训练变的比较慢。
为了解决上面的这些问题,产生了本文2.0 的Dataset数据加载的版本,其最大的改动就是将原本从csv文件获取结节坐标的形式,改为从npy文件中获取。这样,image、mask、Bbox都是一一对应的单个文件了。从后续的实际训练发现,也确实是如此,解决了这个耗时的问题,让训练变的很快。
所以,只要我们将牟定的值进行精简,减少__init__阶段的内存占用,这个问题就应该可以完美解决了。所以,本篇就是遵照这个原则,尽量的在数据预处理阶段,就把能不要的就丢弃,只留下最简单的一一结构。将预处理前置,避免在构建数据阶段调用。
LUNA16数据的预处理,可以参照这里,本篇就是通过这里方式,产生的数据,如下:
- 【3D 图像分割】基于 Pytorch 的 VNet 3D 图像分割6(数据预处理)
- 【3D 图像分割】基于 Pytorch 的 VNet 3D 图像分割7(数据预处理)
- 【3D 图像分割】基于 Pytorch 的 VNet 3D 图像分割8(CT肺实质分割)
- 【3D 图像分割】基于 Pytorch 的 VNet 3D 图像分割9(patch 的 crop 和 merge 操作)
一、搭设数据流框架
在pytorch中,构建训练用的数据流,都遵循下面这样一个结构。其中主要的思路是这样的:
- 在
__init__中,是类初始化阶段,就执行的。在这里需要牟定某个值,将训练需要的内容,都获取到,但尽量少的占用内容和花费时间; - 在
__getitem__中,会根据__init__牟定的那个值,获取到一个图像和标签信息,读取和增强等等操作,最后返回Tensor值; __len__返回的是一个epoch训练牟定值的长度。
下面就是一个简易的框架结构,留作参考,后续的构建数据流,都可以对这里补充。
class myDataset_v3(Dataset):
def __init__(self, data_dir, isTrain=True):
self.data = []
if isTrain:
self.data ···
else:
self.data ···
def __len__(self):
return len(self.data)
def __getitem__(self, index):
# ********** get file dir **********
image, label = self.data[index] # get whole data for one subject
# ********** change data type from numpy to torch.Tensor **********
image = torch.from_numpy(image).float()
label = torch.from_numpy(label).float()
return image, label
在这篇文章中,对这个类里面的参数,进行了详细的介绍,感兴趣的可以直达去学习:【BraTS】Brain Tumor Segmentation 脑部肿瘤分割3(构建数据流)
二、完善框架内容
相信通过前面6、7、8、9四篇博客的介绍,你已经将Luna16的原始数据集,处理成了一一对应的,我们训练所需要的数据形式,包括:
_bboxes.npy:记录了结节中心点的坐标和半径;_clean.nrrd:CT原始图像数组;_mask.nrrd:标注文件mask数组,和_clean.nrrd的shape一样;
还包括一些其他的.npy,记录的都是整个变换阶段的一些量,在训练阶段是使用不到的,这里就不展开了。最最关注的就是上面三个文件,并且是根据seriesUID一一对应的。
如果是这样的数据情况下,我们构建myDataset_v3(Dataset)数据量,思考:在__init__阶段,可以以哪个为锚点,尽量少占用内存的情况下,将所需要的图像、标注信息都可以在__getitem__阶段,依次获取到呢?
那就是seriesUID的文件名。他是可以一拖三的,并且一个列表就可以了,这样是最节省内存的方式。于是我们在__init__阶段的定义如下:
class myDataset_v3(Dataset):
def __init__(self, data_dir, crop_size=(16, 96, 96), isTrain=False):
self.bboxesFile_path = []
for file in os.listdir(data_dir):
if '_bboxes.npy' in file:
self.bboxesFile_path.append(os.path.join(data_dir, file))
self.crop_size = crop_size
self.crop_size_z, self.crop_size_h, self.crop_size_w = crop_size
self.isTrain = isTrain
然后在__len__的定义,就自然而然的知道了,如下:
def __len__(self):
return len(self.bboxesFile_path)
最为重要,且最难的,也就是__getitem__的定义,在这里需要做一下几件事情:
- 获取各个文件的路径;
- 获取文件对应的数据;
- 裁剪出目标
patch; - 数组转成
Tensor。
然后,在定义__getitem__中,就发现了问题,如下:
def __getitem__(self, index):
bbox_path = self.bboxesFile_path[index]
img_path = bbox_path.replace('_bboxes.npy', '_clean.nrrd')
label_path = bbox_path.replace('_bboxes.npy', '_mask.nrrd')
img, img_shape = self.load_img(img_path)
label = self.load_mask(label_path)
zyx_centerCoor = self.getBboxes(bbox_path)
def getBboxes(self, bboxFile_path):
bboxes_array = np.load(bboxFile_path, allow_pickle=True)
bboxes_list = bboxes_array.tolist()
xyz_list = [[zyx[0], zyx[2], zyx[1]] for zyx in bboxes_list]
return random.choice(xyz_list)
主要是因为一个_bboxes.npy记录的结节坐标点,并不只有一个结节。如果将获取bbox的放到__getitem__,就会发现他一次只能裁剪出一个patch,就不可能对多个结节的情况都处理到。所以我这里采用了random.choice的方式,随机的选择一个结节。
但是,这种方式是不好的,因为他会降低结节在学习过程中出现的次数,尽管是随机的,但是相当于某些类型的数据量变少了。同样学习的epoch次数下,那些只有一个结节的,就被学习的次数相对变多了。
为了解决这个问题,直接将结节数与文件名一一对应起来,这样对于每一个结节来说,机会都是均等的了。代码如下所示:
import os
import random
import numpy as np
import matplotlib.pyplot as plt
from tqdm import tqdm
import torch
from torch.utils.data import Dataset
import nrrd
import cv2
class myDataset_v3(Dataset):
def __init__(self, data_dir, crop_size=(16, 96, 96), isTrain=False):
self.dataFile_path_bboxes = []
for file in os.listdir(data_dir):
if '_bboxes.npy' in file:
one_path_bbox_list = self.getBboxes(os.path.join(data_dir, file))
self.dataFile_path_bboxes.extend(one_path_bbox_list)
self.crop_size = crop_size
self.crop_size_z, self.crop_size_h, self.crop_size_w = crop_size
self.isTrain = isTrain
def __getitem__(self, index):
bbox_path, zyx_centerCoor = self.dataFile_path_bboxes[index]
img_path = bbox_path.replace('_bboxes.npy', '_clean.nrrd')
label_path = bbox_path.replace('_bboxes.npy', '_mask.nrrd')
img, img_shape = self.load_img(img_path)
# print('img_shape:', img_shape)
label = self.load_mask(label_path)
# print('zyx_centerCoor:', zyx_centerCoor)
cutMin_list = self.getCenterScope(img_shape, zyx_centerCoor)
if self.isTrain:
rd = random.random()
if rd > 0.5:
cut_list = [cutMin_list[0], cutMin_list[0]+self.crop_size_z, cutMin_list[1], cutMin_list[1]+self.crop_size_h, cutMin_list[2], cutMin_list[2]+self.crop_size_w] ### z,y,x
start1, start2, start3 = self.random_crop_around_nodule(img_shape, cut_list, crop_size=self.crop_size, leftTop_ratio=0.3)
elif rd > 0.1:
start1, start2, start3 = self.random_crop_negative_nodule(img_shape, crop_size=self.crop_size)
else:
start1, start2, start3 = cutMin_list
else:
start1, start2, start3 = cutMin_list
img_crop = img[start1:start1 + self.crop_size_z, start2:start2 + self.crop_size_h,
start3:start3 + self.crop_size_w]
label_crop = label[start1:start1 + self.crop_size_z, start2:start2 + self.crop_size_h,
start3:start3 + self.crop_size_w]
# print('before:', img_crop.shape, label_crop.shape)
# 计算需要pad的大小
if img_crop.shape != self.crop_size:
pad_width = [(0, self.crop_size_z-img_crop.shape[0]), (0, self.crop_size_h-img_crop.shape[1]), (0, self.crop_size_w-img_crop.shape[2])]
img_crop = np.pad(img_crop, pad_width, mode='constant', constant_values=0)
if label_crop.shape != self.crop_size:
pad_width = [(0, self.crop_size_z-label_crop.shape[0]), (0, self.crop_size_h-label_crop.shape[1]), (0, self.crop_size_w-label_crop.shape[2])]
label_crop = np.pad(label_crop, pad_width, mode='constant', constant_values=0)
# print('after:', img_crop.shape, label_crop.shape)
img_crop = np.expand_dims(img_crop, 0) # (1, 16, 96, 96)
img_crop = torch.from_numpy(img_crop).float()
label_crop = torch.from_numpy(label_crop).long() # (16, 96, 96) label不用升通道维度
return img_crop, label_crop
def __len__(self):
return len(self.dataFile_path_bboxes)
def load_img(self, path_to_img):
if path_to_img.startswith('LKDS'):
img = np.load(path_to_img)
else:
img, _ = nrrd.read(path_to_img)
img = img.transpose((0, 2, 1)) # 与xyz坐标变换对应
return img/255.0, img.shape
def load_mask(self, path_to_mask):
mask, _ = nrrd.read(path_to_mask)
mask[mask>1] = 1
mask = mask.transpose((0, 2, 1)) # 与xyz坐标变换对应
return mask
def getBboxes(self, bboxFile_path):
bboxes_array = np.load(bboxFile_path, allow_pickle=True)
bboxes_list = bboxes_array.tolist()
one_path_bbox_list = []
for zyx in bboxes_list:
xyz = [zyx[0], zyx[2], zyx[1]]
one_path_bbox_list.append([bboxFile_path, xyz])
return one_path_bbox_list
def getCenterScope0(self, img_shape, zyx_centerCoor):
cut_list = [] # 切割需要用的数
for i in range(len(img_shape)): # 0, 1, 2 → z,y,x
if i == 0: # z
a = zyx_centerCoor[-i - 1] - self.crop_size_z/2 # z
b = zyx_centerCoor[-i - 1] + self.crop_size_z/2 # y,z
else: # y, x
a = zyx_centerCoor[-i - 1] - self.crop_size_w/2
b = zyx_centerCoor[-i - 1] + self.crop_size_w/2
# 超出图像边界 1
if a < 0:
a = self.crop_size_z
b = self.crop_size_w
# 超出边界 2
elif b > img_shape[i]:
if i == 0:
a = img_shape[i] - self.crop_size_z
b = img_shape[i]
else:
a = img_shape[i] - self.crop_size_w
b = img_shape[i]
else:
pass
cut_list.append(int(a))
cut_list.append(int(b))
return cut_list
def getCenterScope(self, img_shape, zyx_centerCoor):
img_z, img_y, img_x = img_shape
zc, yc, xc = zyx_centerCoor
zmin = max(0, zc - self.crop_size_z // 3)
ymin = max(0, yc - self.crop_size_h // 2)
xmin = max(0, xc - self.crop_size_w // 2)
cutMin_list = [int(zmin), int(ymin), int(xmin)]
return cutMin_list
def random_crop_around_nodule(self, img_shape, cut_list, crop_size=(16, 96, 96), leftTop_ratio=0.3):
"""
:param img:
:param label:
:param center:
:param radius:
:param cut_list:
:param crop_size:
:param leftTop_ratio: 越大,阴性样本越多(需要考虑crop_size)
:return:
"""
img_z, img_y, img_x = img_shape
crop_z, crop_y, crop_x = crop_size
z_min, z_max, y_min, y_max, x_min, x_max = cut_list
# print('z_min, z_max, y_min, y_max, x_min, x_max:', z_min, z_max, y_min, y_max, x_min, x_max)
z_min = max(0, int(z_min-crop_z*leftTop_ratio))
z_max = min(img_z, int(z_min + crop_z*leftTop_ratio))
y_min = max(0, int(y_min-crop_y*leftTop_ratio))
y_max = min(img_y, int(y_min+crop_y*leftTop_ratio))
x_min = max(0, int(x_min-crop_x*leftTop_ratio))
x_max = min(img_x, int(x_min+crop_x*leftTop_ratio))
z_start = random.randint(z_min, z_max)
y_start = random.randint(y_min, y_max)
x_start = random.randint(x_min, x_max)
return z_start, y_start, x_start
def random_crop_negative_nodule(self, img_shape, crop_size=(16, 96, 96), boundary_ratio=0.5):
img_z, img_y, img_x = img_shape
crop_z, crop_y, crop_x = crop_size
z_min = 0#crop_z*boundary_ratio
z_max = img_z-crop_z#img_z - crop_z*boundary_ratio
y_min = 0#crop_y*boundary_ratio
y_max = img_y-crop_y#img_y - crop_y*boundary_ratio
x_min = 0#crop_x*boundary_ratio
x_max = img_x-crop_x#img_x - crop_x*boundary_ratio
z_start = random.randint(z_min, z_max)
y_start = random.randint(y_min, y_max)
x_start = random.randint(x_min, x_max)
return z_start, y_start, x_start
上述就是本次改写后新的数据流完整代码,没有加入数据增强的操作。在训练时,引入了三种多样性:
- 确保
mask有结节目标的情况下,随机的变换结节在patch中的位置; - 全图随机的进行裁剪,主要是产生负样本;
- 直接使用结节为中心点的方式进行裁剪。
这样做的目的,其实是考虑到结节在patch中的位置,可能会影响到最终的预测。因为最后我们在使用的推理阶段,其实是不知道结节在图像中的哪个位置的,只能遍历所有的patch,然后再将预测的结果拼接成一个完整的mask,进而对mask的处理,知道了所有结节的位置。
这就要求结节无论是出现在图像中的任何位置,都需要找到他,并且尽量少的假阳性。
这块是很少看到论文涉及到的内容,我不清楚是不是论文只关于了指标,而忘记了假阳性这样一个附加产物。还有就是这些patch的获取方式,是预先裁剪下来,直接读取patch数组的形式,进行训练的。这种也不好,多样性不够,还比较的麻烦。
这一小节还要讲的,就是getCenterScope和random_crop_around_nodule两个函数。getCenterScope中为什么整除3,是因为多次查看,总结出来的。如果是整除2,就发现所有的结节,都偏下,这点的原因,还没有想明白。知道的求留言。
如果是一个二维的平面,已知中心点,那么找到左上角的最小值,那就应该是中心点坐标,减去二分之一的宽高。但是,在z轴也采用减去二分之一的,发现所有裁剪出来的结节就很靠下。

所以,这里采用了减去三分之一,让他在z轴上,往上移动了一点。这里的疑问还没有搞明白,知道的评论区求指教。
random_crop_around_nodule是控制了裁剪左上角最小值和最大值的坐标,在这个区间内随机的确定,进而使得结节的裁剪,更加的多样性。如下图所示:
我只要想让每一次的裁剪都有结节在,只需要结节左上角的坐标,落在一定的区间内即可。leftTop_ratio参数,就是用于控制左上角的点,远离左上角的距离。
这个值需要自己根据patch的大小自己决定,多次查看很重要。
三、验证数据流
构建好数据量的类函数,还不能算完。因为你不知道此时的数据流,是不是符合你要求的。所以如果能够模拟训练过程,提前看看每一个patch的结果,那就再好不过了。
本章节就是这个目的,我们把图像和mask通通打出来看看,这样就知道是否存在问题了。查看的方法也比较的简单,可以抄过去用到之后自己的项目里。
def getContours(output):
img_seged = output.numpy().astype(np.uint8)
img_seged = img_seged * 255
# ---- Predict bounding box results with txt ----
kernel = np.ones((5, 5), np.uint8)
img_seged = cv2.dilate(img_seged, kernel=kernel)
_, img_seged_p = cv2.threshold(img_seged, 127, 255, cv2.THRESH_BINARY)
try:
_, contours, _ = cv2.findContours(np.uint8(img_seged_p), cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_SIMPLE)
except:
contours, _ = cv2.findContours(np.uint8(img_seged_p), cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_SIMPLE)
return contours
if __name__=='__main__':
data_dir = r"./valid"
dataset_valid = myDataset_v3(data_dir, crop_size=(48, 96, 96), isTrain=False) # 送入dataset
valid_loader = torch.utils.data.DataLoader(dataset_valid, # 生成dataloader
batch_size=1, shuffle=False,
num_workers=0) # 16) # 警告页面文件太小时可改为0
print("valid_dataloader_ok")
print(len(valid_loader))
for batch_index, (data, target) in tqdm(enumerate(valid_loader)):
name = dataset_valid.dataFile_path_bboxes[batch_index]
print('name:', name)
print('image size ......')
print(data.shape) # torch.Size([batch, 1, 16, 96, 96])
print('label size ......')
print(target.shape) # torch.Size([2])
# 按着batch进行显示
for i in range(data.shape[0]):
onePatch = data[i, 0, :, :]
onePatch_target = target[0, :, :, :]
print('one_patch:', onePatch.shape, np.max(onePatch.numpy()), np.min(onePatch.numpy()))
fig, ax = plt.subplots(6, 8, figsize=[14, 16])
for i in range(6):
for j in range(8):
one_pic = onePatch[i * 4 + j]
img = one_pic.numpy()*255.0
# print('one_pic img:', one_pic.shape, np.max(one_pic.numpy()), np.min(one_pic.numpy()))
one_mask = onePatch_target[i * 4 + j]
contours = getContours(one_mask)
for contour in contours:
x, y, w, h = cv2.boundingRect(contour)
xmin, ymin, xmax, ymax = x, y, x + w, y + h
# print('contouts:', xmin, ymin, xmax, ymax)
cv2.drawContours(img, contour, -1, (0, 0, 255), 2)
# cv2.rectangle(img, (int(xmin), int(ymin)), (int(xmax), int(ymax)), (0, 0, 255),
# thickness=1)
ax[i, j].imshow(img, cmap='gray')
ax[i, j].axis('off')
# print('one_target:', onePatch.shape, np.max(onePatch.numpy()), np.min(onePatch.numpy()))
fig, ax = plt.subplots(6, 8, figsize=[14, 16])
for i in range(6):
for j in range(8):
one_pic = onePatch_target[i * 4 + j]
# print('one_pic mask:', one_pic.shape, np.max(one_pic.numpy()), np.min(one_pic.numpy()))
ax[i, j].imshow(one_pic, cmap='gray')
ax[i, j].axis('off')
plt.show()
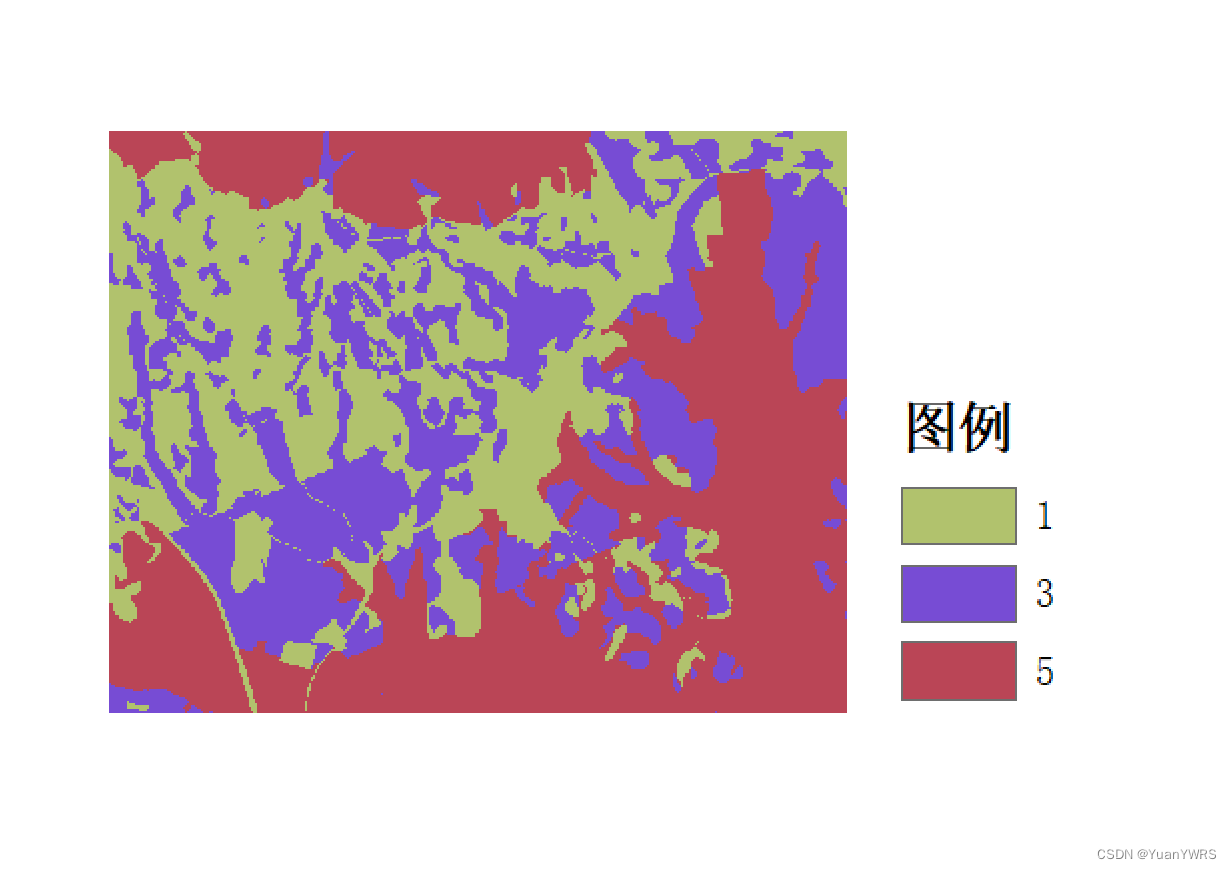
显示出来的图像如下所示:

你可以多看几张,看的多了,也就顺便给验证了结节裁剪的是否有问题。同时,也可以采用训练模型,看看在训练情况下,阳性带结节的样本,和全是黑色的,没有结节的样本占到多少。这也为我们改上面的代码,提供了参考标准。
四、总结
本文其实是对前面博客数据流问题的一个总结,和找到解决问题的方法了。同时将一个验证数据量的过程给展示了出来,方便我们后续更多的其他任务,都是很有好处的。
如果你是一名初学者,我相信该收获满满。如果你是奔着项目来的,那肯定也找到了思路。数据集的差异,主要体现在前处理上,而到了训练阶段,本篇可以帮助你快速的动手。
最后,留下你的点赞和收藏。如果有问题,欢迎评论和私信。后续会将训练和验证的代码进行介绍,这部分同样是重点。