近年来,基于扩增子测序进行物种的功能预测是研究微生物群落功能的主要方面,目前最常用的软件包括Tax4Fun以及PICRUSt2。关于这两款软件的使用方法详可参见凌波微课|扩增子研究第十六讲:扩增子测序结果中的物种功能预测。
Tax4Fun使用最近邻匹配方法,但只有在大量参考基因组可用时才能正常运行;而PICRUSt2依赖于系统发育树和祖先状态重建来预测基因功能,这一点一直存在争议。同时,由于基于部分区域测序的16S rRNA扩增子数据无法在属级之外实现准确分类,因此这些工具可能会输出大量假阳性的结果。
基于这些问题,2021年《Genomics》期刊最新发布了一款综合性的16S rRNA扩增子数据功能预测数据库——MicFunPred。
MicFunPred是一个独立且易于访问的网络服务器(http://micfunpred.microdm.net.in/),可仅使用一组核心基因在属级分类学中预测微生物群落的功能潜力,从而最大限度地减少假阳性预测。
MicFunPred的工作流程如下:
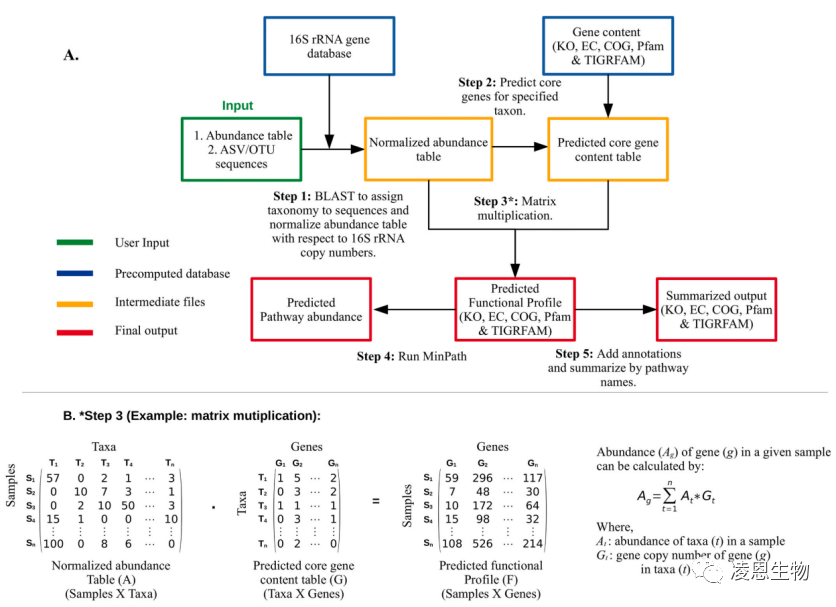
图1 MicFunPred功能预测工作流程
研究者从微生物基因组(IMG)数据库(https://img.jgi.doe.gov/faq.html)下载了人类、水生、植物、陆地和哺乳动物生态系统的共32,453个基因组数据。使用来自这些基因组的16S序列以及Greengenes (v13_5)、SILVA (v132)和EZBiocloud(v2018.05)数据库的序列开发了MicFunPred综合的16S rRNA基因数据库。MicFunPred能够预测KEGG Orthology(KO)、酶委员会(EC)、Pfam、TIGRFAM和COG方面的功能特征。
图2 MicFunPred数据库在线web服务器
研究者使用来自多个扩增子和宏基因组的项目数据来检测MicFunPred的性能,同时还在模拟数据集上进行了测试。在模拟数据集上,MicFunPred显示出比PICRUSt2、Piphillin和Tax4Fun2更高的相关性(Spearman=0.89)和最低的假阳性率。而在七个真实数据集上,MicFunPred平均相关性为0.75。与其他工具相比,MicFunPred速度更快,需要的计算能力较低,并且性能更好。

图3 MicFunPred和其他工具在模拟数据集上的性能比较
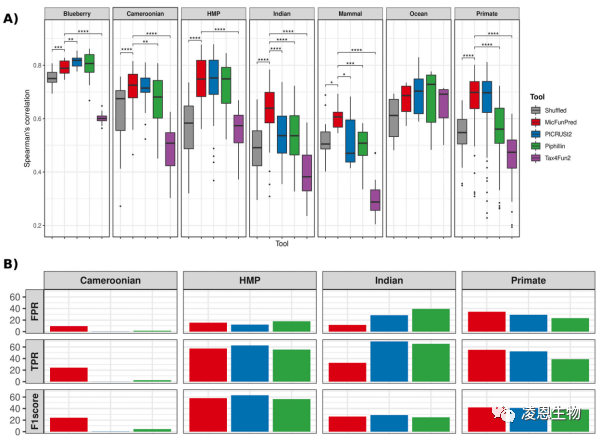
图4 MicFunPred和其他工具在真实数据集上的性能比较
-
MicFunPred网站地址如下:
http://micfunpred.microdm.net.in/
-
数据库下载地址如下:
https://github.com/microDM/MicFunPred

参考文献
MicFunPred: A conserved approach to predict functional profiles from 16S rRNA gene sequence data. Genomics, 2021.




![执行docker restart [CONTAINER ID]命令时会把之前的参数都带上吗](https://img-blog.csdnimg.cn/7f04f0607c1c43ec8f4b383d2076bd57.png)