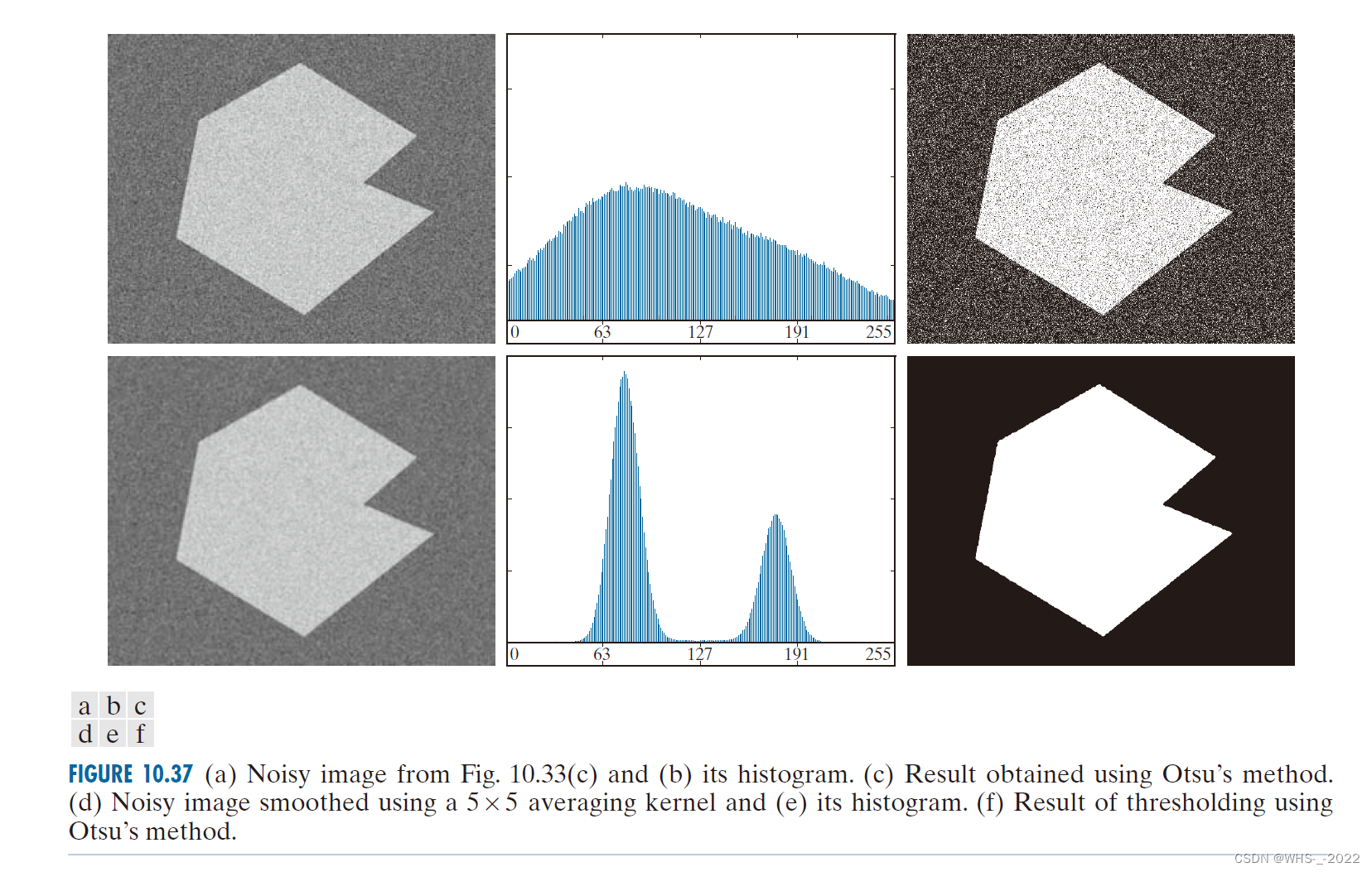
扫码关注下方公粽号,回复推文合集,获取400页单细胞学习资源!

本文共计1884字,阅读大约需要6分钟,目录如下:
-
Symphony 包基本介绍
-
Symphony 包安装
-
Symphony 包使用
-
- 1.使用已有的参考数据集进行细胞注释
- 2.使用自定义的参考数据集进行细胞注释
-
小结
-
获取本文数据和代码
-
代码参考
-
往期单细胞系统教程
Symphony 包基本介绍
Symphony 最早发表于 2021 年 Nature Communications 杂志上的一篇文章,文章题目为"Efficient and precise single-cell reference atlas mapping with Symphony"。发表该文章的 Raychaudhuri Lab 曾于 2019 年在 Nature Methods 杂志上发表单细胞数据整合算法 Harmony。
Symphony 算法首先将 Reference 数据集嵌入 UMAP 空间中,再将 Query 数据集(待注释数据集)嵌入到与 Reference 数据集相同的 UMAP 空间,接着使用 KNN 算法,根据 Reference 数据集,计算距离 Query 细胞最近的 K 个 Reference 细胞邻居,确定最可能的 cell type。
Symphony 具体流程如下:

图 1 Symphony工作流程图(来源:Nat Commun 12, 5890 (2021).)
Symphony 包安装
# 方法一
install.packages("symphony")
# 方法二
# install.packages("devtools")
devtools::install_github("immunogenomics/symphony")
Symphony 包使用
注:
- 读入数据均为 log(CP10k+1)处理过的 logCounts 矩阵。
- 需要保证 Query 数据集与 Reference 数据集处理流程一致。
1.使用已有的参考数据集进行细胞注释
Symphony 包提供以下 8 个参考数据集
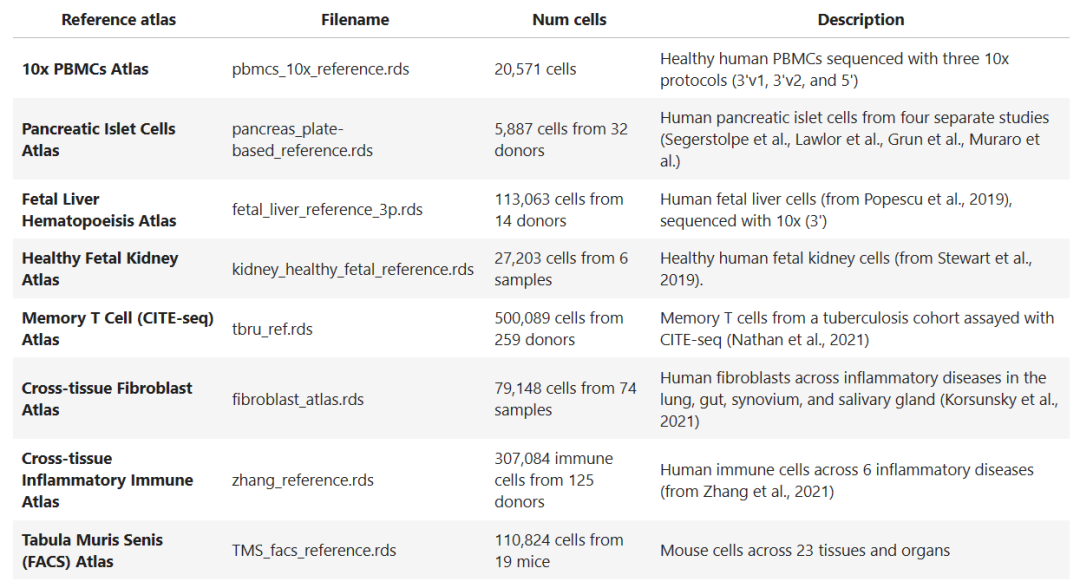
图 2 Symphony提供的参考数据集
-
下载地址:Symphony pre-built single-cell reference atlases | Zenodo
https://zenodo.org/record/5090425#.YOqe_hNKhTY
-
读入方法:
readRDS(参考数据集文件路径)
注:如果想要将 Query 数据 Map(映射)到 Reference 数据集的 UMAP 空间中,则需要下载对应的 UMAP 计算模型
uwot_model
- 通过
reference$save_uwot_path查看uwot_model存放路径,需要将下载的uwot_model放入指定路径
在本次示例中,Reference 数据集由 4 项研究中的人类胰岛细胞构成。Query 数据集来自于 Baron et al. (2016)的人类胰岛细胞数据 。
# 载入相关R包
suppressPackageStartupMessages({
library(symphony)
library(tidyverse)
library(data.table)
library(matrixStats)
library(Matrix)
library(plyr)
library(dplyr)
## 画图包
library(ggplot2)
library(ggthemes)
library(ggrastr)
library(RColorBrewer)
library(patchwork)
library(ggrepel)
})
source("colors.R") # 定义绘图颜色
ref_pancreas = readRDS("data/pancreas_plate-based_reference.rds")
## 绘制参考数据集UMAP图
p = plotReference(
reference = ref_pancreas, # 参考数据集
as.density = F, # True则绘制密度图,False则绘制散点图
title = "Symphony Reference",
color.by = "cell_type",
celltype.colors = pancreas_colors, # 指定颜色
show.legend = T,
show.labels = T,
show.centroids = F
)
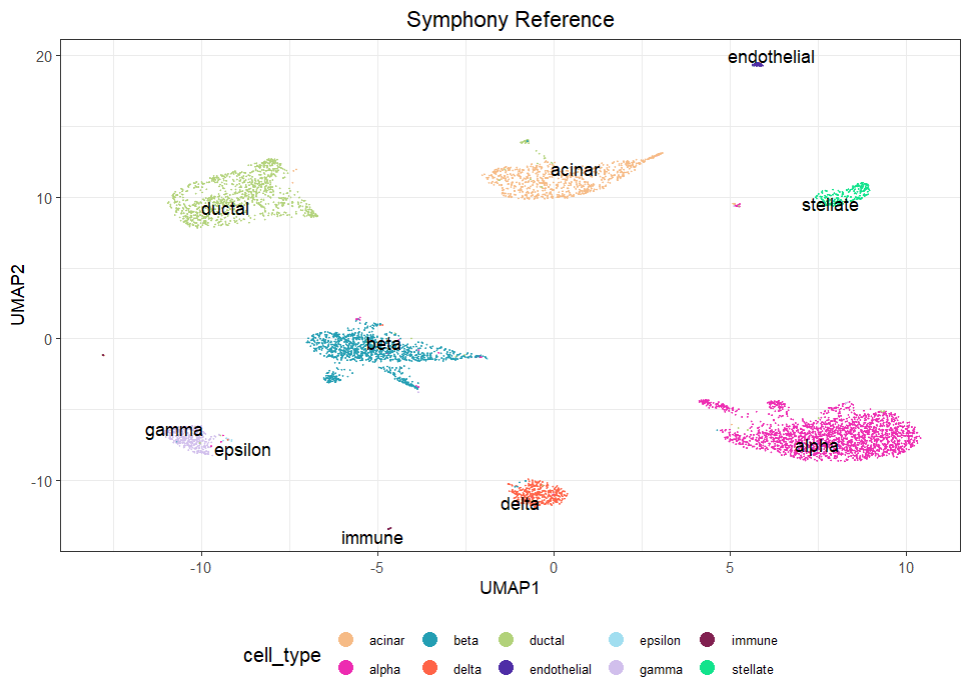
图 3 人类胰岛细胞参考数据集UMAP分布
## 读入query数据集
human_exp_norm = readRDS("data/pancreas_baron_human_exp.rds")
human_metadata = readRDS("data/pancreas_baron_human_metadata.rds")
## 将Query数据集映射到参考数据集
ref_pancreas$save_uwot_path # 查看uwot_model保存路径
query = mapQuery(
exp_query = human_exp_norm,
metadata_query = human_metadata,
ref_obj = ref_pancreas,
vars = c("dataset", "species", "species_donor"),
do_normalize = F, # 是否需要logNormalize
do_umap = T # map到参考数据集的UMAP空间
)
## 根据reference数据集进行注释
query = knnPredict(query, reference, reference$meta_data$cell_type, k = 5)
## 查看Query数据集自动注释结果
colnames(query$umap) = c("UMAP1", "UMAP2")
umap_labels = cbind(query$meta_data, query$umap)
p1 = umap_labels %>%
sample_frac(1L) %>%
ggplot(aes(x = UMAP1, y = UMAP2, col = cell_type_pred_knn)) +
geom_point_rast(size = 0.3, stroke = 0.2, shape = 16) +
theme_bw() +
labs(title = 'Query after Symphony', color = '') +
theme(plot.title = element_text(hjust = 0.5)) +
theme(
legend.position = "none",
legend.text = element_text(size=11),
legend.title = element_text(size = 12)
) +
scale_colour_manual(values = pancreas_colors) +
guides(colour = guide_legend(override.aes = list(size = 3), ncol = 5)) +
theme(strip.text.x = element_text(size = 14))
p1 + p
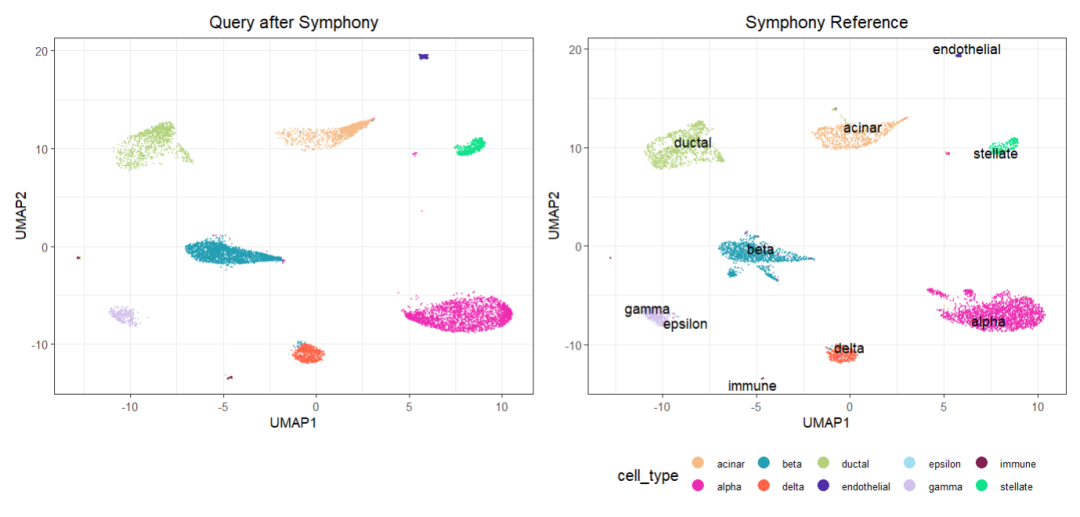
图 4 待注释数据集(Query)注释结果UMAP分布(左);参考数据集UMAP分布(右)
## 与Query数据集提供的注释信息比较
cell_type <- query$meta_data$cell_type
cell_type_pred_knn <- query$meta_data$cell_type_pred_knn
cell_type <- as.character(cell_type)
cell_type_pred_knn <- as.character(cell_type_pred_knn)
table(cell_type == cell_type_pred_knn)
# FALSE TRUE
# 351 8218
2.使用自定义的参考数据集进行细胞注释
本次示例中,将来自 10x 3’v1 和 3’v2 测序的 PBMCs 数据集作为 Reference 数据集,将来自 10x 5’测序的 PBMCs 作为 Query 数据集。
# 读入R包
suppressPackageStartupMessages({
# Analysis
library(symphony)
library(harmony)
library(irlba)
library(tidyverse)
library(data.table)
library(matrixStats)
library(Matrix)
library(plyr)
library(dplyr)
library(tibble)
# Plotting
library(ggplot2)
library(ggthemes)
library(ggrastr)
library(RColorBrewer)
library(patchwork)
library(ggpubr)
library(scales)
})
source('utils.R') # 颜色和绘图函数封装
# 读入数据
exprs_norm = readRDS("data/exprs_norm_all.rds")
metadata = read.csv("data/meta_data_subtypes.csv", row.names = 1)
idx_query = which(metadata$donor == "5'") # 将来自5'测序的数据作为Query数据集
# 构建Reference数据集
ref_exp_full = exprs_norm[, -idx_query]
ref_metadata = metadata[-idx_query, ]
# 构建Query数据集
query_exp = exprs_norm[, idx_query]
query_metadata = metadata[idx_query, ]
# 将Reference构造成Symphony Reference对象
## 筛选高变基因,取高变基因的子集
var_genes = vargenes_vst(
ref_exp_full,
groups = as.character(ref_metadata[["donor"]]), # 按照groups条件分别寻找HVGs,再汇总
topn = 2000)
ref_exp = ref_exp_full[var_genes, ] # 过滤非高变基因
## 计算并保存每个基因表达量的均值和方差
vargenes_means_sds = tibble(symbol = var_genes, mean = Matrix::rowMeans(ref_exp))
vargenes_means_sds$stddev = symphony::rowSDs(ref_exp, vargenes_means_sds$mean)
## 使用算出的基因表达量均值和标准差对数据正态化
ref_exp_scaled = symphony::scaleDataWithStats(
ref_exp, vargenes_means_sds$mean,
vargenes_means_sds$stddev,
1
)
## Run SVD,保存gene loadings
set.seed(0)
s = irlba(ref_exp_scaled, nv = 20)
Z_pca_ref = diag(s$d) %*% t(s$v) # 每个细胞的主成分
loadings = s$u
## Harmony整合
set.seed(0)
ref_harmObj = harmony::HarmonyMatrix(
data_mat = t(Z_pca_ref), # 细胞的pca embedding矩阵
meta_data = ref_metadata,
theta = c(2), # harmony 校正参数theta,值越大校正强度越大
vars_use = c("donor"), # 批次来源
nclust = 100,
max.iter.harmony = 20,
return_object = T, # True时返回完整的Harmony对象,False时仅返回校正后的PCA embeddings
do_pca = F # 是否对输入计算PCA
)
## 将harmony对象转换成Symphony Reference对象
reference = symphony::buildReferenceFromHarmonyObj(
ref_harmObj, # HarmonyMatrix()函数的输出对象
ref_metadata, # 对应的metadata
vargenes_means_sds, # 基因名、表达量均值和方差
loadings, # 高变基因的PCA embedding矩阵,行为基因,列为主成分PCs
verbose = T, # Console进度可视化输出
do_umap = T, # 是否进行UMAP降维
save_uwot_path = "./testing_uwot_model_1" # UMAP降维模型的保存路径
)
reference$normalized_method = "log(CP10k+1)" # 记录标准化方法
## 可视化reference的UMAP
color_cluster = group.colors[6:12]
names(color_cluster) = unique(ref_metadata$cell_type)
umap_labels = cbind(ref_metadata, reference$umap$embedding)
umap_labels %>% ggplot(aes(UMAP1, UMAP2, color = cell_type)) +
geom_point_rast(size = 0.3, stroke = 0.2, shape = 16) +
scale_color_manual(values = color_cluster) +
theme_bw() +
theme(
legend.title = element_text(size = 14),
legend.text = element_text(size = 12),
strip.text.x = element_text(size = 20),
plot.title = element_text(hjust = 0.5)
) +
ggtitle(element_text("reference", hjust = 0.5)) +
guides(colour = guide_legend(override.aes = list(size = 8)))
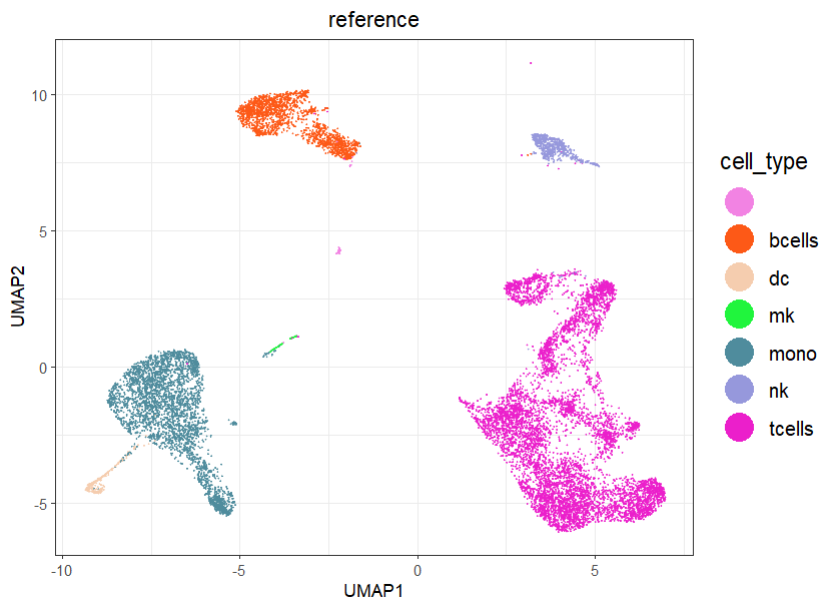
图 5 Reference数据集UMAP分布
# 将Query数据集MAP到Reference数据集上
query = mapQuery(
query_exp, # Query数据集的基因表达矩阵 (genes, cells)
query_metadata, # metadata (cell, attributes)
reference, # Symphony Reference对象
do_normalize = F, # 是否执行log(CP10k+1)标准化
do_umap = T # 是否将Query数据集map到Reference数据集的UMAP空间
)
## 使用KNN算法预测cell type
query = knnPredict(query, reference, reference$meta_data$cell_type, k = 5)
## 根据knn_prob调整cell_type_pred_knn
query_umap_labels = cbind(query$meta_data, query$umap)
query_umap_labels$cell_type_pred_knn = as.character(query_umap_labels$cell_type_pred_knn)
query_umap_labels$cell_type_pred_knn[query_umap_labels$cell_type_pred_knn_prob <= 0.5] = "unknown"
## 绘图
p1 <- query_umap_labels %>% ggplot(aes(UMAP1, UMAP2, color = cell_type_pred_knn)) +
geom_point_rast(size = 0.3, stroke = 0.2, shape = 16) +
scale_color_manual(values = color_cluster) +
theme_bw() +
theme(
legend.position = "none",
strip.text.x = element_text(size = 20),
plot.title = element_text(hjust = 0.5)
) +
ggtitle(element_text("query", hjust = 0.5)) +
guides(colour = guide_legend(override.aes = list(size = 8)))
p1 + p
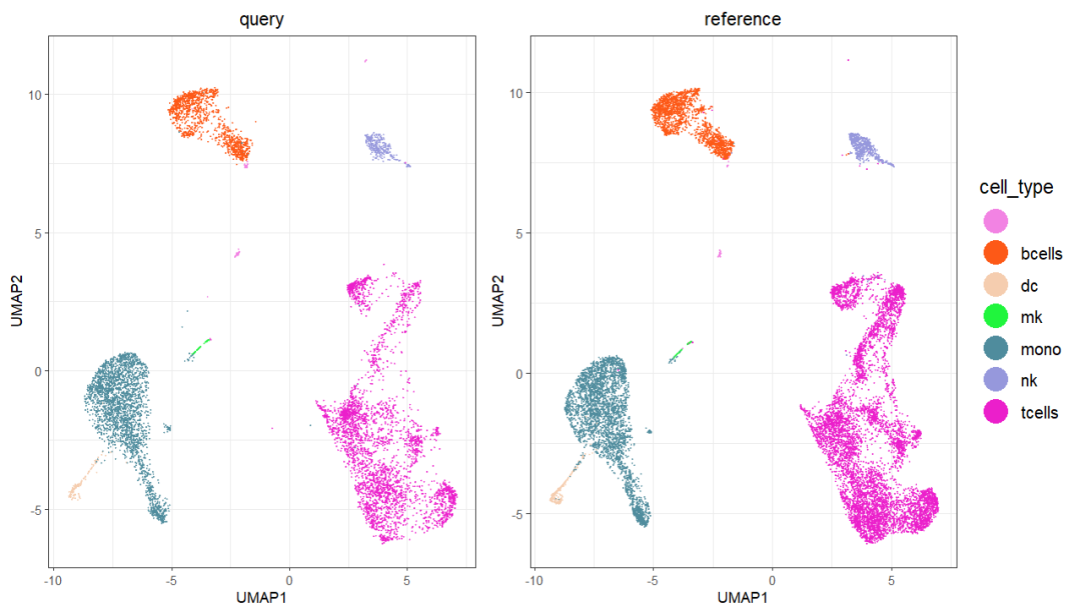
图 6 待注释数据集(Query)注释结果UMAP分布(左);参考数据集UMAP分布(右)
## 与Query数据集提供的注释信息比较
cell_type <- query$meta_data$cell_type
cell_type_pred_knn <- query$meta_data$cell_type_pred_knn
cell_type <- as.character(cell_type)
cell_type_pred_knn <- as.character(cell_type_pred_knn)
table(cell_type == cell_type_pred_knn)
# FALSE TRUE
# 82 7615
小结
可以看出,symphony 包注释结果较为可观。不过在本次分享,Query 和 Reference 数据集均来自同种组织,当使用不同组织的 Reference 数据集进行注释时,结果可能会不太乐观,最好根据自己的样本去搜集相关数据集构建同种的 Reference 数据集。
获取本文数据和代码
关注公粽号后,搜索推文【使用R包Symphony自动注释细胞类型】获取本文代码和数据

代码参考
https://github.com/immunogenomics/symphony/blob/main/vignettes/prebuilt_references_tutorial.ipynb
https://github.com/immunogenomics/symphony/blob/main/vignettes/pbmcs_tutorial.ipynb
往期单细胞系统教程
单细胞分析实录(1): 认识Cell Hashing
单细胞分析实录(2): 使用Cell Ranger得到表达矩阵
单细胞分析实录(3): Cell Hashing数据拆分
单细胞分析实录(4): doublet检测
单细胞分析实录(5): Seurat标准流程
单细胞分析实录(6): 去除批次效应/整合数据
单细胞分析实录(7): 差异表达分析/细胞类型注释
推荐几个细胞注释网站
如何批量查询marker基因(对应的蛋白)会不会在膜上表达?
单细胞分析实录(8): 展示marker基因的4种图形(一)
单细胞分析实录(9): 展示marker基因的4种图形(二)
单细胞分析实录(10): 消除细胞周期的影响
单细胞分析实录(11): inferCNV的基本用法
单细胞分析实录(12): 如何推断肿瘤细胞
单细胞分析实录(13): inferCNV结合UPhyloplot2分析肿瘤进化
单细胞分析实录(14): 细胞类型注释的另一种思路 — CellID
单细胞分析实录(15): 基于monocle2的拟时序分析
单细胞分析实录(16): 非负矩阵分解(NMF)检测细胞异质性
单细胞分析实录(17): 非负矩阵分解(NMF)代码演示
单细胞分析实录(18): 基于CellPhoneDB的细胞通讯分析及可视化 (上篇)
单细胞分析实录(19): 基于CellPhoneDB的细胞通讯分析及可视化 (下篇)
一个对接CellPhoneDB的R包
【代码更新】单细胞分析实录(20): 将多个样本的CNV定位到染色体臂,并画热图
【代码更新】单细胞分析实录(21): 非负矩阵分解(NMF)的R代码实现,只需两步,啥图都有