本文将介绍 24 年 5 月发布的 Alaphafold3,其以“使用 AlphaFold 3 进行生物分子相互作用的精确结构预测”为标题发表在《nature》上,通讯作者为 Jumper。
Jumper 具有物理、化学、生物和计算方面的丰富背景。Jumper 本科学的是物理和数学,本科毕业后,开始攻读理论凝聚态物理学博士学位,但很快就退学了。2008 年,他加入了一家做蛋白质模拟的公司,他开始在超级计算机上模拟蛋白质运动。2011 年,他再次尝试读研究生,他这次选择攻读芝加哥大学的理论化学学位。2017 年,刚刚完成博士学位的 Jumper 加入谷歌 DeepMind,担任研究科学家,领导了AlphaFold 1、2、3的开发。
本文内容主要分为三个部分:
-
AlphaFold3 模型结构; -
AlphaFold3 在不同类型的预测实验中的表现; -
在线使用 AlphaFold3
1. AlphaFold3 模型结构
和 AlphaFold2 类似,AlphaFold3 的框架由四部分组成:输入模块、特征提取模块、编码器、解码器。
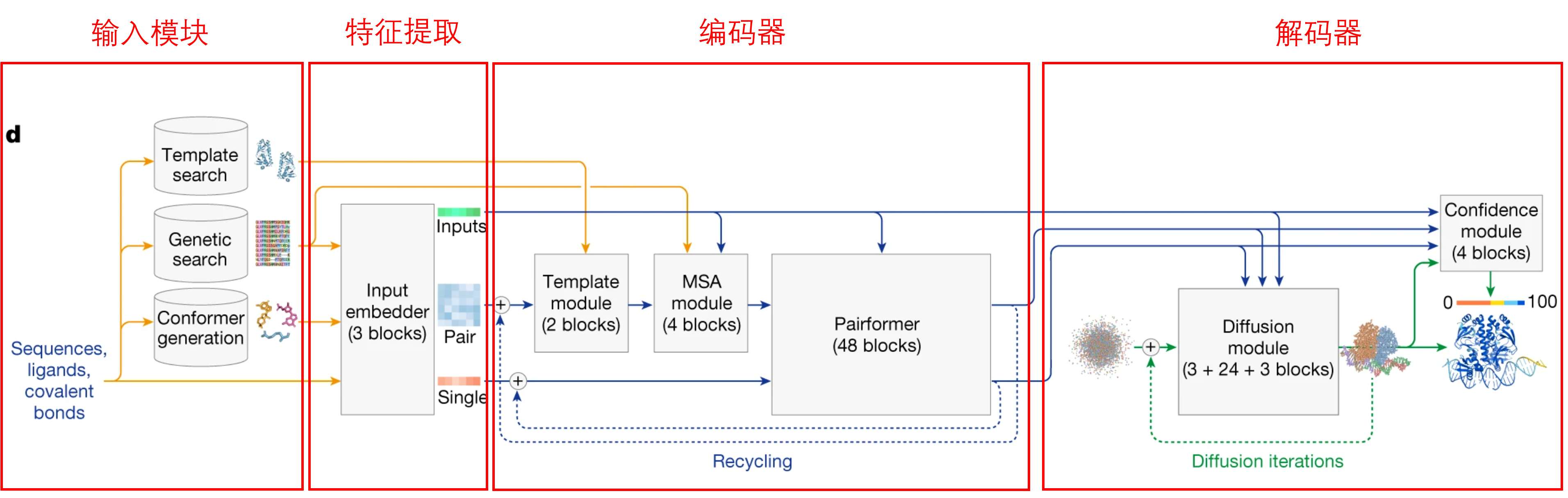

下面分别介绍这四部分:
1.1 输入模块
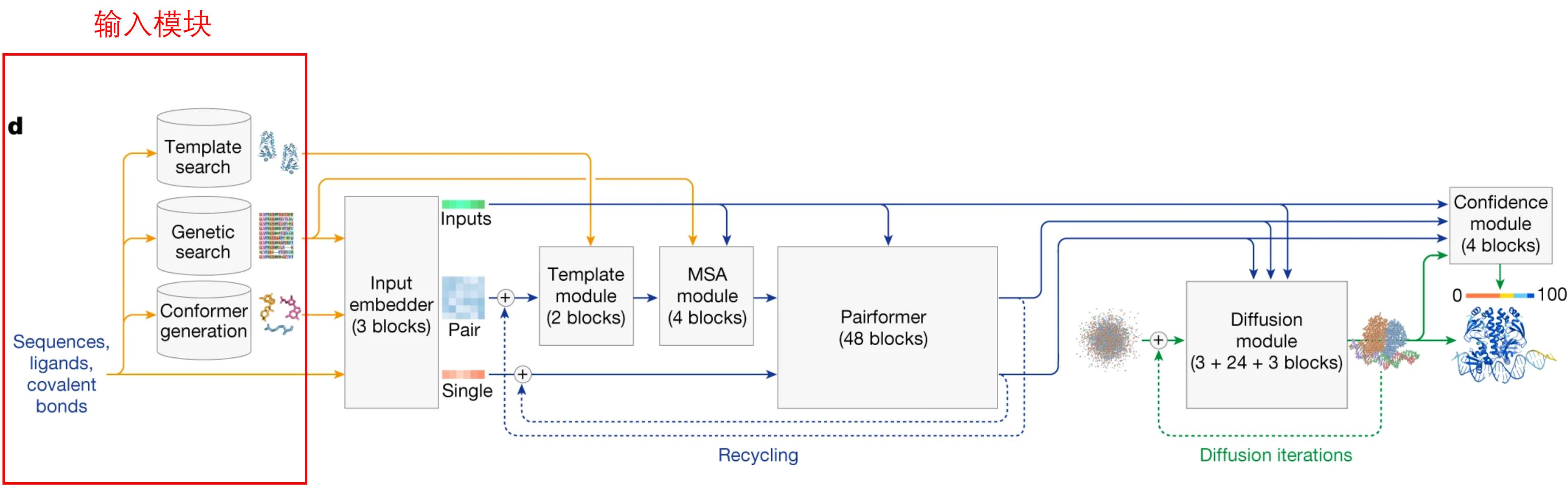
向模型输入序列、配体和共价键,然后执行以下三个操作:
-
从 PDB mmCIF 文件中检索模板结构; -
从 5 个数据库中检索蛋白质序列,从 3 个数据库中检索 RNA 序列,用于多序列比对(MSA),如下图所示:
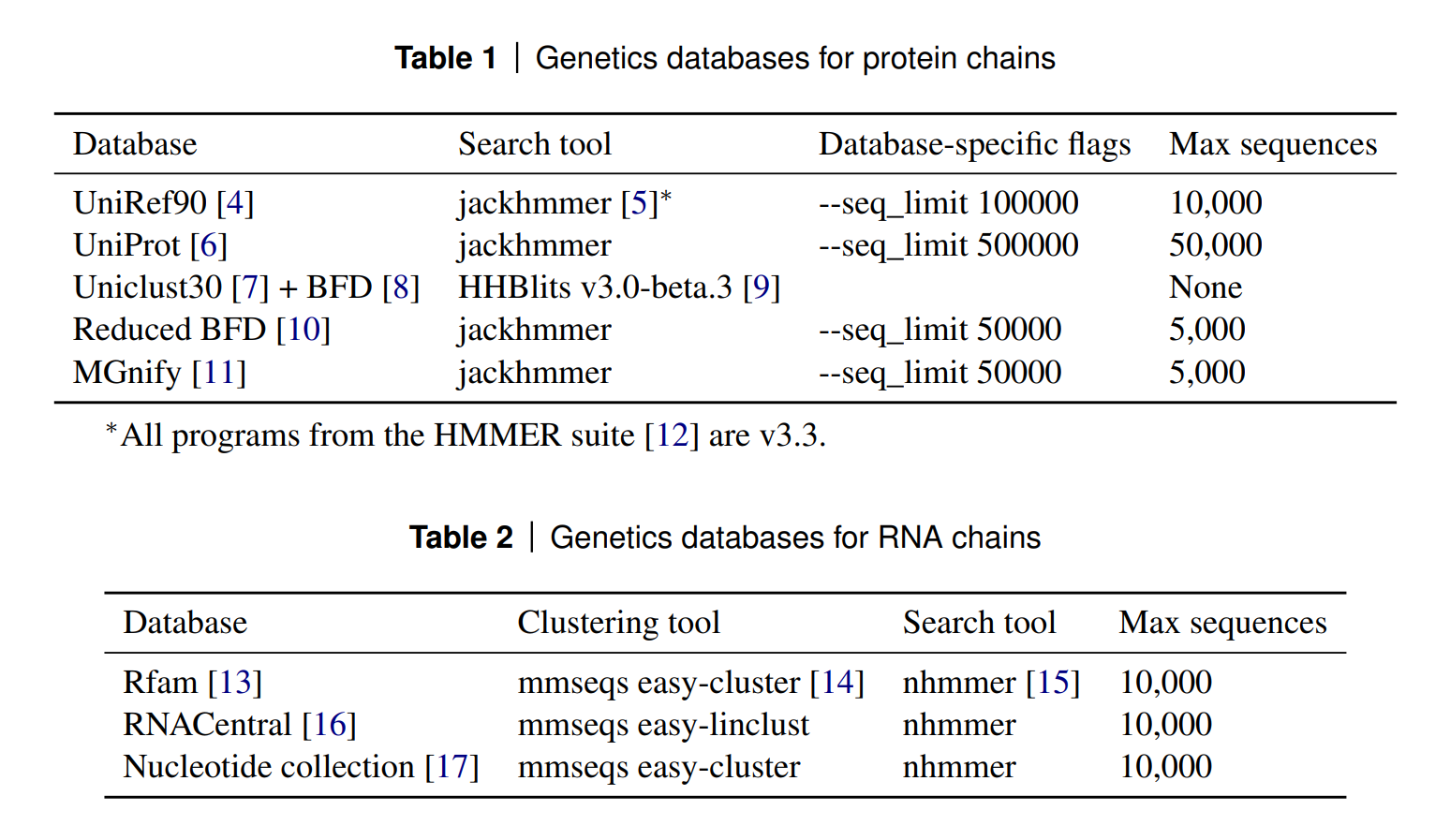
-
生成参考构象。
输入模块总结:输入模块就是将我们输入到模型的内容拿到数据库中去检索相似的模板结构和相似的序列,并生成参考构象,做好数据准备工作。检索的相似序列、生成的构象、输入序列、配体和共价键将会输入到特征提取模块。
1.2 特征提取模块
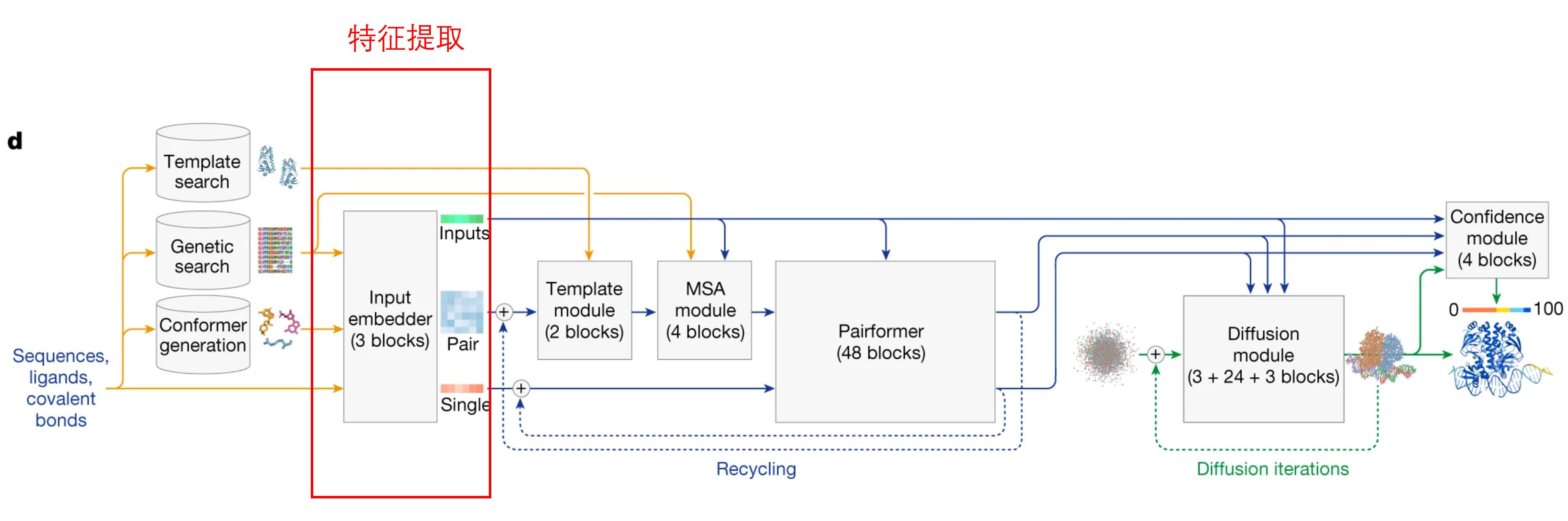
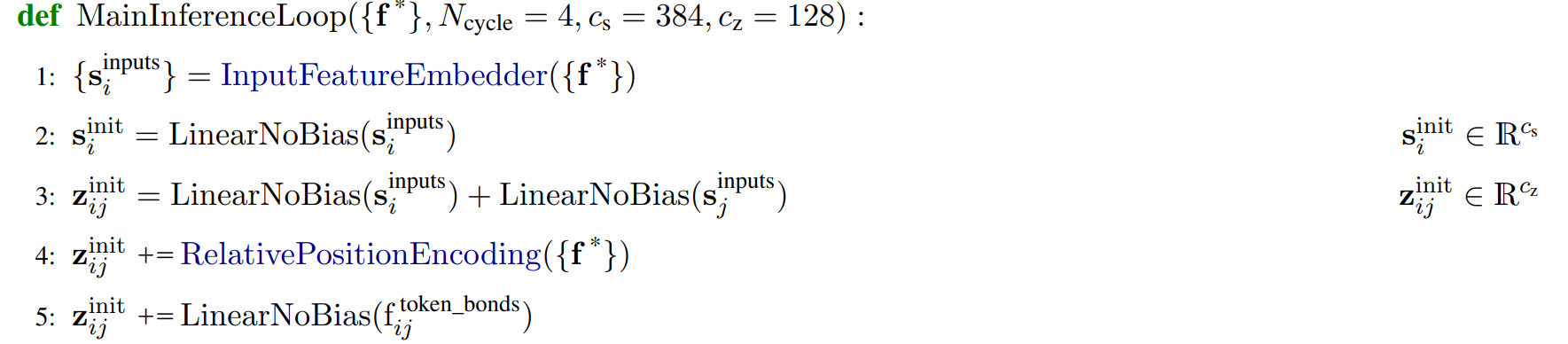
上图省略了将输入到特征提取模块的内容 token 化这一步骤,将输入内容转变为 token 之后,再将这些 token 输入到 embedder 中进行 embedding,然后输出 Input embeddings, Pair embeddings, single embeddings。
token:就是将生物语言用数字表示,举个例子就是,用 1-20 表示氨基酸。
Alphafold3 的 token 方法:
-
标准氨基酸残基:整体作为一个 token -
标准核苷酸残基:整体作为一个 token -
修改过的氨基酸或核苷酸残基:每个原子作为一个 token -
小分子配体:每个原子作为一个 token
embedding:嵌入,列举一个对象的若干个属性,并对属性进行赋值,产生一个描述这个对象的向量。例如,对于姚明这个对象,高属性赋值为 0.9,矮属性赋值为 -0.9,篮球属性赋值为 0.88,男人属性赋值为 1,有钱属性赋值为 0.3......。再例如,对于半胱氨酸这个氨基酸,氢键供体属性赋值为 0.5,氢键受体属性赋值为 0.6,具有可旋转化学键属性赋值为 0.3,表面电荷属性赋值为 -0.88......可以选择许多属性了来描述对象。具体到 Alphafold3,embedding 就是给输入模型的每一个生物序列单元的每一个属性赋值,每一个生物序列单元最终都由一个向量来表示,我们思考一下生物序列一般都有哪些属性,比如有亲水性、电负性、键角、氢键、可旋转共价键等,Alphafold3 为每个token 选择了 384 个属性,所以每个 token 会被 embedding 为一个 384 维的向量。对两两 token 之间的关系进行 embedding 时的属性表是另外选择的,有 128 个属性。
Input embeddings:由伪代码可以看出,它是输入的每个 token 的 embedding,embedding 维度为 384;从模型数据流可以看出,Input embeddings 不参与后面的循环更新,但是它参与后面每一个模块的计算。
single embeddings:由伪代码可以看出,它是由 Input embeddings 乘上一个权重矩阵而得出,embedding 维度为 384。
Pair embeddings:由伪代码可以看出,它表示两两 token 之间的关系,embedding 维度为 128;计算方法为:两个 token 分别乘以一个权重矩阵,结果相加,再加上一个输入 token 的相对位置编码,再加上这两个 token 之间的化学键的信息;Pair embeddings 包含了两两 token 之间的位置关系。
通俗总结特征提取模块:将输入到特征提取模块的内容表示为计算机能看懂的数字,然后再细致地列出他们都有哪些特点,以及他们彼此之间的关系怎么样,然后将这些得到的信息作为编码器的输入内容。
1.3 编码器
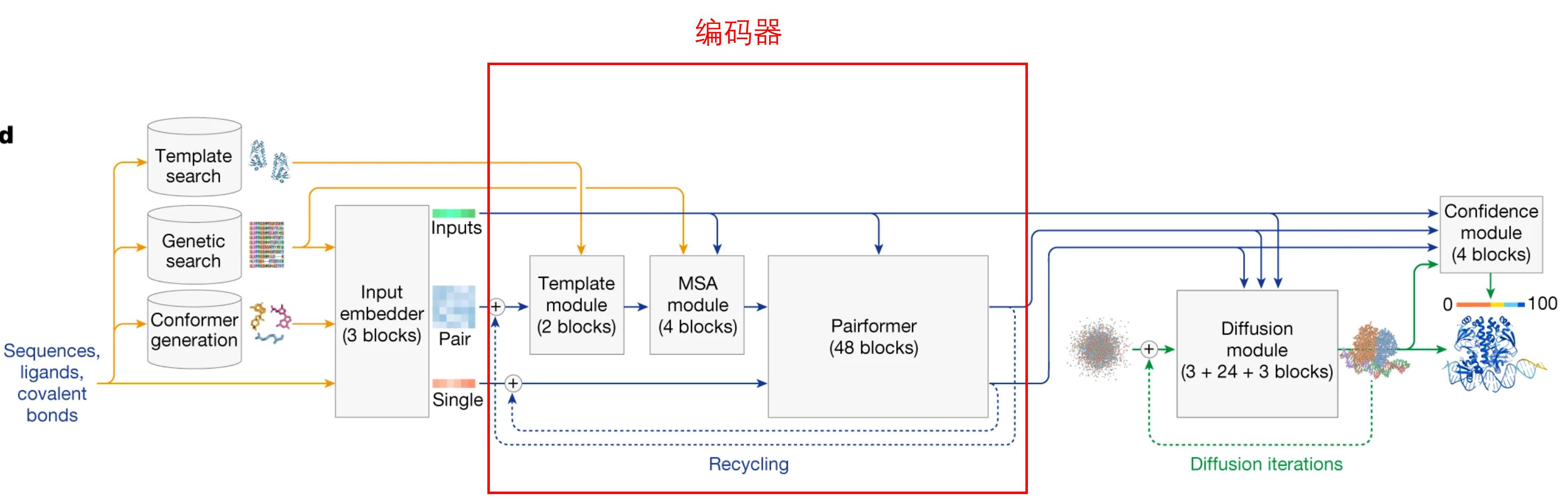
由三个module组成:
-
Template module: 整合在结构数据库中检索到的模板信息,关注对结构更重要的区域,然后再整合特征提取模块输出的 Pair embeddings,得到 Pair representation; -
MSA module: 在上一步的 Pair representation 的基础上,添加多序列比对信息,更新 Pair representation; -
Pairformer module: 是一个 Transformer module,用于更新 Pair representation 和 Single representation(Single representation 由 single embeddings 转化而来,过程略)。
通俗总结编码器:将模板结构信息、特征提取模块输出的 Pair embeddings、多序列比对信息整合在一起生成 Pair representation,然后把【对应输入生物序列原始信息的 Single representation】和【Pair representation】输入到善解数意的 Pairformer module 中,善解数意的 Pairformer module 能力超群,可以把 Single representation 信息和 Pair representation 信息规整的整整齐齐,并聚焦到重要信息,经过多次循环更新之后,输出 Single representation 和 Pair representation。
编码器输出的 Single representation 和 Pair representation 将会输入到 解码器模块。
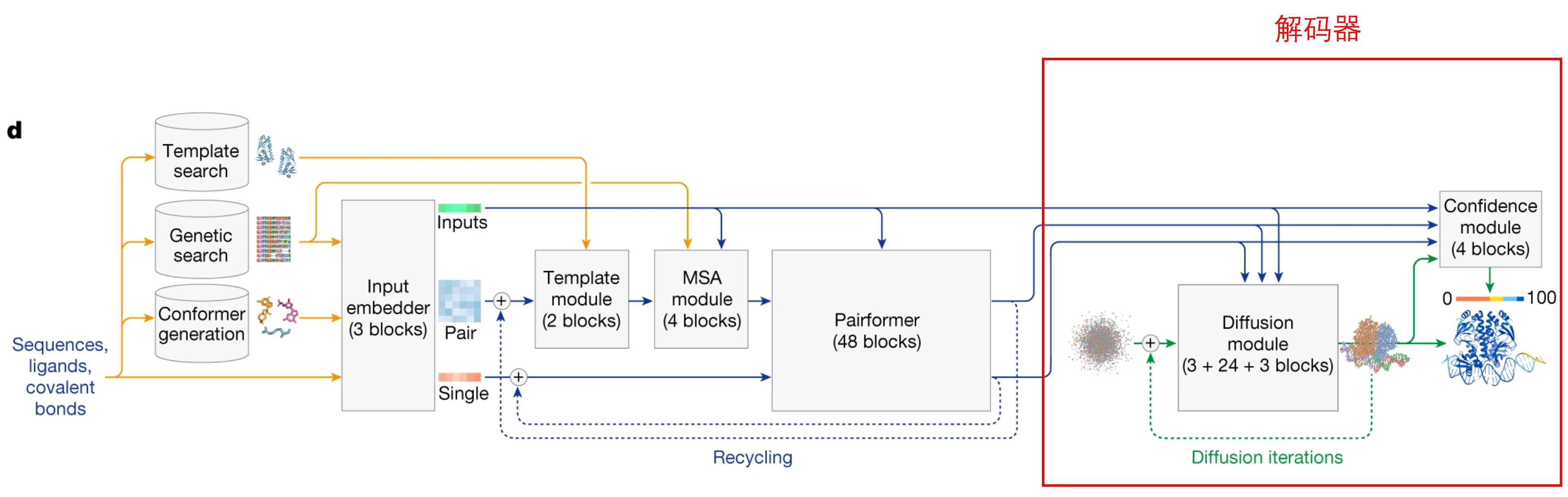
1.4 解码器
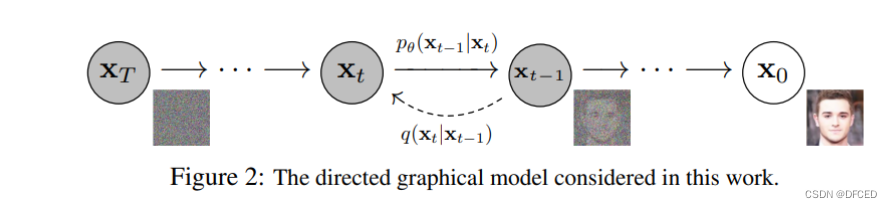
扩散模型:扩散模型(Diffusion Model)是一类生成模型的统称,这类模型的核心是通过逐步加噪和去噪过程生成结果。简单理解,加噪就是给一个模糊的图像添加马赛克,让模糊的图像变得更加模糊;去噪就是清除马赛克,恢复图像的原有清晰度,模型会学习这个清除马赛克的过程,延伸一下就是,模型会学习如何将一个模糊的图像变成一个清晰的图像。
通过一个直观例子了解扩散模型的作用:
解码器由两个 Module 组成:
-
Diffusion Module:使用扩散模型预测复合物结构,具体做法就是,Single representation 信息和 Pair representation 信息会转变为空间位置,这些信息刚开始看上去还是杂乱无章的原子点,看不出结构信息,但是经过扩散模型的多次(加噪和去噪)更新之后,杂乱无章的原子点变成了清新的三维结构。 -
Confidence Module:生成预测结构的预测置信度;使用预测局部距离差异检验(pLDDT)、预测比对误差(PAE)和预测距离误差(PDE)来评估置信度;pLDDT的范围为 0 到 100,值越大,置信度越高,代表预测越准确。
通俗理解解码器:解码器将编码器输出的 Single representation 和 Pair representation 经过扩散模型的反复折腾之后,生成一个清晰的三维结构,并给出这个结构的预测置信度。
2. AF3 在不同类型预测实验中的表现
AlphaFold3 能预测蛋白-小分子互作结构、蛋白-核酸互作结构、共价键修饰和蛋白质结构,在这四个类型的测试中,它的性能都显著高于竞争对手 RoseTTAFold,也高于针对特定类型的对手。
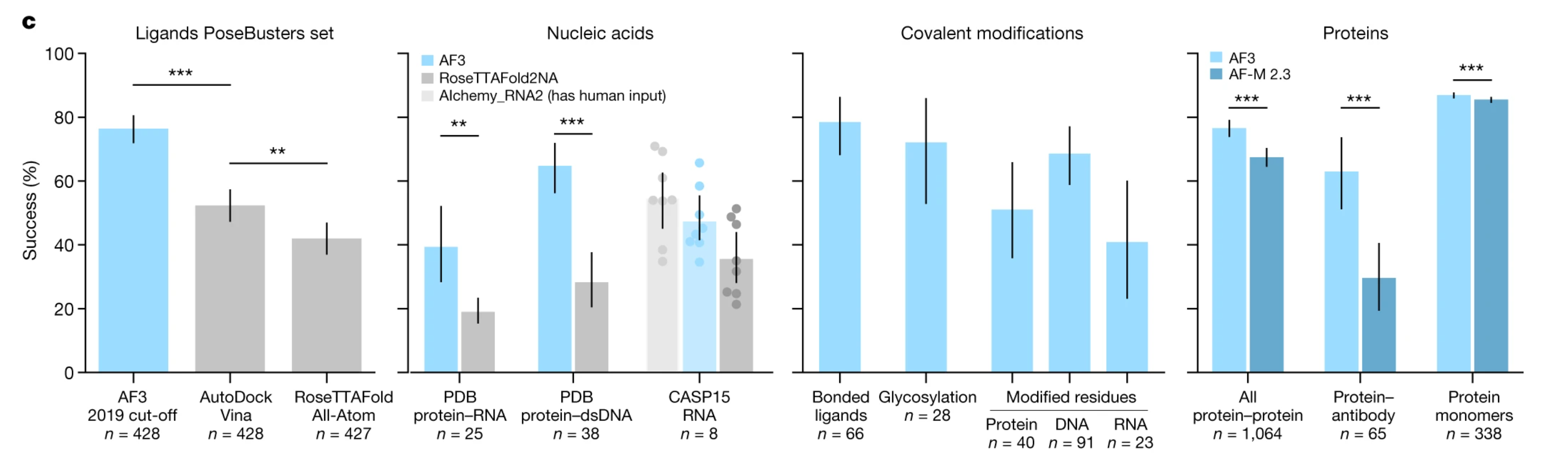
上图从左到右:
预测蛋白-小分子配体互作结构的表现(盲对接,不提供蛋白质结构或指定对接口袋):数据集选择的是 PDB 的 PoseBusters benchmark set,包含 428 个蛋白-配体结构,对比对象选择的是 AutoDock Vina 和 RoseTTAFold All-Atom。AlphaFold3 的表现显著高于 AutoDock Vina 和 RoseTTAFold All-Atom。
预测蛋白-核酸互作结构的表现:对比对象选择的是 RoseTTAFold,在蛋白-RNA 和蛋白-双链DNA 互作结构的预测上,AlphaFold3 的表现都显著高于 RoseTTAFold,在蛋白-RNA 互作结构的预测上,逊色于基于人类专家知识辅助的 Alchemy_RNA2。
预测共价修饰的表现:AF3 能比较准确地预测结合配体、糖基化、修饰的蛋白质残基和核酸碱基等共价修饰。
预测蛋白质复合物结构的表现:对比的是自家的前一代预测多亚基蛋白复合物结构的 Alphafold Mutimer 2.3;由于 Alphafold Mutimer 本身已经足够优秀,留下的提升空间不多,所以在预测蛋白蛋白复合物和蛋白单体之间的结构上的能力提升不大;预测蛋白-抗体复合物结构的能力大幅提高。
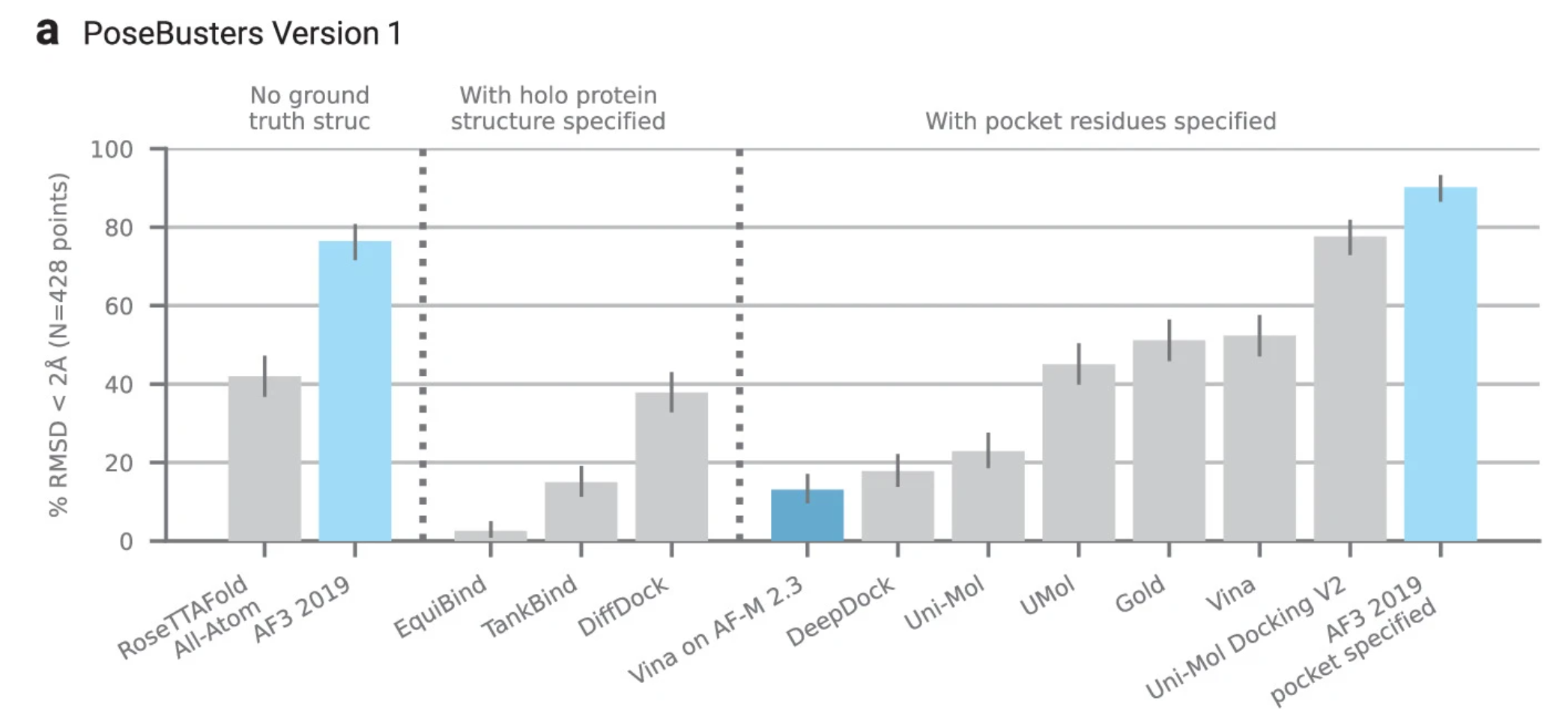
上图是提供蛋白质结构或指定对接口袋情况下,不同方法预测蛋白-小分子配体互作结构的表现对比。可以看到,在提供蛋白质结构时,AF 3定制版本 AF3 2019 的预测表现显著强于其他方法;在指定对接口袋时,AF 3定制版本 AF3 2019 pocket specified 的表现显著强于 AutoDock Vina、DeepDock 等一众专业的分子对接软件。
3. 在线使用 AlphaFold3
AlphaFold 3 可以在 https://www.alphafoldserver.com 使用,但是目前对允许使用的小分子配体和共价修饰有限制,很多还不能使用。如果不限制小分子配体的使用范围,AlphaFold 3 将成为一个非常好用的自动化的分子对接软件。
参考内容:
-
https://www.nature.com/articles/s41586-024-07487-w -
https://www.ebi.ac.uk/training/online/courses/alphafold/inputs-and-outputs/evaluating-alphafolds-predicted-structures-using-confidence-scores/plddt-understanding-local-confidence/ -
https://static-content.springer.com/esm/art%3A10.1038%2Fs41586-024-07487-w/MediaObjects/41586_2024_7487_MOESM1_ESM.pdf -
https://mp.weixin.qq.com/s/vAvwsf_mjRJH4IDU3lvpcg?sessionid=1726477416&subscene=0&ascene=0&fasttmpl_type=0&fasttmpl_fullversion=7382233-en_US-zip&fasttmpl_flag=0&realreporttime=1726479143208&clicktime=1726239340&enterid=1726239340 -
https://www.bilibili.com/video/BV142K6eBEWN/?spm_id_from=333.999.0.0&vd_source=2e0bed8f939119c48817ce61f4f75bdd
本文由 mdnice 多平台发布