我们前面介绍了经典的转录组分析流程:Hisat2 + Stringtie,可以帮助用户快速获得基因的表达量矩阵。 云上生信,未来已来 | 转录组标准分析流程重磅上线!
RNA STAR 也是一款非常流行的转录组数据分析工具。它不仅可以将测序 Reads 比对到参考基因组上,而且还可以输出类似 HTSeq-count 的定量结果。我们今天就介绍一套以 RNA STAR 为比对工具的新流程。
流程简介
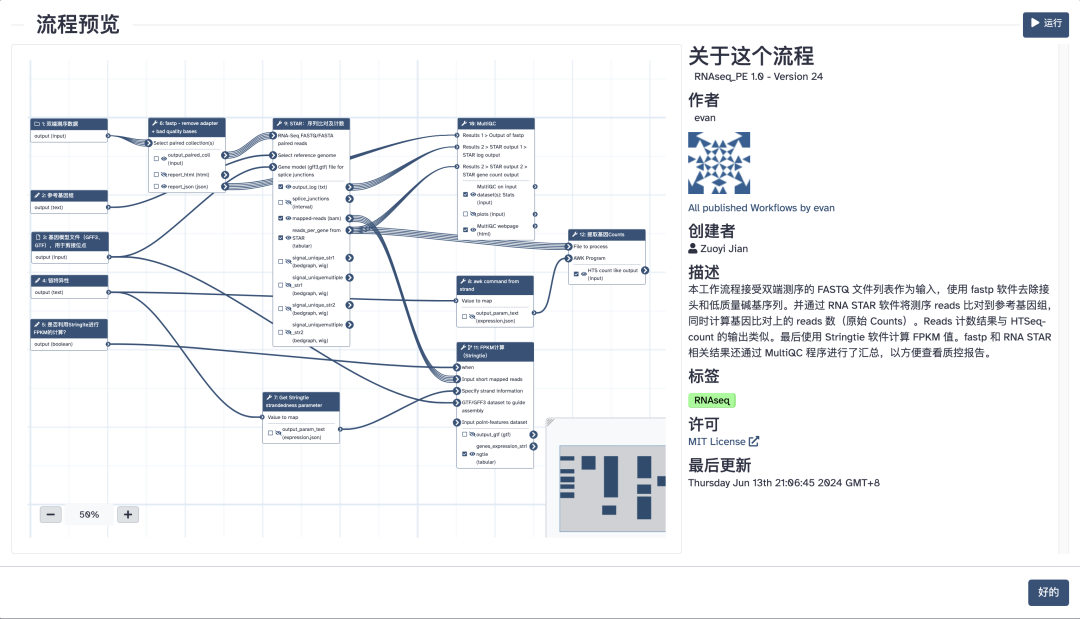
本工作流程接受双端测序的 FASTQ 文件列表作为输入,使用 fastp 软件去除接头和低质量碱基序列。并通过 RNA STAR 软件将测序 reads 比对到参考基因组,同时计算基因比对上的 reads 数(原始 Counts)。Reads 计数结果与 HTSeq-count 的输出类似。最后使用 Stringtie 软件计算 FPKM 值。fastp 和 RNA STAR 相关结果还通过 MultiQC 程序进行了汇总,以方便查看质控报告。

流程预览
点击小眼睛图标,即可预览流程。
运行流程
只需要设置如下参数即可以运行流程:
• 双端测序数据列表。制作方法请参照前述 Hisat2 + Stringtie 文章。
• 参考基因组。
• 基因注释文件。GFF3或GTF格式,可以自行上传,也可以使用平台提供的。
• 链特异性。有 unstranded、forward、reverse 三个选项,根据实际填写即可。
• 是否利用Stringtie进行FPKM的计算?若需要FPKM或TPM值,请选择“是”。

结果解读
RNA STAR 会输出:
• log 文件。即 RNA STAR 运行的日志。
• bam 文件。即比对文件,后续可用于 IGV 人工检查等。
• counts 文件。各样本每个基因的 Counts 数。
• 类 HTSeq-count 文件。
Stringtie 会输出:
• FPKM/TPM列表
MultiQC 会输出:
• fastp 和 RNA STAR 的质控结果汇总。
注意事项
本流程适用于 Bulk RNA 测序数据的定量分析,使用 RNA STAR 这款流行的比对和定量工具。如果您习惯使用 Hisat2 进行比对,请参照此前的推文。
云上转录组分析流程(点击图片跳转)

什么生信流程语言让你极度爽?(点击图片跳转)

推荐阅读:
如何自学生物信息学:从菜鸟到专家
生信人的自我修养:Linux 命令速查手册
经典教程:全转录数据分析实战
网上最全的 R 语言图库(建议收藏)| 简说基因 Recommend
清华大学生物信息学课件资料分享
生物信息学软件:两种风格
新年第一课:从零开始入门Galaxy生信云平台
生物信息学必备的R语言相关参考书 | 简说基因 Recommand
从单细胞数据分析的最佳实践看R与Python两个阵营的博弈
涉嫌侵权,容我解(jiao)释(bian)一下
生物信息学中的可重复性研究
关于简说基因
生信平台
Galaxy中国(UseGalaxy.cn)致力于打造中国人的云上生物信息基础设施。大量在线工具免费使用。无需安装,用完即走。活跃的用户社区,随时交流使用心得。
联系方式
QQ交流群(免费):925694514
微信交流群(免费):加微信好友,注明“Galaxy交流群”
客服微信:usegalaxy