写在前面
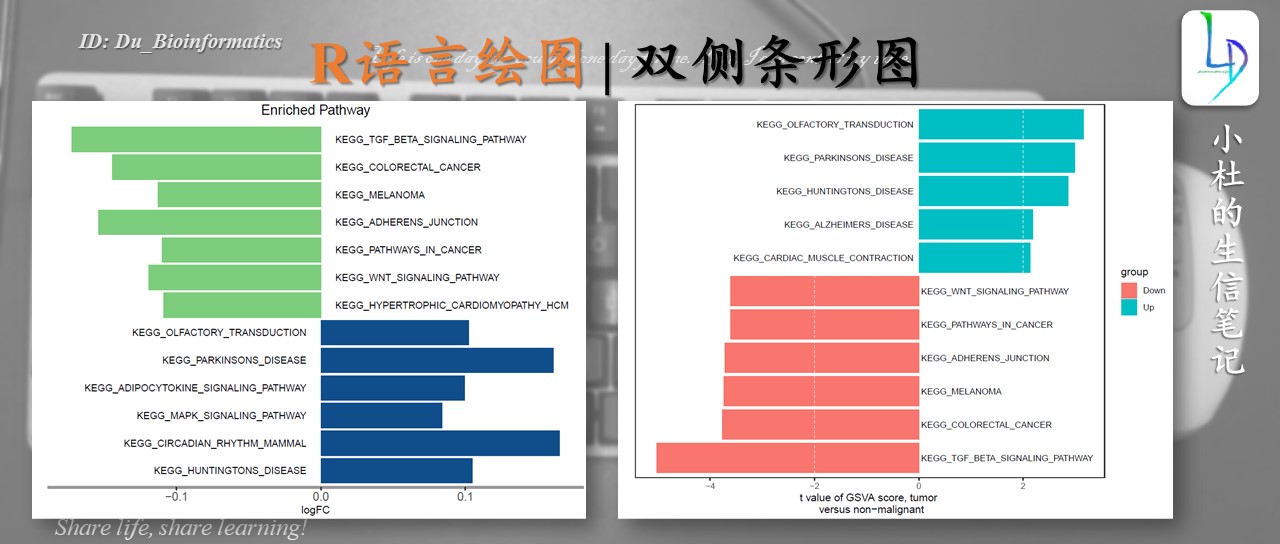
双侧条形图在我们的文章中也是比较常见的,那么这样的图形是如何绘制的呢? 以及它使用的数据类型是什么呢? 这些都是我们在绘制图形前需要掌握的,至少我们知道绘图的数据集如何准备,这样才踏出第一步。
今天的教程,我们会从数据的准备,以及数据如何整理,以及结合自己的绘图过程中遇到问题是如何解决来进行讲解。PS:仅代表个人的观点,以及自己遇到此问题时自己的方法来进行说明。也许,这个并不会死唯一且最好的方法,大家在绘图中请结合自己的能力和方法。
本期教程
获得本期教程
代码口令:20240205
原文链接:R语言绘图教程 | 双侧条形图绘制教程
什么时间及什么数据使用双侧条形图?一般使用此图形,基本是富集图
一般使用此图形,基本是富集图。如GO、KEGG、GSEA、GSVE等等,我们用来表示各个通路在研究中的显著性、得分情况等。
绘图
教程一
这里我们使用已经整理好的数据进行绘图,我们使用Execl进行整理数据。数据结果来源,GO、KEGG、GSEA、GSVE等富集结果。
1. 导入所需的R包
##'@导入所需的R包
library(limma)
library(ggplot2)
library(ggpubr)
library(tidyr)
library(tidyverse)
library(ComplexHeatmap)
2. 导入数据
在这里我们发现,我们有很多个富集通路,但是我们绘图的时候需要这么多吗?应该只需要各别几个。在这里我们可以手动调整,或是通过P值进行筛选。
dt1 <- read.csv("DE_KEGG.input.csv",header = T, row.names = 1)
###'@导入txt文件
#dt1 <- read.table("Input_KEGG.txt",header = T, row.names = 1, sep = '\t',check.names = F)
> dt1[1:5,1:5]
logFC AveExpr t P.Value adj.P.Val
KEGG_TGF_BETA_SIGNALING_PATHWAY -0.1723618 0.020400811 -5.029813 0.000001250 0.000190767
KEGG_COLORECTAL_CANCER -0.1443645 0.007857589 -3.773441 0.000222656 0.010260593
KEGG_MELANOMA -0.1129672 0.041504860 -3.736434 0.000255165 0.010260593
KEGG_ADHERENS_JUNCTION -0.1541395 0.027103539 -3.721243 0.000269769 0.010260593
KEGG_PATHWAYS_IN_CANCER -0.1100711 0.017776668 -3.615438 0.000395646 0.010260593
3. 筛选数据
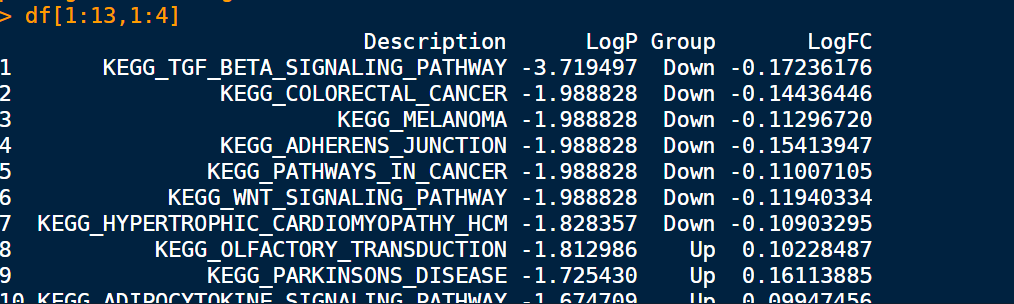
筛选出的作图的数据,这里我们的直接使用Description,LogP和group进行绘图
df <- data.frame(Description = rownames(dt1), LogP = log10(dt1$adj.P.Val), Group = dt1$group, LogFC = dt1$logFC)
4. 调整Description顺序
###'@调整`Description`顺序
df$Description <- factor(df$Description, levels = rev(df$Description))
若是我们这里有自己整理的Description顺序,可以直接在levels()后面加上自己的排序即可。
绘图
- 基础图形
ggplot(df, aes(x = LogFC, y = Description, fill = Group))+
geom_col()+