系列文章目录
利用 eutils 实现自动下载序列文件
提示:写完文章后,目录可以自动生成,如何生成可参考右边的帮助文档
文章目录
- 系列文章目录
- 前言
- 一、获取文献信息
- 二、下载文献PDF文件
- 参考
前言
大家好✨,这里是bio🦖。这次为大家带来自动收集文献信息、批量下载科研论文的脚本(只能批量下载公布在sci-hub上的科研论文)。平常阅读文献时只需要一篇一篇下载即可,并不需要用到批量下载的操作。但有时需要对某领域进行总结或者归纳,就可以考虑使用批量下载咯~
导师下令找文献,学生偷偷抹眼泪。
文献三千挤满屏,只下一篇可不行。
作息紊乱失双休,日夜不分忘寝食。
满腔热血搞科研,一盆冷水当头洛。
(打油诗人作)
一、获取文献信息
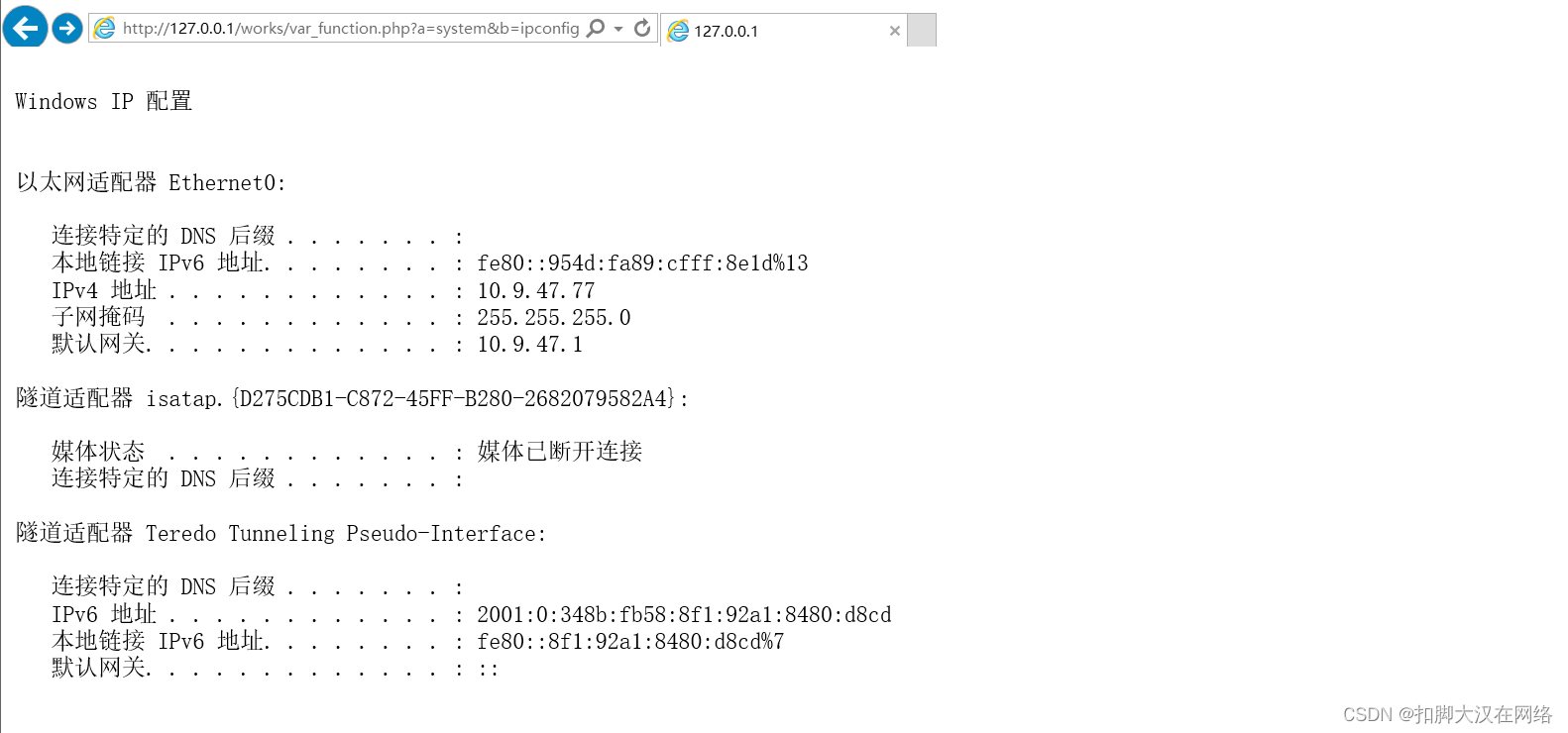
每个人的研究领域不一样,获取文献信息的方式不一样。这里以Pubmed1为例,PubMed是主要用于检索MEDLINE数据库中,生命科学和生物医学引用文献及索引的免费搜索引擎。之前爬取冠状病毒核酸数据使用过eutils,本篇博客也使用eutils去获取文献信息,关于eutils的介绍可以看利用 eutils 实现自动下载序列文件 。这里就不做介绍~
首先构造搜索url,其中term是你检索的关键词,year对应文献发表日期,API_KEY能够让你在一秒内的访问次数从3次提升到10次。这里将term替换为Machine learning,year替换为2022。然后使用requests库获取该url的对应的信息,在使用BeautifulSoup库将其转化为html格式。
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&api_key={API_KEY}&term={term}+{year}[pdat]
代码如下:
import pandas as pd
import requests
from bs4 import BeautifulSoup
import math
import re
import time
API_KEY = "Your AIP KEY"
term = "Machine Learning"
year = "2022"
url_start = f'https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&api_key={API_KEY}&term={term}+{year}[pdat]'
info_page = BeautifulSoup(requests.get(url_start, timeout=(5, 5)).text, 'html.parser')
爬取的结果如下,可以看到结果主要包括许多PMID。然后包括的信息总数<count>31236</count>、最大返回数<retmax>20</retmax>以及结果开始的序号<retstart>0</retstart>。下一步就是根据id获取文章对应的信息。
<?xml version="1.0" encoding="UTF-8" ?>
<!DOCTYPE eSearchResult PUBLIC "-//NLM//DTD esearch 20060628//EN" "https://eutils.ncbi.nlm.nih.gov/eutils/dtd/20060628/esearch.dtd">
<esearchresult><count>31236</count><retmax>20</retmax><retstart>0</retstart><idlist>
<id>37878682</id>
<id>37873546</id>
<id>37873494</id>
... # omitting many results
<id>37786662</id>
<id>37780106</id>
<id>37776368</id>
</idlist><translationset><translation> <from>Machine Learning</from> <to>"machine learning"[MeSH Terms] OR ("machine"[All Fields] AND "learning"[All Fields]) OR "machine learning"[All Fields]</to> </translation></translationset><querytranslation>("machine learning"[MeSH Terms] OR ("machine"[All Fields] AND "learning"[All Fields]) OR "machine learning"[All Fields]) AND 2022/01/01:2022/12/31[Date - Publication]</querytranslation></esearchresult>
可以看到结果也是31236条记录,爬取的信息忠实于实际的信息,可以放心食用~

获取文章的信息也是相同的步骤,首先构造url,然后爬取对应的信息,直接上代码:
API_KEY = "Your AIP KEY"
id_str = '37878682'
url_paper = f'https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=pubmed&api_key={API_KEY}&id={id_str}&rettype=medline&retmode=text'
paper_info = BeautifulSoup(requests.get(url_paper, timeout=(5, 5)).text, 'html.parser')
结果如下,包括PMID、DOI、摘要、作者、单位、发表年份等等信息,你可以从这一步获得的信息中获取你需要的信息如DOI。接下来的就是要爬取文献对应的PDF文件。这是最关键的一步。
PMID- 37878682
OWN - NLM
STAT- Publisher
LR - 20231025
IS - 2047-217X (Electronic)
IS - 2047-217X (Linking)
VI - 12
DP - 2022 Dec 28
TI - Computational prediction of human deep intronic variation.
LID - giad085 [pii]
LID - 10.1093/gigascience/giad085 [doi]
AB - BACKGROUND: The adoption of whole-genome sequencing in genetic screens has
facilitated the detection of genetic variation in the intronic regions of genes,
far from annotated splice sites. However, selecting an appropriate computational
tool to discriminate functionally relevant genetic variants from those with no
effect is challenging, particularly for deep intronic regions where independent
benchmarks are scarce. RESULTS: In this study, we have provided an overview of
the computational methods available and the extent to which they can be used to
analyze deep intronic variation. We leveraged diverse datasets to extensively
evaluate tool performance across different intronic regions, distinguishing
between variants that are expected to disrupt splicing through different
molecular mechanisms. Notably, we compared the performance of SpliceAI, a widely
used sequence-based deep learning model, with that of more recent methods that
extend its original implementation. We observed considerable differences in tool
performance depending on the region considered, with variants generating cryptic
splice sites being better predicted than those that potentially affect splicing
regulatory elements. Finally, we devised a novel quantitative assessment of tool
interpretability and found that tools providing mechanistic explanations of their
predictions are often correct with respect to the ground - information, but the
use of these tools results in decreased predictive power when compared to black
box methods. CONCLUSIONS: Our findings translate into practical recommendations
for tool usage and provide a reference framework for applying prediction tools in
deep intronic regions, enabling more informed decision-making by practitioners.
CI - (c) The Author(s) 2023. Published by Oxford University Press GigaScience.
FAU - Barbosa, Pedro
AU - Barbosa P
AUID- ORCID: 0000-0002-3892-7640
在进行尝试下载文献之前,构建两个函数便于批量爬取信息。get_literature_id和get_detailed_info分别获取文献的PMID以及详细信息。
def get_literature_id(term, year):
API_KEY = "Your AIP KEY"
# pdat means published date, 2020[pdat] means publised literatures from 2020/01/01 to 2020/12/31
url_start = f'https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&api_key={API_KEY}&term={term}+{year}[pdat]'
time.sleep(0.5)
info = BeautifulSoup(requests.get(url_start, timeout=(5, 5)).text, 'html.parser')
time.sleep(0.5)
# translate str to int
year_published_count = int(info.find('count').text)
id_list = [_.get_text() for _ in info.find_all('id')]
for page in range(1, math.ceil(year_published_count/20)):
url_page = f'https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&api_key={API_KEY}&term=pbmc+AND+single+cell+{year}[pdat]&retmax=20&retstart={page*20}'
time.sleep(0.5)
info_page = BeautifulSoup(requests.get(url_page, timeout=(5, 5)).text, 'html.parser')
id_list += [_.get_text() for _ in info_page.find_all('id')]
return id_list, year_published_count
def get_detailed_info(id_list):
API_KEY = "Your AIP KEY"
# PMID DOI PMCID Title Abstract Author_1st Affiliation_1st Journel Pulication_time
extracted_info = []
for batch in range(0, math.ceil(len(id_list)/20)):
id_str = ",".join(id_list[batch*20: (batch+1)*20])
detailed_url = f'https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=pubmed&api_key={API_KEY}&id={id_str}&rettype=medline&retmode=text'
time.sleep(0.5)
detailed_info = BeautifulSoup(requests.get(detailed_url, timeout=(5, 5)).text, 'html.parser')
literature_as_line_list = detailed_info.text.split('\nPMID')[1:]
for literature in literature_as_line_list:
# PMID
pmid = literature.split('- ')[1].split('\n')[0]
# DOI
if '[doi]' in literature:
doi = literature.split('[doi]')[0].split(' - ')[-1].strip()
else:
doi = ""
# PMCID
if "PMC" in literature:
pmcid = literature.split('PMC -')[1].split('\n')[0].strip()
else:
pmcid = ""
# Title
title = re.split(r'\n[A-Z]{2,3}\s', literature.split('TI - ')[1])[0].replace("\n ", "")
if '\n' in title:
title = title.replace("\n ", "")
# Abstract
abstract = literature.split('AB - ')[1].split(' - ')[0].replace("\n ", "").split('\n')[0]
# Author_1st
author = literature.split('FAU - ')[1].split('\n')[0]
# Affiliation_1st
tmp_affiliation = literature.split('FAU - ')[1]
if "AD - " in tmp_affiliation:
affiliation = tmp_affiliation.split('AD - ')[1].replace("\n ", "").strip('\n')
else:
affiliation = ""
# Journel
journel = literature.split('TA - ')[1].split('\n')[0]
# Publication time
publication_time = literature.split('SO - ')[1].split(';')[0].split('. ')[1]
if ':' in publication_time:
publication_time = publication_time.split(':')[0]
extracted_info.append([pmid, doi, pmcid, title, abstract, author, affiliation, journel, publication_time])
return extracted_info
爬取的部分结果如下图所示。有些文章没有DOI号,不信的话可以尝试在PubMed中搜索该文章对应的PMID33604555看看~。

二、下载文献PDF文件
关于下载文献的PDF文件,这里是从SciHub中爬取的,不是从期刊官方,部分文章可以没有被SciHub收录或者收录的预印版,因此,不能 保证上文中获取的信息就能从SciHub中全部下载成功。如果不能访问SciHub,自然就不能爬取对应的内容了,可以考虑买个VPN,科学上网~。
爬取SciHub上的文章需要构建一个访问头的信息,不然回返回403禁止访问。然后将获取的内容保存为PDF格式即可。其中从SciHub中爬取文献PDF文件参考了 用Python批量下载文献2
data = pd.read_csv('/mnt/c/Users/search_result.csv')
doi_data = data[~data['DOI'].isna()]
doi_list = doi_data["DOI"].tolist()
pmid_list = doi_data["PMID"].tolist()
for doi, pmid in zip(doi_list, pmid_list):
download_url = f'https://sci.bban.top/pdf/{doi}.pdf?#view=FitH'
headers = {"User-Agent": "Mozilla/5.0 (Macintosh; Intel Mac OS X 10_15_7) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/93.0.4577.63 Safari/537.36"}
literature = requests.get(download_url, headers=headers)
if literature.status_code != 200:
print(f"this paper may have not downloading permission, it's doi: {doi}")
else:
with open(f'/mnt/c/Users/ouyangkang/Desktop/scraper_literature/{pmid}.pdf', 'wb') as f:
f.write(literature.content)
爬取结果如下图所示,成功下载了199篇~(这里的关键词不是机器学习,提供的doi数量是522,下载成功率为38%)。
参考
.Pubmed official websity ↩︎
用Python批量下载文献 ↩︎